Транскрипционная регуляторная днк гена хомяка ef-1
Иллюстрации
Показать всеИзобретение относится к генной инженерии, конкретно к очищению и выделению полинуклеотидов, регулирующих транскрипцию генов млекопитающих, и может быть использовано для регуляции экспрессии гетерологичных полинуклеотидов, получения трансгенных животных и для идентификации родственных регуляторных последовательностей ДНК. ДНК, содержащую транскрипционную регуляторную ДНК гена хомяка EF-1α, выделяли путем скрининга геномной библиотеки к ДНК яичника китайского хомячка (СНО-К1). Конструируют химерный полинуклеотид, включающий выделенную регуляторную ДНК гена хомяка EF-1α, оперативно связанную с генной последовательностью, кодирующей целевой белковый продукт, отличный от белка, кодируемого геном хомяка EF-1α. Полученный химерный полинуклеотид используют в составе экспрессионной плазмиды для трансформирования или трансфицирования клетки-хозяина. Для увеличения транскрипции целевого гена в клетке-хозяине интегрируют ДНК, содержащую регуляторную ДНК гена хомяка EF-1α, в геномную ДНК клетки-хозяина в положение, оперативно связанное с целевым геном. Изобретение обеспечивает повышение в 3-11 раз уровней экспрессии мРНК оперативно связанных гетерологичных полинуклеотидов, 14 с. и 17 з.п. ф-лы, 3 табл.
Реферат
Предпосылки к созданию изобретения
Транскрипция любого из установленных генов эукариот осуществляется с участием одной из трех РНК-полимераз, каждая из которых действует на определенную подгруппу генов. В то время как транскрипция рибосомальных РНК проходит с участием РНК-полимеразы I, а малые рибосомальные РНК и тРНК транскрибируются РНК-полимеразой III, кодирующие белок ДНК-последовательности и основная часть малых ядерных РНК транскрибируются с участием РНК-полимеразы II. Для каждого типа генов при осуществлении транскрипции необходимо взаимодействие соответствующей полимеразы с промоторными генными последовательностями и образование устойчивого комплекса инициации транскрипции. В общем виде, для осуществления транскрипции от любой из трех полимераз необходимо также взаимодействие некоего фактора связывания с промоторной последовательностью и рекогниция фактора связывания вторым фактором, который тем самым обеспечивает взаимодействие полимеразы с генной последовательностью. И если этот механизм включает минимум условий для осуществления транскрипции с участием РНК-полимераз I и III, то процесс, приводящий к транскрипции с участием РНК-полимеразы II, является более сложным.
По-видимому, из-за большого разнообразия генных последовательностей, транскрибированных РНК-полимеразой II, и в связи с тем, что типы регуляции для этих генов высоковариабельны в пределах одной клетки и при переходе от одной клетки к другой, на транскрипцию с участием РНК-полимеразы II оказывают влияние многочисленные факторы транскрипции в комплексе инициации в дополнение к взаимодействиям других связывающих белков с регуляторными последовательностями ДНК, не являющимися промоторами. Эти другие связывающие белки могут активировать транскрипцию выше основного уровня или подавлять ее полностью. Репрессорное связывание может быть также оценено как способ предотвращения активации с учетом наблюдений о том, что основной уровень транскрипции у высших эукариот обычно очень низкий. С другой стороны, активация обычно является конечным откликом на какой-либо физиологический сигнал, и для образования активного комплекса инициации транскрипции требуется или удаление репрессорного связывающего белка, или изменение в структуре хроматина.
В коре образования транскрипционного комплекса находится, являсь при этом необходимым условием для основных уровней экспрессии генов, промоторная последовательность, называемая ТАТА-боксом, располагающаяся против хода транскрипции от начального сайта транскрипции полимеразы II. ТАТА-бокс является сайтом связывания для вездесущего фактора транскрипции, обозначаемого TFIID, однако, как уже упоминалось, у большинства генов на транскрипцию от промоторной последовательности оказывают сильное влияние дополнительные регуляторные последовательности ДНК, которые могут либо усиливать, либо подавлять транскрипцию генов. Элементы ДНК такого типа находятся в вариабельных положениях по отношению к кодирующим последовательностям гена и к ТАТА-боксу. Эти дополнительные регуляторные элементы транскрипции часто функционируют как тканеспецифичные или клеточноспецифичные.
При экспрессии рекомбинантных белков особенно важен выбор регуляторной ДНК, которая включает промоторную ТАТА-последовательность и дополнительные регуляторные элементы, совместимые с механизмом транскрипции клетки-хозяина. По этой причине обычно отдают предпочтение регуляторной ДНК, которая является эндогенной по отношению к клетке-хозяину. В качестве альтернативы значительные успехи достигнуты при использовании регуляторной ДНК, полученной из вирусных геномных последовательностей, для оценки широты распространения вирусов вообще и для демонстрации активности вирусной регуляторной ДНК в различных типах клеток. Хорошо изучены и широко используются для экспрессии рекомбинантного белка вирусные регуляторные ДНК, включая, например, ранний промотор/энхансер гена вируса SV40 (Dijekema EMBO J.4, 761, 1985), повторяющаяся ДНК с длинным окончанием вируса саркомы Рауса (Gorman и др., Proc. NatI. Acad. Sci. USA, 79, 6777, 1982 b), фрагменты бычьего вируса папилломы (Sarver и др., Mol. Cell. Biol., 1, 486, 1981) и элементы промотора/энхансера человеческого цитомегаловируса (Boshart и др., Cell, 41, 521, 1985). Несмотря на большое разнообразие типов клеток, для которых было продемонстрировано функционирование вирусных регуляторных ДНК, не исключено, что существуют невирусные промоторные/энхансерные элементы ДНК, допускающие повышенный уровень транскрипции рекомбинантных белков посредством более эффективного использования механизмов транскрипции клетки-хозяина.
Следовательно, в этой области существует необходимость в идентификации промоторных/энхансерных последовательностей регуляторной ДНК для усиления экспрессии рекомбинантного белка и обеспечения высокого выхода целевого белкового продукта. Особенно важной является необходимость идентификации таких промоторных/энхансерных регуляторных ДНК, которые наиболее эффективно могут использоваться в клетках млекопитающих для усиления воспроизведения in vitro рекомбинантных белков, гликозилирующихся по механизму, сходному с механизмом гликозилирования белков при экспрессии белка in vivo. Менее вероятно, что экспрессированные таким способом и введенные в терапевтических или профилактических целях белки окажутся антигенными, более вероятно, что они окажутся физиологически активными. Регуляторные последовательности ДНК такого типа пригодны для внедрения в клетки-хозяева для усиления экспрессии генов, эндогенных по отношению к этим клеткам, или генов, введенных ранее в геном клетки-хозяина с помощью хорошо известных приемов, широко используемых в практике.
Краткое описание изобретения
Настоящее изобретение связано с очищенными и выделенными полинуклеотидами из клеток хомяка, которые регулируют транскрипцию генов.
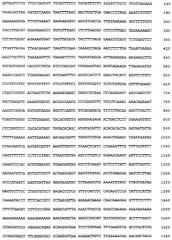
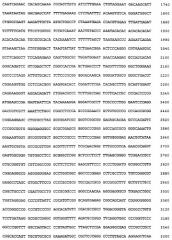
Полинуклеотиды включают последовательности регуляторных ДНК в 5’-положении к участкам трансляции гена яичника китайского хомяка EF-1α . Предпочтительные ДНК по изобретению обозначены как регуляторные ДНК CHEF1 и включают ДНК примерно в 3,7 т.н., с протяженностью от сайта рестрикции SpeI до инициирующего метионинового кодона (ATG) белка ef-1α . Изобретение охватывает также полинуклеотиды с менее чем 3,7 т.н., способные усиливать транскрипцию оперативно связанного гена. Активные фрагменты ДНК по изобретению, характеризующиеся способностью регулировать (т.е., усиливать) транскрипцию генов, легко идентифицируются с помощью делеционных исследований, широко применяемых специалистами. Регуляторные ДНК по изобретению включают полинуклеотиды, выделенные из природных источников, таких как культивированные клетки, а также полинуклеотиды, полученные энзиматическим или химическим синтезом. Так, в одном из примеров своего осуществления изобретение обеспечивает способ получения ДНК CHEF1 из библиотеки генов. В качестве альтернативы ДНК может быть получена энзиматическим синтезом с использованием, например, полимеразной цепной реакции (ПЦР) или только химическим синтезом, в котором последовательно связываются одиночные нуклеотиды, или перекрывающиеся олигонуклеотиды гибридизуются и лигируются. Последовательность ДНК из наиболее предпочтительных примеров осуществления изобретения расположена в последовательности с идентификационным номером 1 (SEQ ID NO:1). Кроме того, в изобретении использованы последовательности ДНК, которые при определенных условиях гибридизуют с ДНК, находящейся в SEQ ID NO:1. Определенные условия включают промывание при температуре около 65° С в буфере, содержащем примерно 2xSSC (хлорид натрия-цитрат натрия) и примерно 0,1% SDS (додецилсульфат натрия), или другие равноценные условия.
Кроме того, изобретение охватывает плазмидную ДНК, включающую регуляторные полинуклеотиды CHEF1. Плазмиды по данному изобретению могут также включать последовательности ДНК, кодирующие целевой белок или РНК, оперативно связанную с полинуклеотидными последовательностями CHEF1. Кроме того, изобретение охватывает клетки-хозяева, трансформированные, трансфицированные и/или электропорированные с применением полинуклеотидов или плазмид по изобретению. Плазмидная ДНК по изобретению может быть интегрирована в геном клетки-хозяина или может существовать в закрытой кольцевой форме. Предпочтительные плазмиды по данному изобретению, наиболее подходящие для встраивания целевых последовательностей ДНК, включают плазмиды pDEF14 и pDEF2. Бактериальные клетки, трансформированные с помощью плазмид pDEF14 и pDEF2, были заложены на хранение 9 апреля 1997 года и 4 марта 1997 года соответственно в Американскую коллекцию типов культур, (12301 Parklawn Drive, Rockville, Maryland 20852) под регистрационными номерами 98389 и 98343 соответственно.
Изобретение охватывает также линейную векторную ДНК, содержащую полинуклеотиды последовательности CHEF1. Векторы по изобретению могут быть получены из вирусных источников или синтезированы in vitro. Кроме того, изобретение охватывает клетки-хозяева, трансфицированные или электропорированные с применением линейных векторных последовательностей. ДНК этого типа применимы, в частности, для экспрессии гетерологичных генных последовательностей, оперативно связанных с ДНК-последовательностями CHEF1, или для сайт-направленной гомологичной рекомбинации, при которой последовательности CHEF1 могут быть включены в геном клетки-хозяина для того, чтобы модулировать экспрессию оперативно связанной последовательности.
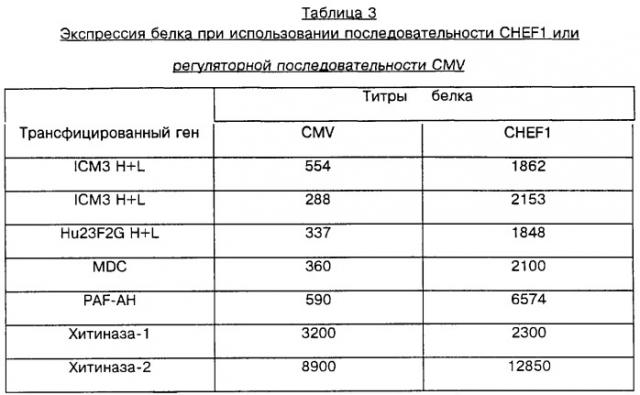
Кроме того, изобретение охватывает молекулы химерной рекомбинантной ДНК, в которых ДНК-последовательность CHEF1 оперативно связана (т.е. промотирует транскрипцию) с генной последовательностью, кодирующей целевой белковый продукт. Обычно химерными считаются молекулы, содержащие домены, которые ассоциируются с последовательностями дикого типа не в нормальных условиях; в настоящем изобретении химерная ДНК может содержать часть или всю регуляторную ДНК-последовательность CHEF1 в ассоциации с ДНК-последовательностью, отличающейся от кодирующего белок хомяка гена EF-1α . Белки, кодируемые химерными молекулами, включают физиологически активные белки, части или субъединицы активных белков, а также маркерные или репортерные белки. Полинуклеотидные последовательности, кодирующие белковый продукт, могут быть получены из комплементарных ДНК (кДНК), геномных ДНК, синтетических ДНК или ДНК, полученных путем комбинаций этих типов молекул. Белковые продукты могут быть эндогенными (т.е., присутствующими в геноме клеток яичника китайского хомяка СНО в норме, в отсутствие введения ДНК путем трансформации, трансфекции и т.п.) по отношению к клеткам СНО. Кроме того, белковые продукты могут быть кодированы экзогенными источниками, причем экзогенный источник, например синтезированная ДНК, может отличаться от генома клеток СНО. Предпочтительные химерные молекулы по изобретению включают молекулы, в которых ДНК CHEF1 оперативно связана с ДНК, кодирующей: а) тяжелую цепь анти-ICАМ3 антитела ICM3, б) легкую цепь ICM3, в) тяжелую цепь анти-CD11/CD18 антитела hu23F2G, г) легкую цепь hu23F2G, д) хитиназу, е) ацетилгидролазу фактора активации тромбоцитов (PAF-AH), ж) полученный на основе макрофагов хемокин (MDC). Бактериальные клетки-хозяева, трансформированые с плазмидной ДНК, включая химерные молекулы, были заложены на хранение 1 апреля 1997 года в Американскую коллекцию типов культур под регистрационными номерами (а) 98381, (б) 98382, (в) 98383, (г) 98384, (д) 98385, (е) 98386 и (ж) 98387 соответственно.
Кроме того, изобретение связано с клетками-хозяевами, трансформированными или трансфицированными с помощью ДНК-последовательностей CHEF1 по данному изобретению. Предпочтительными клетками-хозяевами являются полученные от китайского хомяка (Cricetutus griseus) клетки, в которых, как предполагается, регуляторная ДНК CHEF1 обеспечивает высокие уровни экспрессии белка. Однако изобретение охватывает и другие типы клеток, полученные от альтернативных видов, включая, например, армянских хомяков (Cricetulus migratoris) или сирийских (золотых) хомяков (Mesocricetus auratus), а также клетки животных других биологических видов. Клетки могут быть получены из легкого, яичника, брюшины, мышц, поджелудочной железы, почек, меланомы или из соматических источников, а также из всего плода или эмбриона. Клетки-хозяева по изобретению, перечисленные выше, а также другие типы клеток, например миеломная клеточная линия, в которой, как предполагается, регуляторная ДНК CHEF1 обеспечивает высокие уровни транскрипции, могут быть получены от Американской коллекции типов культур или в качестве альтернативы культивированы непосредственно от источников животного происхождения.
Рекомбинантные молекулы по изобретению, включающие регуляторную ДНК-последовательность CHEF1, обеспечат повышенные уровни экспрессии мРНК оперативно связанных гетерологичных (т.е. не находящихся в геноме хозяина без введения полинуклеотидов трансфекцией, трансформацией и т.п.) полинуклеотидов. В зависимости от природы полинуклеотида, связанного с полинуклеотидными последовательностями CHEF1, усиленная транскрипция в конечном итоге приведет к повышенным уровням полипептидов или повышенным уровням различных типов РНК в тех случаях, когда связанный полинуклеотид кодирует, например, транспортную РНК, рибосомальную РНК, малую ядерную РНК и т.п. Типы РНК могут также включать антисмысловую РНК комплементарную мРНК, транскрибированную от эндогенной или экзогенной генной последовательности. Усиленная полипептидная трансляция неизбежно зависела бы от наличия соответствующего трансляционного сигнала в мРНК.
Кроме того, изобретение обеспечивает методы, с помощью которых ДНК-последовательность CHEF1 включается в специфические сайты геномной ДНК клеток-хозяев для повышения уровней транскрипции эндогенной генной последовательности. Для включения всей ДНК CHEF1 или ее части может быть использована гомологичная рекомбинация. В модифицированных таким способом клетках-хозяевах ДНК CHEF1 оперативно связана с кодирующими ДНК последовательностями. Смотри, например, публикацию международной заявки WO 94/12650, публикацию международной заявки WO 92/20808 и международной заявки WO 91/09955. В конечном итоге, изобретение охватывает измененные геномные последовательности ДНК, в которые включена регуляторная ДНК-последовательность CHEF1.
В качестве альтернативы изобретение охватывает клетки-хозяева, в которых экзогенная ДНК включена в смежное положение и в оперативный участок по отношению к присутствующей в геноме ДНК CHEF1. Клетки-хозяева этого типа включают линию клеток СНО, и внедренная последовательность заменяет либо ДНК, кодирующую EF-1α , либо внедряется между регуляторной ДНК CHEF1 и ДНК, кодирующей EF-1α . Изобретение охватывает также и другие отличающиеся от клеток линии СНО клетки-хозяева, в которых ДНК CHEF1 уже включена в геном, а дополнительная ДНК включается в оперативно связанный участок.
Кроме того, изобретение обеспечивает методы усиления транскрипции целевого гена, включая этап введения в клетку-хозяина полинуклеотида, содержащего хомячковую регуляторную последовательность EF-1α , таким образом, что регуляторная последовательность интегрируется в геномную ДНК хозяина в опреративно связанный участок по отношению к целевому гену. Помимо этого изобретение охватывает способ усиления транскрипции целевого гена, включающего этап введения в клетку-хозяина полинуклеотида, содержащего хомячковую регуляторную последовательность EF-1α и последовательность, являющуюся мишенью, упомянутая последовательность-мишень построена таким образом, что позволяет регуляторным последовательностям интегрировать в положение, оперативно связанное с целевым геном, кодирующим белок, отличающийся от белка, кодируемого EF-1α . Способы по изобретению включает средство усиления транскрипции генов, которые являются эндогенными по отношению к клеткам линии СНО, а также способы усиления транскрипции генов, которые являются экзогенными по отношению к клеткам линии СНО.
Рекомбинантные молекулы по изобретению могут использоваться для получения трансгенных животных, когда химерная рекомбинантная ДНК, включающая ДНК CHEF1, оперативно связанную с целевой ДНК-последовательностью, вводится в развивающуюся зародышевую или соматическую клетку животного. Химерные рекомбинантные молекулы могут вводиться, например, микроинъекцией, и если она осуществляется с использованием зародышевых клеток, то в результате все клетки животного могут содержать рекомбинантную ДНК по данному изобретению. В качестве альтернативы химерная рекомбинантная ДНК по изобретению может вводиться в клетки эмбриона, в этом случае ДНК по изобретению может включиться в большинство клеток полученного животного.
ДНК CHEF1 используют также для идентификации родственных регуляторных последовательностей ДНК, которые могут способствовать повышенной генной экспрессии помимо той, которая возможна для последовательности CHEF1. Подобным образом, знание ДНК-последовательностей CHEF1 позволяет конструировать синтетические ДНК или путем синтеза de novo, или путем однократной либо многократной модификации последовательностей CHEF1, причем получаемые синтетические ДНК содержат последовательности, подобные CHEF1, но способные промотировать более высокие уровни транскрипции генов.
Подробное описание изобретения
Настоящее изобретение иллюстрируется следующими примерами. Пример 1 описывает клонирование хомячкового гена EF-1α и родственной последовательности CHEF1 регуляторной ДНК. Пример 2 связан с субклонированием и анализом регуляторной последовательности CHEF1. В примере 3 приведена характеристика промоторного полинуклеотида CHEF1, в примере 4 приведено описание конструкции различных экспрессионных векторов, которые включают ДНК CHEF1. Пример 5 описывает анализ методом трансфекции, использованный для определения эффективности регуляторной ДНК CHEF1. Пример 6 приводит подробности сравнения уровней экспрессии рекомбинантного белка с использованием или регуляторной ДНК CHEF1, или промотора цитомегаловируса (CMV). В примере 7 исследованы различия регуляторной способности ДНК CHEF1 различной протяженности.
Пример 1
Клонирование последовательности CHEF1
Для выделения хомячкового гомолога человеческого гена EF-1α скринировали библиотеку геномной к ДНК яичника китайского хомяка (СНО-К1) (Stratagene, La Jolla, CA).
Библиотеку СНО-К1, продукт частичного расщепления с помощью рестриктазы Sau3A в клонирующем векторе λ FIX®II, получали от фирмы Stratagene (La Jolla, CA) в штаммах клетки-хозяина XL-1-Blue MRA и XL1-Blue MRA (P2). Клетки-хозяева получали в соответствии с предложенным производителем протоколом со следующими модификациями.
Краткое изложение методики: клетки из первичной культуры в глицерине переносили в виде слоя в планшеты со средой LB, не содержащей антибиотика. Единичные колонии собирали для инокуляции жидкой среды LB и выращивали до достижения поздней логарифмической фазы и оптической плотности при 600 нм, OD600, равной примерно 0,9. В этот момент клетки или закладывались на хранение в соответствии с предложенным производителем протоколом, или немедленно использовались, как описано ниже.
При получении культур для посева единичные колонии, извлеченные из планшетов, как описано выше, использовали для инокуляции; клетки выращивали в 50 мл среды LB, уравновешенной 0,5 мл 1 М раствора сульфата магния и 1 мл 10% мальтозы в деионизированной воде. После выращивания в течение ночи при 30° С клетки собирали с помощью центрифугирования в настольной центрифуге (2000 об/мин в течение 5 минут при комнатной температуре) и вновь суспендировали в 10 мМ сульфате магния при значении OD600, составляющем 0,5.
Фаг λ , предоставленный производителем, разбавляли в буфере SM (получен из 1 л основного раствора, содержащего 5,8 г хлорида натрия, 2,0 г моногидрата сульфата магния, 50 мл 1 М трис-HCl, рН 7/5, и 5 мл 2% (вес/объем) желатина) с кратностью 10-105, 2 мкл каждого разбавления прибавляли к 400 мкл клеток-хозяев в 10 мМ сульфате магния (OD600≈0,5). Полученную смесь инкубировали в течение 15 минут при 37°С для того, чтобы дать возможность фагу прикрепиться к клеточному слою, прибавляли агар (0,75% агароза LBM при 48° С), каждую смесь наносили дважды на пластинки с агарозой LBM. Результаты выражали в виде титра 3× 106 бляшкообразующих единиц (БОЕ)/мкл фагового раствора.
Свежие клетки-хозяева получали, как описано выше, перед скринированием библиотеки. Приблизительно 50000 БОЕ фага прибавляли к 600 мкл клеток-хозяев (при OD600≈0,5) с 6,5 мл 0,75% агарозы LBM при 48° С. Смесь наносили на пластины с агарозой и инкубировали в течение примерно 8 часов при 37° С. Затем пластины охлаждали в течение 5 часов при 4° С для того, чтобы предотвратить прилипание верхнего слоя агарозы к нитроцеллюлозной мембране. На пластины помещали мембраны ВА-85 с размером пор 0,45 мкм (фирма S+S, Keene, NH), перенос осуществляли в течение двух минут. Мембраны удаляли с пластин и перенесенную ДНК (Т-ДНК) денатурировали в смеси 1,5 М хлорид натрия, 0,5 М гидроксид натрия в течение двух минут. Фильтры нейтрализовали в течение 5 минут в смеси 1,5 М хлорид натрия, 1,0 М трис-НСl (рН 8,0), блотировали на бумагу Ватман 3-ММ и сушили в вакууме при 80° С в течение примерно 1,5-2 часов.
Человеческую последовательность кДНК EF-1α (Uetsuki и др., J. Biol. Chem., 264, 5791-5798, 1989) с 95% идентичностью к кодирующему участку СНО EF-1α , как было показано ранее (Hayashi и др., J. Biochem., 106, 560-563, 1989), использовали в качестве зонда для скрининга библиотеки и получали, как описано ниже. Зонд в 1,4 т.н. человеческого гена EF-1α получали из плазмиды М0107, вектора pRc/CMV (фирмы Invitrogen, San Diego, CA), содержащего человеческую кДНК EF-1α , включенную в сайт EcoRI. Для того чтобы подтвердить, что плазмида М0107 содержит ожидаемую человеческую кДНК, включенную ДНК секвенировали с 3’- и 5’-праймерами вектора.
94-17 AGGCACAGTCGAGGCTGATC SEQ ID NO:2
94-18 TTCCAGGGTCAAGGAAGGCA SEQ ID NO:3
Первые 263 п.н. вставки были более чем на 90% идентичны опубликованной человеческой кодирующей последовательности EF-1α , и, поскольку точное определение последовательности на 3’-конце было затруднено, с ожидаемой последовательностью можно было совместить небольшие участки. Вместе эти совмещения указывали на то, что плазмида кодировала желаемую последовательность.
Человеческую вставку удаляли полностью из плазмиды путем расщепления рестриктазой EcoRI, полосу кДНК в 1.4 т.н. дважды очищали на геле, используя набор для гелевой экстракции QIAquick фирмы QIAGEN в соответствии с предложенным производителем протоколом. ДНК элюировали 50 мкл буфера ТЕ, полученный раствор концентрировали до объема 25 мкл с помощью концентратора Microcon-10 (фирмы Amicon, Beverly, МА), аликвоту метили с 32Р-α -dTTP и 32Р-α -dСТР, используя набор для мечения произвольно праймированных ДНК фирмы Boehringer Mannheim в соответствии с предложенным производителем протоколом. Меченый зонд очищали, используя вращающуюся колонку G50 для удаления не включившихся нуклеотидов и сравнения очищенного зонда с аликвотой до вращения, в которой включение радиоактивной метки составило 46%, при счете 5× 105 имп./мин/мкл.
Нитроцеллюлозные мембраны, полученные, как описано выше, зондировали следующим образом. Основной прегибридизационный/гибридизационный буферный раствор включал 22,5 мл буфера 20× SSC, 30,0 мл 50х раствора Денхардта, 3,0 мл 1 М фосфатного буфера (рН 6,8) (69 мл 1 М NаН2РO4, 31 мл 1 М Na2HPO4), 0,75 мл 20% SDS, 78,75 мл дистиллированной воды. Для прегибридизации кипятили 1,4 мл (10 мг/мл) ДНК из молок лососевых (фирмы Stratagene) с 0,6 мл дистиллированной воды в течение 5 минут, затем прибавляли воду до 7 мл и 72 мл основного буферного раствора. Фильтры инкубировали при 65° С не менее 2 часов в прегибридизационном буфере.
Для гибридизации смешивали 30 мкл зонда, 200 мкл (10 мг/мл) ДНК из молок лососевых и 770 мкл дистиллированной воды, кипятили в течение 5 минут, затем прибавляли до 36 мл основной буфер с 3 мл дистиллированной воды. Прегибридизационный раствор удаляли с фильтров, прибавляли гибридизационный буфер, фильтры инкубировали при 65° С в течение ночи. По окончании гибридизации фильтры промывали в течение трех часов при 65° С, трижды меняя буфер, содержащий 2xSSC и 0,1% SDS, после чего проводили авторадиографию в течение 48 часов.
Идентифицировали 47 положительных колоний и переносили их в 1 мл буфера SM, содержащего 20 мкл хлороформа. Для удаления возможных псевдогенов, лишенных одного или более интронов, были сконструированы праймеры для полимеразной цепной реакции (ПЦР) так, чтобы каждый фланкировал два интрона: праймеры 95-136 (SEQ ID NO: 4) и 95-137 (SEQ ID NO: 5), перекрывающие интроны 2 и 3; праймеры 95-138 (SEQ ID NO: 6) и 95-139 (SEQ ID NO: 7), перекрывающие интроны 3 и 4; праймеры 95-140 (SEQ ID NO: 8) и 95-141 (SEQ ID NO: 9), перекрывающие интроны 4 и 5; праймеры 95-142 (SEQ ID NO: 10) и 95-143 (SEQ ID NO: 11), перекрывающие интроны 6 и 7.
95-136 (SEQ ID NO: 4) GCCACCTGATCTACAAATGT
95-137 (SEQ ID NO: 5) GAGATACCAGCCTCAAATTC
95-138 (SEQ ID NO: 6) ATGTGACCATCATTGATGCC
95-140 (SEQ ID NO: 8) GTTGGAATGGTGACAACATG
95-141 (SEQ ID NO: 9) CAGGTTTTAAAACACCAGTC
95-142 (SEQ ID NO: 10) AATGACCCACCAATGGAAGC
95-143 (SEQ ID NO: 11) ACAGCAACTGTCTGCCTCAT
Предполагаемый размер продуктов ПЦР при использовании матричной ДНК СНО EF-1α . был обоснован размером и расположением интронов в опубликованной человеческой последовательности EF-1α . Каждая ПЦР включала 2 мкл фага, 2,5 мкл каждого праймера из соответствующей пары (100 мкг/мл), 2 мкл 2 мМ смеси dNTP (дезоксинуклеозидтрифосфатов), 2,5 мкл 10х буфера для ПЦР (фирмы Roche Molecular Systems, Branchburg, NJ), 1,5 мкл 25 мМ хлорида магния, 0,125 мкл полимеразы Taq (5 единиц/мл) (фирмы Perkin Elmer) и 11,8 мкл дистиллированной воды. Амплификацию проводили в течение 4 минут при 94° С, затем 30 циклов по 1 минуте при 90° С, 2 минуты при 50° С и 4 минуты при 72° С. Продукты амплификации разделяли на 1,2% агарозном геле в буфере 1х ТАЕ. Было найдено, что из 47 бляшек 3 кодируют истинные гены (# 2, 7 и 40), содержащие все интроны, три положительных образца были подвергнуты третичному скринингу следующим образом. Культуры для посева получали, как описано выше, при разбавлении 10-105 исходных культур, приготовленных для каждой из трех положительных бляшек. От 20 до 50 выделенных от каждой культуры бляшек скринировали методом ПЦР, получая следующие результаты. Клон #2 дал два положительных образца (обозначенных 2.12 и 2.17) из двадцати скринированных, клон #7 дал один положительный образец (обозначенный 7.44) из пятидесяти скринированных, клон #40 дал один положительный образец (обозначенный 40.24) из сорока скринированных. ДНК фага выделяли из каждого из четырех образцов следующим образом.
Клетки-хозяева линии XL-1-Blue MRA (P2) вносили в планшеты с агарозой LB и выращивали в течение ночи при 37° С. Собирали единичную колонию для того, чтобы инокулировать 50 мл среды LB (содержащей 0,2% мальтозу и 10 мМ сульфат магния), культуру выращивали в течение ночи при 30° С. Клетки собирали центрифугированием в настольной центрифуге при 2000 об/мин в течение 5 минут при комнатной температуре, затем клетки вновь суспендировали в 50 мл 10 мМ сульфата магния. Вновь суспендированные клетки-хозяева (50 мкл) смешивали со 100 мкл основной культуры каждого положительного фага и инкубировали в течение 15 минут при 37° С, давая возможность фагу прикрепиться к клеткам. Прибавляли приблизительно 500 мкл среды LBM, смесь встряхивали в течение 2 часов при 37° С. Прибавляли дополнительные 200 мкл клеток-хозяев, инкубацию продолжали в течение 15 минут при 37° С. Прибавляли верхний слой агара (8 мл 75% агарозы в среде LBM при 48° С), после чего смесь высевали и выращивали в течение ночи при 37° С.
После выращивания в течение ночи в каждый планшет прибавляли λ -разбавитель (10 мМ Трис-HCI, рН 7,5, 10 мМ сульфат магния), планшеты осторожно встряхивали в течение 2 часов. Разбавитель удаляли и центрифугировали в настольной центрифуге в течение 10 минут при 4000g. К каждому 1 мкл супернатанта прибавляли 1 мг/мл РНКазы А и 1 мг/мл ДНКазы I, смесь инкубировали в течение 15 минут при 37° С. Прибавляли равный объем буфера для осаждения (20% PEG 8000 (полиэтиленгликоль), 2 М хлорид натрия, 20 мМ Трис-HCl, рН 7,5, 10 мМ сульфат магния), затем инкубировали во льду в течение одного часа, после чего смесь центрифугировали при 8000 g в течение 20 минут, супернатант удаляли, осадок сушили на воздухе в течение 10 минут при комнатной температуре. Осадок вновь суспендировали в 500 мкл буфера ТЕ, непродолжительно центрифугировали для того, чтобы удалить макрочастицы, супернатант переносили в чистую пробирку на 1,65 мл. Прибавляли приблизительно 2,5 мкл 20% SDS (с пятиминутной инкубацией при 65° С), затем прибавляли 2,5 мкл при концентрации 10 мг/мл протеиназы К (с инкубацией в течение 1 часа при 65° С) и 10 мкл 5 М раствора хлорида натрия. Смесь экстрагировали один раз равным объемом смеси фенол-хлороформ и один раз равным объемом хлороформа. Прибавляли равный объем изопропанола, затем инкубировали при -70° С в течение 3 часов, после чего смесь центрифугировали в течение 15 минут с предельной скоростью. Полученный осадок промывали 70% этанолом, сушили на воздухе и вновь суспендировали в 100 мкл буфера ТЕ.
Пример 2
Субклонирование регуляторной последовательности EF-1α
Для того чтобы определить размер включенной последовательности ДНК EF-1α , ДНК фага, полученную, как описано в примере 1, расщепляли с помощью рестриктазы NotI, полученные рестрикционные фрагменты разделяли на агарозном геле в 0,6% 1хТАЕ (Трис-ацетат-ЭДТА). Клоны 2.12 и 2.17 расщеплялись по одинаковому образцу с появлением полос в 11 т. н. и 4,5 т. н. помимо ожидаемых фланкирующих λ -фрагментов в 19 т.н. и 10 т.н. Клоны 7.44 и 40.24 также расщеплялись по одному образцу с появлением полос в 12 т.н. и 7 т.н., что в совокупности с данными расщепления клонов 2.12 и 2.17 указывало на наличие внутреннего сайта рестрикции NotI в последовательности ДНК EF-1α . Фрагменты включения клонов 2.12 и 7.44 субклонировали следующим образом.
ДНК фага (60 мкл), полученную, как описано в примере 1, расщепляли рестриктазой NotI, после чего расщепленную ДНК осаждали добавлением 20 мкл 3 М раствора ацетата натрия и 400 мкл 100% этанола. Осажденную ДНК собирали с помощью центрифугирования, промывали 200 мкл 70% этанола, сушили на воздухе в течение 15 минут, вновь суспендировали в 20 мкл ТЕ, нагревали при 65° С в течение 10 минут, затем прибавляли 2 мкл загружающего буфера для электрофореза. ДНК разделяли, используя гель-электрофорез, полосы 4.5 т.н., 7 т.н., 11 т.н. и 12 т.н. выделяли в виде срезов агарозы. ДНК экстрагировали из каждого среза, используя набор QlAquick фирмы QIAGEN для экстракции геля в соответствии с предложенным производителем протоколом. Чистоту полосы и концентрацию определяли путем разделения аликвоты в 5 мкл каждого выделенного фрагмента в агарозном геле в 0,6% 1х ТАЕ.
Индивидуальные фрагменты по отдельности лидировали в расщепленный рестриктазой Notl вектор pBluescript SW+. При лигировании фрагментов 11 и 12 т.н. линеаризованный вектор перед введением фрагмента-вставки обрабатывали щелочной фосфатазой. Использовали 2 мкл каждого лигирования для электропорации 40 мкл электрокомпетентных клеток XL-1 Blue. Трансформированные клетки высевали на пластины с LBM/carb-агарозой с 40 мкл 5% X-gal (5-бром-4-хлор-3-индолил-β -D-галактопиранозида) в диметилформамиде (DMF) и 20 мкл 0,1 М IPTG (изопропил-β -D-тиогалактопиранозида) на пластину. Клетки инкубировали в течение ночи при 37° С, наутро пластины охлаждали до 4° С для усиления интенсивности голубой окраски.
При вторичном скрининге белые клоны окрашивали на пластинах с LBM/carb-агарозой, содержащих Х-gаl и IPTG, как описано выше, клоны, которые оставались белыми на следующий день, выращивали в 3 мл LBM/carb в течение ночи при 37° С. Плазмидную ДНК из белых клонов, выращенных в течение ночи, получали, используя систему очистки ДНК WIZARD Plus Minipreps (фирмы Promega, Madison, Wl) в соответствии с предложенным производителем протоколом.
Рестрикционный анализ выделенной плазмидной ДНК свидетельствовал о том, что фрагменты 4,5 т.н., 7 т.н. и 12 т.н. были успешно лигированы в вектор pBluescript SW+, полученные плазмиды обозначили pSK/EF1.4.5, pSK/EF1.7 и pSK/ЕП.12 соответственно.
Каждую плазмиду затем получали, используя набор миди-преп фирмы QIAGEN в соответствии с предложенным производителем протоколом. ПЦР проводили на каждом новом векторе, титруя матричную ДНК и используя праймеры кодирующего участка (SEQ ID NO: 4-11, применяемые парами, как описано выше, для скрининга псевдогенов, лишенных одного или более интронов). Титрование проводили после того, как было обнаружено, что при высоких концентрациях все три фрагмента включают полностью кодирующий участок EF-1α , что подразумевало возможное перекрестное загрязнение трех плазмидных препаратов. Для титрования ПЦР проводили в условиях, содержащих 2,5 мкл матричной ДНК в концентрациях 0,001, 0,01, 0,1, 1 или 10 нг/мкл, 2,5 мкл 10х ПЦР-буфера (фирмы Perkin-Elmer), 2,0 мкл 2 мМ смеси dNTP, 1,5 мкл 25 мМ хлорида магния, 0,125 мкл полимеразы Тао (фирмы Perkin-Elmer) и 11,4 мкл дистиллированной воды. Амплификацию проводили при следующих условиях: 4 минуты при 94° С, затем 30 циклов по 1 минуте при 90° С, 2 минуты при 50° С, 4 минуты при 72° С. Результаты указывали на то, что весь кодирующий участок EF-1α находится внутри фрагментов в 4,5 т.н. и 7 т.н.
Рестрикционную карту для каждой вставки составляли с использованием набора нерадиоактивного картирования генов FLASH (фирмы Stratagene), предназначенного для применения с вектором λ FIX II®. Однако вместо ДНК фага использовали плазмидную ДНК, вырезанную с помощью рестриктазы NotI из векторов pSK, описанных выше. По существу протокол картирования не отличался от предложенного производителем. В кратком виде, плазмиды секвенировали с обратными праймерами М13 (SEQ ID NO: 12) и обратными праймерами М13 (SEQ ID NO: 13) (комплементарными участкам, находящимся внутри последовательности множественного клонирования вектора pBluescript SW+) для того, чтобы локализовать для каждой вставки праймерные последовательности ТЗ (SEQ ID NO: 14) и Т7 (SEQ ID NO: 15), являющиеся внутренними по отношению к сайту рестриктазы NotI.
М13 GTAAAACGACGGCCAGT SEQ ID NO: 12
M13rev GGAAACAGCTATGACCATG SEQ ID NO: 13
Т3 AATTAACCCTCACTAAAGGG SEQ ID NO: 14
T7 GTTAATACGGACTCACTATAGGGC SEQ ID NO: 15
Праймеры Т7 и Т3 использовали в качестве зондов в протоколе картирования генов. Поскольку было показано, что вставка EF-1α включает внутренний сайт NotI, предполагалось, что пары фрагментов (4,5 т.н./11 т.н. и 7 т.н./12 т.н.) включают одну или другую праймерную последовательность, и, таким образом, установлено, что вставки 4,5 и 12 т.н. включают праймерную последовательность T7, тогда как вставка 7 т.н. содержит последовательность Т3.
Вставки 4,5 т.н. и 7 т.н., содержащие кодирующий участок последовательности EF-1α , вырезали из вектора с помощью рестриктазы NоtI. Отщепленную ДНК разделяли на агарозном геле, отделенные фрагменты срезали с геля и ДНК выделяли из каждого среза, используя набор для экстракции геля QIAquick фирмы QIAGEN в соответствии с предоставленным изготовителем протоколом. Картирование осуществляли, используя семь различных ферментов и продукты частичной рестрикции. Продукты реакции разделяли на агарозном геле, ДНК переносили на нейлоновую мембрану Duralon-UV и использовали в качестве зонда олигонуклеотид Т3 или Т7. Оценивали параметры полос и создавали рестрикционные карты.
Плазмиду pSK/EF-1.7 скринировали с внутренними праймерами, первоначально созданными для скрининга с помощью ПЦР (SEQ ID NO: 4-11, ранее описанные праймеры 95-136-95-143), чтобы быть уверенными в том, что генная последовательность отвечает ранее идентифицированному белку. Определяли ориентацию кодирующего участка и создавали дополнительные праймеры, что позволило осуществить секвенирование до внутреннего сайта NotI. Определяли последовательность всего кодирующего участка и сравнивали ее с ожидаемой последовательностью кДНК, описанной Hayashi и др. (см. выше). Анализ последовательности подтвердил, что выделенная ДНК действительно кодирует такие же последовательности EF-1α , как описанные Hayashi и др., (см. выше). Кроме того, последовательность первого экзона была идентична ранее опубликованной 5’-концевой последовательности кДНК хомячковой EF-1α , (Hayashi и др., см. выше), были идентифицированы семь интронов, образующие структуру, подобную таковой для человеческого гомолога.
Последовательность и рестрикционная карта, полученные от фрагмента в 7 т.н., указывали на то, что часть 5’-фланкирующего интрона и промотор локализованы во фрагменте в 12 т.н. в 5’-положении по отношению к внутреннему сайту NotI. Фрагмент SpeI/NotI в 3 т.н. в 5’-положении по отношению к вну