Способ экспрессного определения устойчивости к рифампицину у микобактерий туберкулеза
Иллюстрации
Показать всеИзобретение относится к фтизиобактериологии и может быть использовано для лабораторной диагностики множественно-лекарственного туберкулеза. Для решения поставленной задачи определяют специфические мутации в гене rpoB полимеразной цепной реакцией с аллель-специфическими праймерами для кодонов 516, 526, 531. При отсутствии мутаций в данном кодоне происходит кодон-зависимая аллель-специфическая ПЦР-амплификация фрагмента гена rpoB; наличие мутации в позиции, соответствующей 3'-концу праймера, ведет к неспариванию праймера и матрицы и неспособности полимеразы к синтезу комплементарного фрагмента. Таким образом, наличие мутации (RIF-резистентного фенотипа) приводит к отсутствию индикаторного фрагмента. Одновременно проводят три реакции ПЦР, направленные на три кодона 516, 526, 531, а результат ПЦР анализируют электрофорезом в горизонтальном агарозном мини-геле. При отсутствии одного из индикаторных фрагментов 167, 181, 214 пн судят о наличии мутации и устойчивости микобактерий к рифампицину. Использование способа позволяет быстро и надежно определять устойчивость микобактерий туберкулеза к рифампицину как маркеру полирезистентности. 3 ил.
Реферат
Изобретение относится к медицине, а именно к фтизиобактериологии, и может быть использовано для лабораторной диагностики множественно-лекарственно-устойчивого туберкулеза.
Эффективное применение современных схем лечения туберкулеза затруднено селекцией и циркуляцией штаммов Mycobacterium tuberculosis, устойчивых к основным противотуберкулезным препаратам 1 ряда, в частности к рифампицину (RIF), изониазиду (INH), стрептомицину (STR). Множественно-лекарственно-устойчивыми (МЛУ, англ. - MDR, multiple-drug resistant) определяют штаммы МБТ, устойчивые к RIF и INH. Поскольку устойчивость к рифампицину сопровождает, как правило, резистентность к INH, этот препарат служит маркером мультирезистентности, и определение устойчивости к RIF имеет наибольшее значение для выбора адекватной терапии. Традиционные фенотипические методы определения лекарственной чувствительности требуют больших затрат времени (до 1,5 месяцев) и даже внедрение более современных методик (например, Bactec - 6-10 дней) не позволяет обеспечить действительно экспрессный анализ. Применение молекулярно-генетических методов исследования генетических детерминант устойчивости, не требующих длительного выращивания чистой культуры, позволяет решить эту проблему. Показано, что у 90-98% RIF-устойчивых штаммов резистентность связана с мутациями в гене rpoB, который кодирует бета-субъединицу ДНК-зависимой РНК-полимеразы (мишенью для RIF в микробной клетке). 95-98% Мутаций в rpoB расположено в коротком участке 81 пн (кодоны 507-533 [1]), при этом более 85% мутаций выявлено в трех кодонах 516, 526, 531 [2]. Применяемые в настоящее время многочисленные методы для определения мутаций в rpoB либо определяют факт их наличия (PCR-SSCP [З], RNA/RNA mismatch [4]) либо выявляют конкретные мутации (дот-блот [5], InnoLIPA [6], микрочипы [7]), и при этом золотым стандартом остается прямое секвенирование ДНК. Недостатками указанных методов являются большая или меньшая трудоемкость и длительность, сложность и/или высокая стоимость оборудования и не всегда превосходная воспроизводимость и интерпретируемость результата [3, 8].
В качестве прототипа предлагается метод полимеразной цепной реакции фрагмента гена rpoB в "реальном времени".(метод "молекулярных маячков" [9]); при этом внутренний олигонуклеотид взаимодействует с продуктом ПЦР только при отсутствии в нем мутаций резистентности и выделяет флуоресцентное излучение. Этот метод требует специальный термоциклер, который в 3-6 раз превышает по цене оборудование для стандартной ПЦР и агарозного гель-электрофореза.
Задачей предлагаемого изобретения является разработка способа быстрой и надежной детекции устойчивости микобактерий туберкулеза к рифампицину на основе выявления мутаций в трех кодонах в гене rpoB.
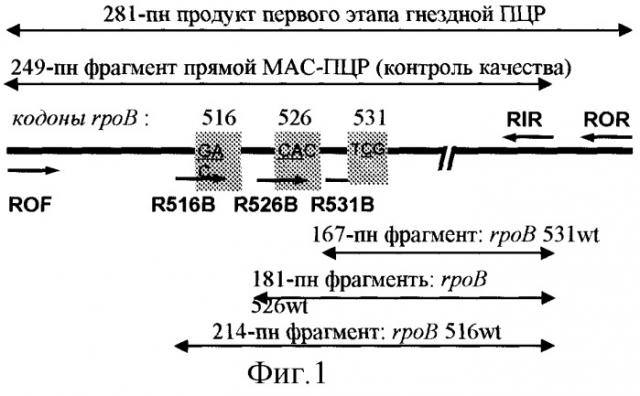
Поставленная задача достигается определением специфических мутаций в гене rpoB полимеразной цепной реакцией с аллель-специфическими праймерами для кодонов 516, 526, 531. При отсутствии мутаций в данном кодоне происходит кодон-зависимая аллель-специфическая амплификация фрагмента гена rpoB, наличие мутации в позиции, соответствующей 3'-концу праймера, ведет к неспариванию праймера и матрицы и неспособности полимеразы к синтезу комплементарного фрагмента. Таким образом, наличие мутации (RIF-резистентного фенотипа) приводит к отсутствию индикаторного фрагмента. Одновременно проводится три реакции ПЦР, направленные на три кодона 516, 526, 531. Прямые аллель-специфические праймеры (R516B, R526B, R531B) соответствуют по своим 3'-концам вторым основаниям соответствующих кодонов (фиг.1). При этом выявляются мутации в первых и вторых основаниях данных кодонов, что соответствует >85% RIF-устойчивых штаммов [2, 4, 6, 8, 10]. Анализ результатов ПЦР проводится электрофорезом в горизонтальном агарозном мини-геле.
Преимущества предлагаемого способа:
- простое оборудование для стандартной ПЦР и агарозного гель-электрофореза)
- быстрота (один день от момента выделения ДНК)
- простая и однозначная интерпретация результатов
- возможность быстрого анализа больших количеств коллекций ДНК для оценки применимости метода для данной территории.
Способ осуществляется следующим образом.
Выделение ДНК из культуры на среде Левенштейна-Йенсена проводят по van Embden et al. [11] при выделении очищенной ДНК и по Mazars et al. [12] при получении клеточных лизатов. В первом случае суспендируют 1 петлю культуры в общем объеме 400 мкл буфера ТЕ 1х и инкуюируют 20 мин при 85°С. Дальнейшую обработку проводят с использованием лизоцима, протеиназы К, додецилсульфата натрия и цетилтриметиламмонийбромида. Полученный лизат обрабатывают смесью хлороформ-изоамиловый спирт (24:1), центрифугируют, осаждают изопропанолом, промывают 70% этанолом, осадок высушивают и растворяют в 30-50 мкл ТЕ х0.5. При получении грубого препарата ДНК (клеточного лизата) 1 петлю культуры суспендируют в 100 мкл буфера ТЕ 1х, кипятят 20 мин, центрифугируют 15 мин (12000 об/мин), и полученный супернатант используют для ПЦР.
Фиг.1. Схематическое изображение фрагмента гена rpoB анализируемого аллель-специфическими ПЦР. wt (wild type) - дикий аллель соответствующего кодона rpoB. Короткие стрелки обозначают праймеры, длинные двусторонние стрелки представляют продукты ПЦР или постоянные (249 пн) или аллель-специфические (167, 181, или 214 пн). Подчеркнуты основания в кодонах 516 526, 531, где были выявлены мутации.
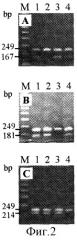
Фиг.2. Анализ препаратов очищенной ДНК из штаммов М. tuberculosis методом МАС-ПЦР. Кодоны rpoB. 531 (А.), 526 (В.), 516 (С.). Дорожки: 1 - H37Rv (дикие аллели гроВ), 2 - штаммы с мутантными аллелями rpoB531 (TCG или TTG), 3 - штаммы с мутантными аллелями rpoB526 (GAC, ТАС, или СТС), 4 - штаммы с мутантными аллелями rpoB516 (GTC, ТАС, или GGC). М - маркер молекулярных весов "100 bp DNA Ladder" (Amersham).
I. Вариант прямой мультиплексной аллель-специфической ПЦР (МАС-ПЦР). Материал для реакции - чистая ДНК достаточно хорошего качества и количества. Проводят три одновременных реакции, направленные на три кодона rpoB и содержащих три праймера: два внешних (одинаковые для всех реакций, ROF и RIR; Фиг.1) и один аллель-специфический праймер (в зависимости от кодона - R516B, R526B или R531B). Продукты ПЦР разделяются агарозным 1.5% гель-электрофорезом. Отсутствие одного из индикаторных фрагментов (167 или 181 или 214 пн) свидетельствует о наличии мутации в кодоне 531 или 526 или 516 соответственно и, следовательно, о RIF-резистентности (Фиг.2). Фрагмент длиной 249 пн амплифицируется внешними консервативными праймерами и служит для контроля качества реакции ПЦР (ложноотрицательных результатов). Следующие праймеры были использованы для трех реакций MAC-ПЦР для анализа трех кодонов гена rpoB: два внешних праймера, прямой ROF (5'-GTCGCCGCGATCAAGGA) и обратный RIR (5'-TGACCCGCGCGTACAC), и внутренние праймеры R516B (5'-GCTGAGCCAATTCATGGA), или R526B (5'-GTCGGGGTTGACCCA), или R531B (5'-ACAAGCGCCGACTGTC). Очищенная ДНК (0.1-0.5 мкл) добавляется к смеси ПЦР (конечный объем 20 мкл), содержащей MgCl2 (3mM для rpoB526- и rpoB531-ПЦР или 1.5 тМ для rpoB516-ПЦР), 1 U rTaq ДНК полимеразы (MBI Fermentas), 200 μМ каждого из дНТФ, внешние праймеры ROF (I pmole) и RIR (20 pmole для rpoB526- и rpoB531-ПЦР, или 10 pmole для rpoB516-ПЦР) и один из аллель-специфических внутренних праймеров R531B (35 pmole) или R526B (30 pmole), или R516B (15 pmole). rpoB526-ПЦР и rpoB531-ПЦР проводили в термоциклере РТС-100 (MJ Research, Inc.) в следующих условиях: 96°С, 3'; 5 циклов 95°С, 45 с, 74°С, 1', 72°С, 20 с; 5 циклов 95°С, 40 с, 73°С, 50 с, 72°С, 20 с; 22 цикла 94°С, 50 с, 70°С, 40 с, 70°С, 20 с; заключительная элонгация 72°С, 3'. Условия rpoB516-ПЦР: 96°С, 3'; 30 циклов 95°С, 50 с, 65°С, 40 с, 72°С, 20 с; 72°С, 3'. Продукты ПЦР (10 μ1) разделяли электрофорезом в 1,5% агарозном геле и визуализировали на УФ-трансиллюминаторе.
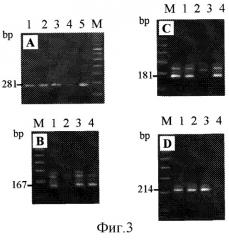
Фиг.3. Анализ клеточных лизатов из штаммов М. tuberculosis методом гнездной аллель-специфической ПЦР (ГАС-ПЦР). А. первая ПЦР для амплификации фрагмента rpoB с внешними праймерами ROF и ROR (фиг.1). В, С, D - анализ трех кодонов rpoB, 531, 526, and 516 соответственно аллель-специфическими ПЦР. Дорожки: 1 - H37Rv (дикие аллели гроВ), 2 - штаммы с мутантными аллелями rpoB53l (TCG или TTG), 3 - штаммы с мутантными аллелями rpoB526 (GAC, ТАС, или СТС), 4 - штаммы с мутантными аллелями rpoB516 (GTC, ТАС, или GGC). М - маркер молекулярных весов "100 bp DNA Ladder" (Amersham).
II. Вариант двушаговой гнездной аллель-специфической ПЦР (ГАС-ПЦР). Материал для реакции - клеточные лизаты (пробы с малым количеством ДНК). Сначала проводится амплификация (внешние праймеры ROF и ROR, фиг.1) большего фрагмента rpoB длиной 281 пн при увеличенном количестве циклов ПЦР. Качество ПЦР проверяется электрофорезом в агарозном мини-геле и визуализируется в УФ. В зависимости от яркости фрагмента в последующую аллель-специфическую ПЦР берется от 1 до 5 мкл продукта первой ПЦР (ср. фиг.3А, дорожки 4 и 5). Далее проводят одновременно три аллель-специфические ПЦР, в которых используют в качестве субстрата продукт первой ПЦР (281 пн), обратный консервативный праймер (RIR) и один из аллель-специфических праймеров (в зависимости от анализируемого кодона) (фиг.1). Отсутствие одного из индикаторных фрагментов (167 или 181 или 214 пн), как и в случае с прямой МАС-ПЦР, говорит о наличии мутации в соответствующем кодоне и RIF-устойчивости (фиг.3B-D). При этом рекомендуется при проведении аллель-специфических ПЦР включать в качестве положительного контроля штамм H37Rv (дикий аллель rpoB) и в качестве отрицательного контроля штамм с известной мутацией в определенном кодоне (516 или 526 или 531). Первая ПЦР с (внешними праймерами) проводится в следующих условиях: 96°С, 3'; 38 циклов (30 - для чистой ДНК)) 95°С, 50 с, 62°С, 40 с, 72°С, 30 с; 72°С, 3'. Очищенный препарат ДНК (0,1-0,5 мкл) или клеточный лизат (7-10 мкл) добавляется в реакционную смесь (конечный объем 30 мкл), содержащую 5 pmole каждого из праймеров ROF и ROR, 1.5 mM MgCl2, 1 U rTaq ДНК полимеразы (MBI Fermentas), и 200 μМ каждого из дНТФ. Амплифицированный 281, пн фрагмент (5 μ|) анализируется в 1,5% агарозном геле, и для примерной оценки количества визуализируется в УФ-трансиллюминаторе. Второй этап ГАС-ПЦР включает три специфические (для дикого аллеля гроВ) реакции ПЦР, проводимые в одинаковых условиях: 96°С, 3'; 5 циклов 95°С, 45 с, 74°С, 30 с, 72°С, 20 с; 5 циклов 95°С, 40 с, 73°С, 40 с, 72°C, 20 с; 12 циклов 95°С, 40 с, 70°С, 40 с, 72°С, 20 с; заключительный синтез 72°С, 3'. Продукт первой ПЦР (от 1 до 5 мкл в зависимости от концентрации - см. Фиг.3А, дорожки 4 и 5) добавляется к смеси ПЦР (конечный объем 20 мкл), содержащей 3 mM MgCl2, 1 U rTaq ДНК полимеразы (MBI Fermentas), 200 μМ каждого из дНТФ, обратный консенсусный праймер RIR (20 pmole для rpoB531- и rpoB526-ПЦР, и 10 pmole для rpoB516-ПЦР) и один из аллель-специфических праймеров R531B (20 pmole), или R526B (20 pmole), или R516B (10 pmole). Продукт ПЦР (10 мкл) анализируется в 1,5% агарозном геле.
Условия обоих вариантов предлагаемого метода были отработаны на 36 штаммах с известным секвенсом rpoB и имевшими следующее распределение аллелей rpoB: дикий тип - 13, 531TTG - 7, 531TGG - 2, 516GTC - 4, 516ТАС - 3, 516GGC - 1, 522CAG - 1, 526ТАС - 2, 526GAC - 3.526CTC - 1.
Пример 1. Анализ 233 проб ДНК RIF-устойчивых (фенотипически) штаммов, выделенных от эпидемиологически несвязанных больных в 1997-2002 гг. (Санкт-Петербург, Ленинградская и Новгородская области) был проведен методом простой одношаговой МАС-ПЦР. Мутации в одном из трех анализировавшихся кодонов rpoB были выявлены в 192 (82.4%) штаммов. Другие исследования, проведенные на Северо-Западе России, показали наличие данных мутаций в 85 из 90 (94.4%) штаммов [4, 10, 13]. Таким образом, этот метод обеспечивает достаточно высокую чувствительность определения устойчивости МБТ к рифампицину. Следует отметить, что генетическое определение устойчивости к RIF на основе мутаций в rpoB в принципе не обеспечивает 100% определение чувствительности, так как не выявляет штаммы, имеющие другие механизмы устойчивости [2]. Анализ методом МАС-ПЦР выявил мутации в rpoB у 2 из 97 RIF-чувствительных штаммов; таким образом, специфичность метода составила 97.9%.
Пример 2. Анализ проб из 27 клеточных лизатов был проведен методом гнездной аллель-специфической ПЦР. Во всех случаях результаты анализа лизатов и очищенной ДНК из тех же штаммов совпадали.
Литература
1. Telenti, A., P.Imboden, F.Marchesi, D.Lowrie, S.Cole, M.J.Colston, L.Matter, K.Schopfer, and T.Bodmer. 1993. Detection of rifampicin-resistant mutations in Mycobacterium tuberculosis. Lancet. 341:647-650.
2. Ramaswami, S.V., and J.M.Musser. 1998. Molecular genetic basis of antimicrobial agent resistance in Mycobacterium tuberculosis: 1998 up date. Tuberc. Lung Dis. 79:3-29.
3. Bobadilla-del-Valle M., Ponce-de-Leon М., Arenas-Huertero C., Vargas-Alarcon G., Kato-Maeda А, Small P.M., Couary P., Ruiz-Palacios G.M., Sifuentes-Osomio J. 2001. rpoB Gene mutations in rifampin-resistant Mycobacterium tuberculosis identified by polymerase chain reaction single-stranded conformational polymorphism. Emerg Infect Dis 7:1010-3.
4. Mokrousov, I., I.Filliol, E.Legrand, C.Sola, T.Otten, E.Vyshnevskaya, E.Limeschenko, B.Vyshnevskiy, O.Narvskaya, and N.Rastogi. 2002. Molecular characterization of multiple-drug-resistant Mycobacterium tuberculosis isolates from North-Western Russia and analysis of rifampin resistance using RNA/RNA mismatch analysis as compared to the line probe assay and sequencing of the rpoB gene. Res. Microbiol. 153:213-219.
5. Victor, Т., A.M.Jordaan, A. van Rie, G.D. van der Spuy, M.Richardson, P.D. van Helden, and R.Warren. 1999. Detection of mutations in drug resistance genes of Mycobacterium tuberculosis by a dot-blot hybridization strategy. Tuberc. Lung Dis. 79:343-348.
6. De Beenhouwer H., Z.Lhiang, G.Jannes, W.Mijs, L.Machtelinckx, R.Rossau, H.Traore, and F.Portaels. 1995. Rapid detection of rifampicin resistance in sputum and biopsy specimens from tuberculosis patients by PCR and line probe assay. Tubercle Lung Dis. 76:425-430.
7. Mikhailovich, V., S.Lapa, D.Gryadunov, A.Sobolev, B.Strizhkov, N.Chernyh, O.Skotnikova, O.Irtuganova, A.Moroz, V.Litviniv, M.Vladimirskii, M.Perelman, L.Chernoussova, V.Erokhin, A.Zasedatelev, and A.Mirzabekov. 2001. Identification of rifampin-resistant Mycobacterium tuberculosis strains by hybridization, PCR, and lygase detection reaction on oligonucleotide microchips. J. Clin. Microbiol. 39:2531-2540.
8. van Rie, A., R.Warren, I.Mshanga, A.M.Jordaan, G.D. van der Spuy, M.Richardson, J.Simpson, R.P.Gie, D.A.Enarson, N.Beyers, P.D.van Helden, and Т.С.Victor. 2001. Analysis for a limited number of gene codons can predict drug resistance of Mycobacterium tuberculosis in a high-incidence community. J. Clin. Microbiol. 39:636-641.
9. Piatek, A.S., A.Telenti, M.R.Murray, H.El-Hajj, W.R.Jacobs, Jr., F.R.Kramer, and D.Alland. 2000. Genotypic analysis of Mycobacterium tuberculosis in two distinct populations using molecular beacons: implication for rapid susceptibility testing. Antimicrob. Agents Chemother. 44:103-110.
10. Toungoussova, O., P.Sandven, A.Mariandyshev, N.Nizovtseva, G.Bjune, and D.A.Caugant. 2002. Spread of drug-resistant Mycobacterium tuberculosis strains of the Beijing genotype in the Archangel Oblast, Russia. J. Clin. Microbiol. 40:1930-1937.
11. van Embden, J.D.A., M.D.Cave, J.T.Crawford, J.W.Dale, K.D.Eisenach, B.Gicquel, P.Hermans, C.Martin, R.McAdam, T.M.Shinnik, et al. 1993. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J. Clin. Microbiol. 31:406-409.
12. Mazars E., Lesjean S., Banuls A.-L., Gilbert M., Vincent V., Gicquel В., Tibayrenc M., Locht C., Supply P. 2001. High-resolution mini-satellite-based typing as a portable approach to global analysis of Mycobacterium tuberculosis molecular epidemiology, Proc. Natl. Acad. Sci. USA. 98:1901-1906.
13. Marttila, H.J., H.Soini, E.Eerola, E.Vyshnevskaya, B.I.Vyshnevskiy, T.F.Otten, A.V.Vasilyef, and M.K.Viljanen. 1998. A Ser315Thr substitution in KatG is predominant in genetically heterogeneous multidrug-resistant Mycobacterium tuberculosis isolates originating from the St.Petersburg area in Russia. Antimicrob. Agents Chemother. 42:2443-2445.
Способ экспрессного определения устойчивости к рифампицину у микобактерий туберкулеза путем определения специфических мутаций в гене rpoB, отличающийся тем, что применяют полимеразную цепную реакцию с аллель специфическими праймерами для кодонов 516, 526, 531 и при отсутствии одного из индикаторных фрагментов 167, 181, 214 пн судят о наличии мутации и устойчивости микобактерий к рифампицину.