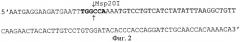Способ идентификации полиморфизма +1385g/a гена rnasel
Иллюстрации
Показать всеИзобретение относится к области медицины и молекулярной биологии и может быть использовано для идентификации полиморфизма +1385G/A гена RNASEL при обследовании больных вирусными инфекциями. Способ включает амплификацию участка длиной 109 п.н. гена RNASEL путем проведения полимеразной цепной реакции с использованием комплементарного праймера 5′AATGAGGAAGATGAATTTGGC3′ и антикомплементарного праймера 5′TGTTTTGTGGTTGCAGATCC3′, рестрикцию продуктов амплификации с помощью эндонуклеазы Msp201, электрофоретическое разделение полученных фрагментов и выявление рестриктов с длиной 20 и 89 п.н., свидетельствующих о наличии аллелей полиморфизма +1385G/A гена RNASEL. Способ позволяет с высокой точностью определить полиморфизм +1385G/A гена RNASEL. Экономичность способа позволяет использовать его при скрининговых исследованиях. 3 ил.
Реферат
Изобретение относится к области медицины и молекулярной биологии и может быть использовано для идентификации полиморфизма +1385G/A гена RNASEL при обследовании больных вирусными инфекциями.
Ген, кодирующий синтез белка рибонуклеазы L, находится на хромосоме 1q25 [Squire J, Zhou A, Hassel BA. et al. Localization of the interferon-induced, 2-5A-dependent RNase gene (RNS4) to human chromosome 1q25. Genomics. 1994 Jan 1; 19 (1): 174-5]. Описан целый ряд полиморфизмов данного гена: G59S, I97L, I220V, G296V, S322F, R462Q, Y529C и D541E, среди которых только R462Q (+1385G/A) вариант, впервые описанный [Rokman A, Ikonen Т, Seppala EH. et al. Germline alterations of the RNASEL gene, a candidate HPC1 gene at 1q25, in patients and families with prostate cancer. Am J Hum Genet. 2002 May; 70 (5): 1299-304], связан со снижением активности фермента в три раза [Casey G, Neville PJ, Plummer SJ. et al. RNASEL Arg462Gln variant is implicated in up to 13% of prostate cancer cases. Nat Genet. 2002 Dec; 32 (4): 581-3], остальные варианты по активности рибонуклеазы L не отличались от нормального аллеля. Замена аргинина глутамином, обусловленная полиморфизмом +1385G/A гена RNASEL, приводит к снижению способности образования активной димерной формы рибонуклеазы L из неактивной мономерной [Xiang Y, Wang Z, Murakami J. et al. Effects of RNase L mutations associated with prostate cancer on apoptosis induced by 2′,5′-oligoadenylates. Cancer Res. 2003; 60: 6795-801].
Имеющиеся к настоящему времени сведения о значении рибонуклеазы L для противовирусного ответа и апоптоза и наличие функционального полиморфизма в позиции +1385 делает анализ генотипа по этому маркеру крайне актуальным.
Для идентификации полиморфизма +1385G/A гена RNASEL предложен способ пиросеквенирования [Wang L, McDonnell SK, Elkins DA, Slager SL, Christensen E, Marks AF, Cunningham JM, Peterson BJ, Jacobsen SJ, Cerhan JR, Blute ML, Schaid DJ, Thibodeau SN. Analysis of the RNASEL gene in familial and sporadic prostate cancer. Am J Hum Genet. 2002 Jul; 71 (1): 116-23]. Способ предусматривает амплификацию участка гена RNASEL, разделение полученного 145 - п.н. фрагмента на два одноцепочечных фрагмента с помощью магнитных частиц Dynabeads, монопольным производителем которых является фирма Dynal AS, и последующее пиросеквенирование на аппарате PSQ96 (Pyrosequencing) производства компании Biotage AB. Данный способ позволяет с высокой точностью определить полиморфизм +1385G/A гена RNASEL, однако является дорогостоящим и вследствие этого абсолютно непригоден для низкобюджетного скрининга.
Известен способ аллель-специфической амплификации [Casey G, Neville PJ, Plummer SJ, Xiang Y, Krumroy LM, Klein EA, Catalona WJ, Nupponen N, Carpten JD, Trent JM, Silverman RH, Witte JS. RNASEL Arg462Gln variant is implicated in up to 13% of prostate cancer cases. Nat Genet. 2002 Dec; 32 (4): 581-3], согласно которому используют аллельспецифические праймеры 3′, участки которых комплементарны к аллелям гена RNASEL, кодирующим А и G соответственно.
Согласно данным экспертов Международной федерации клинической химии и лабораторной медицины [Nollau P, Wagener С. Methods for detection of point mutations: performance and quality assessment. IFCC Scientific Division, Committee on Molecular Biology Techniques. Clin Chem. 1997 Jul; 43 (7): 1114-28], серьезным ограничением в использовании этого способа является возможность получения ложноположительных и ложноотрицательных результатов. Характерным является трудность подбора и поддержания условий амплификации (концентрации ионов магния, температуры отжига праймеров).
Технический результат, достигаемый изобретением, заключается в повышении точности способа при минимальных финансовых затратах.
Сущность изобретения заключается в достижении указанного технического результата в способе идентификации полиморфизма +1385G/A гена RNASEL, предусматривающем амплификацию участка длиной 109 н.п. гена RNASEL путем проведения полимеразной цепной реакции с использованием комплементарного праймера 5′AATGAGGAAGATGAATTTGGC3′ и антикомплементарного праймера 5′TGTTTTGTGGTTGCAGATCC3′, рестрикцию продуктов амплификации с помощью эндонуклеазы Msp20l, электрофоретическое разделение полученных фрагментов и выявление аллелей полиморфизма +1385G/A гена RNASEL.
Участок гена, в котором локализован полиморфизм, не содержит сайта рестрикции ни одной из имеющихся в настоящее время эндонуклеаз. Существующие компьютерные программы по подбору праймеров не имеют функции внедрения искусственного сайта рестрикции и не позволяют произвести подбор олигонуклеотидов-праймеров для амплификации искомого фрагмента гена вследствие используемого алгоритма учета соотношения GC оснований. В связи с этим последовательность праймера определена вручную с созданием искусственного сайта рестрикции в области 3′ участка форвардного праймера.
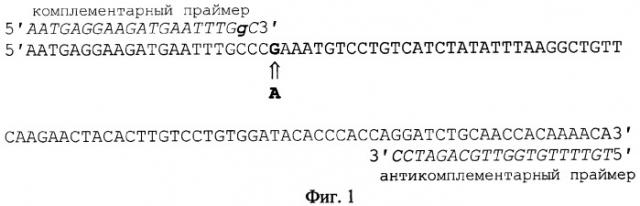
На фиг.1 представлена схема амплификации фрагмента гена RNASEL; на фиг.2 - схема рестрикционного анализа вариантного аллеля +1385А; на фиг.3 - картина электрофоретического разделения рестрикционных фрагментов участка гена RNASEL.
Амплификациию участка длиной 109 н.п. гена RNASEL (каталог GenBank L10381, пары нуклеотидов с 1466 по 1574), где локализован полиморфизм +1385G/A, проводят методом полимеразной цепной реакции с использованием комплементарного праймера 5′AATGAGGAAGATGAATTTGgC3′, предпоследний нуклеотид которого заменен на G для создания искусственного сайта рестрикции в ампликоне (фиг.1). При наличии А в позиции +1385 гена RNASEL фермент Msp20l, имеющий сайт узнавания 5′tgg/cca3′, расщепляет ампликон на два фрагмента 20 и 89 п.н. (фиг.2). При наличии G в позиции +1385 ампликон интактен к действию эндонуклеазы.
Метод ПЦР-ПДРФ является «золотым стандартом» для идентификации биаллельных полиморфизмов и по заключению экспертов Международной федерации клинической химии и лабораторной медицины [Nollau P, Wagener С. Methods for detection of point mutations: performance and quality assessment. IFCC Scientific Division, Committee on Molecular Biology Techniques. Clin Chem. 1997 Jul; 43 (7): 1114-28] обладает 100% специфичностью. При этом он не требует больших экономических затрат.
Способ осуществляют, например, следующим образом.
ПЦР проводят в объеме 20 мкл на амплификаторе Терцик («ДНК-Технология», Россия). Реакционная смесь содержит 200 мкМ каждого из четырех дезоксинуклеозидтрифосфатов, 1 единицу Taq полимеразы, 2 мМ MgCl2, 16 мМ (NH4)2SO4, 65 мМ Tris-HCl, pH 8,8, 0,01% Твин-20, 0,25 мкМ каждого праймера 5′AATGAGGAAGATGAATTTGGC3′ и 5′TGTTTTGTGGTTGCAGATCC3′ и 10-50 нг исследуемой пробы ДНК. ПЦР-программа включает предварительную денатурацию при 94°С 4 минуты, 30 циклов в режиме: денатурация при 94°С - 1 минута, отжиг праймеров при 58°С 1 минута, элонгация при 72°С 1 минута и финальную инкубацию при 72°С 10 минут.
Для рестрикционного анализа используют эндонуклеазу рестрикции Msp20l производства ООО «Сибэнзим», Россия в соответствии с рекомендациями, содержащимися в аналитическом паспорте фермента. К 20 мкл амплификата добавляют 2 е.а. фермента, 3 мкл 10-кратного буфера SE-буфера W (10 mM Tris-HCl (рН 8.5 при 25°С); 10 mM MgCl2; 100 mM NaCl; 1 mM DTT), 0,3 мкл BSA (10 мг/мл) и 6,7 мкл бидистиллированной воды. Содержимое пробирок перемешивают, ценрифугируют и инкубируют при +37°С в суховоздушном термостате в течение 16 часов.
После проведения рестрикционного анализа фрагменты ДНК разделяют электрофоретически в 8% полиакриламидном геле. Разделение фрагментов осуществляют при напряжении 150 В до момента достижения маркерным красителем (бромфеноловым синим) нижнего края геля. Гель помещают на 5-10 минут в раствор, содержащий 0,5 мг/мл бромида этидия, промывают водой, после чего визуализируют фрагменты ДНК в проходящем ультрафиолетовом свете.
На фиг.3 представлены результаты электрофореза с использованием системы для документации гелей Gel Doc XR System (Bio-Rad Laboratories, США). Дорожка 3 - фрагмент 109 п.н., гомозигота G/G. Дорожки 4, 6, 7 - фрагменты 109, 89 и 20 п.н., гетерозиготы G/A. Дорожки 2 и 5 - фрагменты 89 и 20 п.н., гомозиготы А/А. Дорожки 1, 8 - ДНК-маркер 100 bp (10 фрагментов от 100 до 1000 bp).
Использование заявленного способа позволяет с высокой точностью определить полиморфизм +1385G/A гена RNASEL. Экономичность способа позволяет использовать его при скрининговых исследованиях.
Способ идентификации полиморфизма +1385G/A гена RNASEL, предусматривающий амплификацию участка длиной 109 п.н. гена RNASEL путем проведения полимеразной цепной реакции с использованием комплементарного праймера 5′AATGAGGAAGATGAATTTGGC3′ и антикомплементарного праймера 5′TGTTTTGTGGTTGCAGATCC3′, рестрикцию продуктов амплификации с помощью эндонуклеазы Msp201, электрофоретическое разделение полученных фрагментов и выявление рестриктов с длиной 20 и 89 п.н., свидетельствующих о наличии аллелей полиморфизма +1385G/A гена RNASEL.