Способ диагностики аллелей t30n, s52g, i199v и r200q гена prkag3
Иллюстрации
Показать всеИзобретение относится к молекулярной генетике. Предложен способ определения полиморфизма гена - маркера мясной продуктивности свиней PRKAG3. Изобретение применимо в молекулярной генетике для диагностики аллелей с целью использования их в селекции животных. 5 ил.
Реферат
Изобретение относится к молекулярной генетике, в частности к способам определения полиморфизма генов - маркеров мясной продуктивности свиней.
Ген гамма-субъединицы протеинкиназы А (PRKAG3, RN-ген) впервые был открыт у свиней породы гемпшир. Он локализован на хромосоме 15 и имеет размер 5888 п.о., состоит из 11 экзонов и 10 интронов и кодирует пропептид длиной 464 аминокислоты (Milan, D., Jeon, J.T., Looft, C., Amarger, V., Robic, A., Thelander, M., Rogel-Gaillard, C., Paul, S., Iannuccelli, N., Rask, L., Ronne, H., Lundstrom, K., Reinsch,N., Gellin, J., Kalm, E., Roy, P.L., Chardon, P. and Andersson, L. A mutation in PRKAG3 associated with excess glycogen content in pig skeletal muscle. // Science, 2000, 288, 1248-1251). В настоящее время известно 8 генетически обусловленных аллельных вариантов гена PRKAG3 - T30N, S52G, P53L, молчащие мутации в позиции 193 (GCT→GCC) и в позиции 194 (TTG→CTG), I199V, R200Q и молчащая мутация в позиции 372 (ТСС→ТСТ). Наиболее значимые с точки зрения селекции свиней мутации T30N, S52G, I199V, R200Q.
Известен способ определения полиморфизма PRKAG3 методом прямого секвенирования продуктов ПЦР (Cheung, Р.С.F., Salt, I.P., Davies, S.P., Hardie, D.G., Carling, D. Characterization of AMP-activated protein kinase gamma-subunit isoforms and their role in AMP binding. // Biochem. J., 2000, 346, 659-669), однако данный способ характеризуется большими временными и материальными затратами и, кроме того, требует использования дорогостоящего оборудования (ДНК-секвенаторов). Все это делает его непригодным для рутинного практического применения.
Известен способ определения полиморфизма гена PRKAG3 методом ПЦР в режиме реального времени, однако наряду с необходимостью использования дорогостоящего оборудования данный метод характеризуется высокими материальными затратами, требует проведения реакций в особых оптически прозрачных пробирках, а также использования праймеров и зондов, меченных флуоресцентными метками (Milan, D., Jeon, J. - T., Looft, С., Amarger, V., Robic, A., Thelander, M., Rogel-Gaillard, C., Paul, S., Iannuccelli, N., Rask, L., Ronne, H., Lundstrom, K., Reinsch, N., Gellin, J., Kalm, E., Le Roy, P., Chardon, P., Andersson, L.: A mutation in PRKAG3 associated with excess glycogen content in pig skeletal muscle. // Science, 2000, 288, 1248-1251). Все это ограничивает возможность рутинного использования метода как в исследованиях, так и в практике.
Известен способ определения вариантов гена PRKAG3 методом ПЦР-ПДРФ-анализа, взятый в качестве прототипа (Meadus W.J., MacInnis R., Dugan M.E. R., Aalhus J.L. A PCR-RFLP method to identify the RN - gene in retailed pork chops. // Can J. Anim. Sci, 2002, 82, 449-51). Данный способ основан на амплификации двух фрагментов гена PRKAG3, содержащих исследуемые мутации и последующим гидролизе этих фрагментов эндонуклезами рестрикции и дальнейшим разделением фрагментов в 3%-агарозном геле. Это позволяет проводить генотипирование животных с относительно невысокими материальными затратами. Однако данный способ характеризуется трудоемкостью, сложностью интерпретации результатов и большими временными затратами.
Задача изобретения заключалась в расширении арсенала способов диагностики мутаций T30N, S52G, I199V и R200Q гена PRKAG3 для определения генотипов животных по данному гену, снижении трудоемкости и временных затрат предлагаемого способа.
Технический результат изобретения достигается тем, что предложен способ диагностики аллелей T30N, S52G, I199V и R200Q гена PRKAG3, включающий амплификацию двух фрагментов - фрагмента 1 гена PRKAG3 длиной 270 п.о., включающего часть экзона 1 и содержащего специфические мутации в позиции 30 и 52 аминокислотной последовательности, и фрагмента 2 гена PRKAG3 длиной 118 п.о., включающего часть экзона 3 и специфические точковые мутации в позиции 199 и 200 аминокислотной последовательности, с использованием биотинилированных реверсивных праймеров:
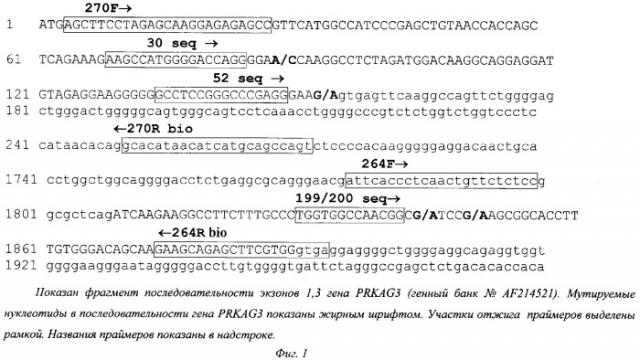
270 F (позиции 8242 до 8266) AGCTTCCTAGAGCAAGGAGAGAGCC
270 R bio (позиции 8321 до 8342) ACTGGCTGCATGATGTTATGTGC
264 F (позиция 8196 до 8218) ATTCACCCTCAACTGTTCTCTCC
264 R bio (позиция 8376 до 8396) TCACCCACGAAGCTCTGCTTC
Особенностью которого является то, что последующее генотипирование продуктов реакции проводят посредством пиросеквенирования с использованием генетических зондов:
30 Seq (позиция 69 до 85) AAGCCATGGGGACCAGG
52 Seq (позиция 135 до 150) GCCTCCGGGCCCGAGG
199/200 Seq (позиция 1831 до 1843) TGGTGGCCAACGG
зондов 30 Seq и 52 Seq в мультиплексной системе генотипирования для фрагмента 1 и зонда 199/200 Seq в симплексной системе анализа для фрагмента 2.
Нами были подобраны две пары специфических праймеров и 3 секвенирующих зонда (генный банк №АF214521, www.ncbi.nlm.nih.gov) фиг.1:
270 F (позиции 8242 до 8266) AGCTTCCTAGAGCAAGGAGAGAGCC
270 R bio (позиции 8321 до 8342) ACTGGCTGCATGATGTTATGTGC
264 F (позиция 8196 до 8218) ATTCACCCTCAACTGTTCTCTCC
264 R bio (позиция 8376 до 8396) TCACCCACGAAGCTCTGCTTC
30 Seq (позиция 69 до 85) AAGCCATGGGGACCAGG
52 Seq (позиция 135 до 150) GCCTCCGGGCCCGAGG
199/200 Seq (позиция 1831 до 1843) TGGTGGCCAACGG
Теоретические выкладки определения полиморфизма гена PRKAG3 в позициях T30N, S52G и I199V, R200Q методом пиросеквенирования в виде гистограмм представлены на фиг.2 и 3.
Пример. Контрольное использование предложенного способа для идентификации генотипов гена PRKAG3 было проведено на 100 свиньях пород йоркшир, ландрас и дюрок («Троснянский бекона. Орловская обл.). ДНК выделяли из ткани методом Кавасаки по ранее опубликованной методике (Зиновьева Н.А., Попов А.Н., Эрнст Л.К. и др. Методические рекомендации по использованию метода полимеразной цепной реакции в животноводстве. Дубровицы, 1998, 47 с.).
1. ПЦР1 и ПЦР2 проводили в конечном объеме 40 мкл с использованием праймеров 270 F и 270 R Bio в ПЦР1 и 264 F и 264 R Bio в ПЦР2 в количестве по 10 пкмоль каждого в стандартном температурно-временном режиме.
2. В продукты ПЦР1 и ПЦР2 вносили секвенирующие зонды 30 Seq 52 Seq и 199/200seq и проводили денатурацию в течение 2 мин при 90°С.
3. Проводили пиросеквенирование денатурированных ПНР-продуктов на PSQ 96 МА.
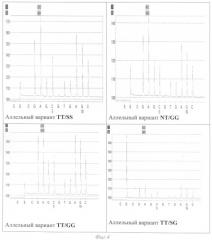
4. Генетические профили полиморфизма гена PRKAG3 в виде результирующих пирограмм представлены на фиг.4 и 5.
Таким образом, предложенный способ позволил осуществить одновременную диагностику аллелей T30N, S52G, I199V и R200Q гена PRKAG3 и определить соответствующие генотипы животных. При этом обеспечивалось сокращение затрат времени на выполнение генотипирования. Если при использовании ПЦР-ПДРФ-анализа требуется 12 часов, то пост-ПЦР-анализ-96 проб в предлагаемом нами способе диагностики длится 14 мин, пробоподготовка занимает 20 мин.
Изобретение применимо в молекулярной генетике животных для диагностики аллелей T30N, S52G, I199V и R200Q и определения генотипов гена PRKAG3 с целью последующего использования результатов в популяционной генетике, разведении и селекции свиней.
Способ диагностики аллелей T30N, S52G, I199V и R200Q гена PRKAG3, включающий амплификацию двух фрагментов гена-фрагмента 1 гена PRKAG3 длиной 270 п.о., включающего часть экзона 1 и содержащего специфические мутации в позиции 30 и 52 аминокислотной последовательности, и фрагмента 2 гена PRKAG3 длиной 118 п.о., включающего часть экзона 3 и специфические точковые мутации в позиции 199 и 200 аминокислотной последовательности, с использованием биотинилированных реверсивных праймеров
270F (позиции 8242 до 8266)AGCTTCCTAGAGCAAGGAGAGAGCC
270R bio (позиции 8321 до 8342) ACTGGCTGCATGATGTTATGTGC
264F (позиция 8196 до 8218)ATTCACCCTCAACTGTTCTCTCC
264R bio (позиция 8376 до 8396) TCACCCACGAAGCTCTGCTTC
отличающийся тем, что последующее генотипирование продуктов реакции проводят посредством пиросеквенирования с использованием генетических зондов:
30 Seq (позиция 69 до 85)AAGCCATGGGGACCAGG
52 Seq (позиция 135 до 150)GCCTCCGGGCCCGAGG
199/200 Seq (позиция 181 до 1843)TGGTGGCCAACGG
зондов 30 Seq и 52 Seq в мультиплексной системе генотипирования для фрагмента 1 и зонда 199/200 Seq в симплексной системе анализа для фрагмента 2.