Виртуальные наборы фрагментов нуклеотидных последовательностей
Иллюстрации
Показать всеИзобретение относится к олигонуклеотидным зондам и их композициям, которые могут использоваться для анализа репрезентативной выборки генома. Предлагается алгоритм, использующий преобразование генома и вспомогательную структуру данных для быстрого и точного подсчета количества конкретного нуклеотидного мотива в геноме. Подобный алгоритм может быть использован для идентификации олигонуклеотидных зондов по настоящему изобретению. Также предоставляются системы и способы для анализа различий в числе копий нуклеотидных последовательностей и сравнения уровня метилирования между двумя геномами. Изобретение предлагает упрощенный способ анализа генома за счет виртуального представления, с минимальным риском перекрестной гибридизации и возможностью избежать завышенного или заниженного представления некоторых последовательностей при анализе генома. 8 н. и 63 з.п. ф-лы, 12 ил., 2 табл.
Реферат
Область техники, к которой относится изобретение
Настоящее изобретение в целом относится к молекулярной биологии. Более конкретно, настоящее изобретение относится к материалам и способам получения нуклеотидных последовательностей, которые являются характерными для ДНК данного источника (например, генома).
Уровень техники
Благодаря глобальным способам анализа генома ученым удалось понять причины и взаимосвязи в патофизиологии рака и других заболеваний или состояний с генетическим компонентом. Такие способы включают кариотипирование, определение плоидности, сравнительную геномную гибридизацию (CGH), анализ репрезентативных различий (RDA) (см., например, патент США №5436142) и анализ геномных репрезентативных выборок (WO 99/23256, опубликованную 14 мая 1999). Обычно такие способы включают либо использование зондов для подробного исследования экспрессии конкретных генов, либо изучение изменений в самом геноме.
Используя олигонуклеотидные матрицы, эти способы могут использоваться для получения высокоэффективного изображения генетических изменений в клетках. Однако эти способы требуют данных о последовательностях конкретных зондов. В частности, эта необходимость ограничивает применение кДНК матрицы, поскольку такие матрицы детально исследуют только ограниченный набор генов. Их применение также ограничено в случае скрининга всего генома, поскольку большинство олигонуклеотидов, разработанных для матрицы, могут быть не представлены в исследуемой популяции, что приводит к недостаточному или неэффективному анализу.
Сущность изобретения
Настоящее изобретение относится к композиции и способам, которые могут использоваться для исследования популяций молекул нуклеиновых кислот. Такие композиции и способы могут использоваться для анализа сложных геномов (например, геномов млекопитающих), необязательно, совместно с технологией микроматриц. В настоящем изобретении охарактеризовано множество по меньшей мере 100 молекул нуклеиновых кислот (А), где (а) каждая молекула нуклеиновой кислоты специфически гибридизуется с последовательностью генома, состоящей по меньшей мере из Z пар оснований; и (b) по меньшей мере Р% указанного количества молекул нуклеиновых кислот: (i) имеют длину по меньшей мере К нуклеотидов; (ii) специфически гибридизуются по меньшей мере с одной молекулой нуклеиновой кислоты, присутствующей или предсказанной, что она присутствует, в репрезентативной выборке, полученной из указанного генома, где указанная репрезентативная выборка имеет не более R% сложность указанного генома; и (iii) не более чем Х точных совпадений L1 нуклеотидов с указанным геномом (или указанной репрезентативной выборкой) и не менее Y точных совпадений L1 нуклеотидов с указанным геномом (или указанной репрезентативной выборкой); и (В), где (а) Z≥1×108; (b) 300≥K≥30; (c) 70≥R≥0,001; (d) P≥90-R; (e) целое число, ближайшее к (log4(Z)+2)≥L1≥целого числа, ближайшего к log4(Z); (f) Х представляет собой целое число, ближайшее к D1×(K-L1+1); (g) Y представляет собой целое число, ближайшее к D2×(K-L1+1); (h) 1,5≥D1≥1; (i) 1≥D2≥0,5.
В некоторых вариантах осуществления (1) множество молекул нуклеиновых кислот содержит по меньшей мере 500; 1000; 2500; 5000; 10000; 25000; 50000; 85000; 190000; 350000; или 550000 молекул нуклеиновых кислот; (2) Z равно по меньшей мере 3×108, 1×109, 1×1010 или 1×1011; (3) R равно 0,001, 1, 2, 4, 10, 15, 20, 30, 40, 50 или 70; (4) Р не зависит от R и равно по меньшей мере 70, 80, 90, 95, 97 или 99; (5) D1 равно 1; (6) L1 равно 15, 16, 17, 18, 19, 20, 21, 22, 23 или 24; (7) Р равно 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100; и/или (8) К равно 40, 50, 60, 70, 80, 90, 100, 110, 120, 140, 160, 180, 200 или 250. В некоторых вариантах осуществления идентичность молекулы нуклеиновой кислоты, которая специфически гибридизуется с другой молекулой нуклеиновой кислоты, составляет по меньшей мере 90% последовательности по сравнению с последовательностью такой же длины другой молекулы нуклеиновой кислоты. В дополнительных вариантах осуществления последовательность идентична по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или по меньшей мере на 100%.
В некоторых вариантах осуществления каждая из указанного Р% указанного множества молекул нуклеиновых кислот дополнительно имеет не более А точных совпадений L2 и не менее В точных совпадений L2 нуклеотидов с указанным геномом, где (а) L1>L2≥целое число ближайшее к log4(Z)-3; (b) А представляет собой целое число, ближайшее к D3×((K-L2+1)×(Z/4L 2)); (c) В представляет собой целое число, ближайшее к D4×((K-L2+1)×(Z/4L 2)); (d) 4≥D3≥1; (е) 1≥D4≥0,5.
Репрезентативная выборка ДНК-популяции может быть получена расщеплением по специфичным последовательностям, например, осуществляемым рестрикционной эндонуклеазой. Репрезентативная выборка также может быть получена из другой репрезентативной выборки. То есть полученная репрезентативная выборка является сложной репрезентативной выборкой.
Молекулы нуклеиновых кислот по настоящему изобретению могут быть идентифицированы способом, предусматривающим: (а) расщепление указанного генома in silico рестрикционным ферментом с получением множества предсказанных молекул нуклеиновых кислот; (b) получение виртуальной репрезентативной выборки указанного генома путем идентификации предсказанных молекул нуклеиновых кислот, длина каждой из которых составляет 200-1200 пар оснований включительно, где сложность виртуальной репрезентативной выборки указанного генома составляет 0,001%-70% включительно; (с) осуществление отбора олигонуклеотида с длиной 30-300 нуклеотидов включительно, и по меньшей мере с 90% идентичностью в отношении последовательности предсказанной молекулы нуклеиновой кислоты (b); (d) расчет сложности указанной виртуальной репрезентативной выборки относительно указанного генома; (е) идентификацию всех участков нуклеотидов L1, встречающихся в указанном олигонуклеотиде; и (f) подтверждение того, что частота встречаемости каждого из указанных участков в указанном геноме удовлетворяет различным заранее заданным требованиям.
Молекулы нуклеиновых кислот по настоящему изобретению могут использоваться в качестве зондов для анализа образца ДНК. Эти зонды могут быть иммобилизованы на поверхности твердой фазы, в том числе на полутвердую поверхность. Твердая фаза включает в себя, без ограничений, нейлоновые мембраны, нитроцеллюлозные мембраны, стеклянные пластинки и микросферы (например, парамагнитные микробусины). В некоторых вариантах осуществления положение молекул нуклеиновых кислот на указанной твердой фазе является известным, например, как такое, которое используют в формате микроматрицы. В настоящем изобретении также описан способ анализа образца нуклеиновой кислоты (например, геномной репрезентативной выборки), где указанный способ предусматривает (а) гибридизацию образца с нуклеотидными зондами по настоящему изобретению; и (b) определение с какой из указанного множества молекул нуклеиновых кислот гибридизуется указанный образец.
В настоящем изобретении также описан способ анализа различия числа копий геномной последовательности двух геномов, где указанный способ предусматривает: (а) получение двух меченых репрезентативных выборок для визуализации, где каждый набор фрагментов получен из соответствующего генома с помощью по меньшей мере одного и того же рестрикционного фермента; (b) приведение в контакт этих двух репрезентативных выборок с нуклеотидными зондами по настоящему изобретению для того, чтобы обеспечить возможность гибридизации между репрезентативными выборками и зондами; (с) анализ уровня гибридизации двух репрезентативных выборок с набором зондов, где разница указанных уровней и числа зондов из набора указывает на различие числа копий двух геномов относительно геномной последовательности, на которую нацелен указанный элемент. В некоторых вариантах осуществления репрезентативные выборки отличаются тем, что являются мечеными; и/или тем, что приведение в контакт двух репрезентативных выборок осуществляют одновременно.
Настоящее изобретение дополнительно относится к способу сравнения уровня метилирования геномной последовательности двух геномов, где указанный способ предусматривает получение двух меченых репрезентативных выборок для визуализации фрагментов соответствующих геномов, где каждую репрезентативную выборку получают способом, чувствительным к метилированию. Например, первую репрезентативную выборку первого генома получают с использованием первого рестрикционного фермента, а вторую репрезентативную выборку второго генома получают с использованием второго рестрикционного фермента, где указанный первый и второй рестрикционные ферменты «узнают» один и тот же сайт рестрикции, но один является чувствительным к метилированию, а другой нет. Последовательности, содержащие метил-С, также могут быть химически расщеплены после получения репрезентативной выборки при помощи рестрикционного фермента, нечувствительного к метилированию, таким образом, что репрезентативная выборка, полученная из метилированного генома, будет отличаться от репрезентативной выборки, полученной из неметилированного генома. Затем две репрезентативные выборки приводят в контакт с зондами по настоящему изобретению для того, чтобы обеспечить возможность гибридизации репрезентативной выборки и зондов. Затем проводят анализ гибридизации двух репрезентативных выборок с зондами, где различие уровня гибридизации между репрезентативными выборками относительно конкретного зонда указывает на различие уровня метилирования между двумя геномами относительно последовательности генома, на которую нацелен указанный зонд.
Аналогичные способы также могут использоваться для анализа полиморфизма сложного генома, как показано ниже.
Некоторые варианты осуществления по настоящему изобретению относятся к алгоритму для точной и эффективной визуализации и подсчета числа «слов» в геноме. Такой алгоритм, иногда в настоящем описании называемый механизмом поиска или mer-механизмом, использует преобразование генома (например, преобразование Барроуза-Уиллера (Burrows-Wheeler)) и вспомогательную структуру данных для подсчета количества конкретных «слов» в геноме. «Слово» означает нуклеотидную последовательность определенной длины.
В общем случае, механизм производит поиск конкретного «слова» путем поиска последней «буквы слова». Затем он продолжает поиск «буквы», непосредственно предшествующей последней «букве». Если первая непосредственно предшествующая «буква» найдена, то затем производит поиск второй «буквы», непосредственно предшествующей последней «букве слова», и так далее до тех пор, пока не будет найдено все «слово». Если предшествующие «буквы» не были найдены, то делается вывод о том, что «слово» в геноме отсутствует. Если первая «буква слова» найдена, то частота ее встречаемости равна частоте встречаемости этого конкретного «слова».
Преимущество этого конкретного алгоритма состоит в его использовании для реализации нескольких конкретных применений, включая изучение генома, что обсуждается ниже.
Другие отличительные признаки и преимущества настоящего изобретения будут очевидными из нижеприведенных чертежей, подробного описания и формулы изобретения.
Краткое описание чертежей
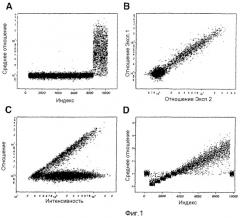
На фиг.1А-1D показаны предсказанные информационные данные и точность измерений с помощью матрицы c использованием микроматриц, содержащих 10000 олигонуклеотидов. На фиг.1А показаны результаты, где гибридизованные образцы являются репрезентативной выборкой, полученной с использованием BglII, и репрезентативная выборка, полученная с использованием BglII, повторно рестриктированных по сайту расщепления HindIII. Ось Y (Среднее отношение) представляет собой среднее измеренное отношение двух гибридизаций, репрезентативной выборки, подвергшихся повторной рестрикции, и нормальной репрезентативной выборки, приведенное на графике в логарифмическом масштабе. Ось Х (Индекс) представляет собой условный индекс, построенный таким образом, что зонды, полученные из фрагментов, определенных как имеющие фрагменты с внутренним сайтом HindIII, находятся с правой стороны. На фиг.1В показана воспроизводимость продублированных экспериментов для получения среднего отношения на фиг.1А. Ось Y (отношение Эксп.1) представляет собой измеренное отношение эксперимента 1 и ось Х (отношение Эксп.2) представляет собой измеренное отношение эксперимента 2. Обе оси изображены на графике в логарифмическом масштабе. На фиг.1С изображено нормализованное отношение по оси Y как функция интенсивности образца, который не является повторно рестриктированным, по оси Х. Как отношение, так и интенсивность изображены на графике в логарифмическом масштабе. На фиг.1D представлены данные, полученные путем моделирования. Ось Х (Индекс) представляет собой условный индекс. Зонды, в группах 600, обнаруживают увеличение числа копий, слева направо. Зонды, примыкающие со стороны 600, обнаруживают нормальное число копий. Ось Y (Среднее отношение) представляет собой среднее отношение, приведенное на графике в логарифмическом масштабе.
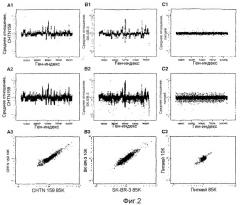
На фиг.2А1-2А3, 2В1-2В3 и 2С1-2С3 показаны геномные профили для образца первичного рака молочной железы (CHTN159), с анеуплоидными ядрами, в сравнении с диплоидными ядрами от того же пациента (фиг.2А1-2А3), линии клеток рака молочной железы, в сравнении с эталоном нормальной мужской особи (фиг. 2В1-2В3), и нормальной мужской особи с эталоном нормальной мужской особи (фиг. 2С1-2С3), с использованием печатной матрицы 10К (фиг.2А1, 2В1 и 2С1) и с использованием фотопечатной матрицы 85К (фиг.2А2, 2В2 и 2С2). В каждом случае (фиг.2А1, 2В1 и 2С1 и фиг.2А2, 2В2 и 2С2) ось Y представляет собой среднее отношение, а ось Х (Ген-индекс) представляет собой индекс, который графически представляет расположение зондов в том геномном порядке, в котором они представлены в хромосомах, и дает возможность визуализировать весь геном от хромосомы 1 до Y. На фиг.2А3, 2В3 и 2С3 показано соответствие отношений, измеренных для зондов-«братьев», представленных в микроматрицах 10К и 85К. Ось Y представляет собой измеренное отношение для микроматрицы 10К, а ось Х представляет собой измеренное отношение для микроматрицы 85К.
На фиг.3А-3D показано несколько хромосом с изменяющимися флуктуациями числа копий в случае анализа клеточной линии опухоли SK-BR-3 в качестве сравнения с нормальным эталоном. Ось Y (Среднее отношение) представляет среднее отношение двух гибридизаций в логарифмическом масштабе. Ось Х (Ген-индекс) представляет собой индекс геномных координат. На фиг.3А представлены флуктуации числа копий, идентифицированных для хромосомы 5, на фиг.3В - для хромосомы 8, на фиг.3С - для хромосомы 17 и на фиг.3D - для хромосомы Х.
На фиг.4А-4D показана средняя сегментация, вычисленная в случае анализа SK-BR-3 в сравнении с нормальным эталоном (фиг.4А и фиг.4В) и CHNT159 (фиг.4С и фиг.4D). На фиг.4А-4D ось Y соответствует значению среднего сегмента для каждого зонда в логарифмическом масштабе. На фиг.4А и фиг.4С каждая из осей Х (индекс среднего сегмента) приведена с возрастающим значением своей назначенной средней сегмента. На фиг.4В и фиг.4D ось Х (Ген-индекс) представляет собой геномный индекс, который, как описано выше, представляет весь геном последовательно. Изображенные наверху данные среднего сегмента представляют собой сетку числа копий, экстраполированную из данных матрицы с использованием формул в данном описании (горизонтальные линии). Вычисленное число копий для каждой горизонтальной линии находится справа от сетки.
На фиг.5А-5D по оси Y (Среднее отношение SK-BR-3) изображено среднее отношение двух гибридизаций SK-BR-3 в сравнении с нормальным эталоном в логарифмическом масштабе. Ось Х (Ген-индекс) представляет собой геномный индекс. На фиг.5А показан участок Х хромосомы с участком потери. Изображенное поверх измеренного отношения матрицы представляет собой вычисленное значение сегментации. На фиг.5В показан участок хромосомы 8 (c-myc, расположенный с правой стороны от центра графика) в случае результатов для SK-BR-3 в сравнении с нормальным эталоном. Изображенные поверх данные представляют собой значения сегментации для SK-BR-3 в сравнении с нормальным эталоном с диагональной штриховкой и значения сегментации для первичной опухоли CHTN159 с вертикальной штриховкой. На фиг.5С показано нарушение в хромосоме 5, показывающее разрешающую способность 85К в сравнении с матрицей 10К. Результаты получены в случае SK-BR-3 в сравнении с нормальным эталоном. Незаштрихованные кружки соответствуют результатам для печатной матрицы 10К, а заштрихованные кружки соответствуют результатам для фотопечатной матрицы 85К. Горизонтальные линии соответствуют оценке числа копий, исходя из моделирования значений средних сегментов. На фиг.5D показано сравнение SK-BR-3 с нормальным эталоном, изображающим область гомозиготной делеции в хромосоме 19. Значение среднего сегмента изображено в виде белой линии, а сетка представляет собой оценки числа копий, как описано выше.
На фиг.6А-6D показаны результаты в случае нормы в сравнении с нормой, идентичные изображенным на фиг.2С2 за исключением того, что синглетные зонды отфильтрованы, как описано в тексте. На фиг.6В показано последовательное сравнение экспериментов для небольшого участка хромосомы 4. Ось Y представляет собой среднее отношение в логарифмическом масштабе. Ось Х представляет собой Ген-индекс. Заштрихованные (85К) и незаштрихованные (10К) кружки получены в случае сравнения SK-BR-3 с нормой. Пустые треугольники соответствует сравнению пигмея с нормальным эталоном. На фиг.6С показано нарушение, обнаруженное в нормальной популяции в хромосоме 6. Заштрихованные кружки нанесены исходя из среднего отношения в случае анализа пигмея по отношению к нормальному эталону. Линия с вертикальной штриховкой соответствует значению среднего сегмента для пигмея в сравнении с нормальным эталоном. Линия с диагональной штриховкой соответствует значению среднего сегмента для SK-BR-3 в сравнении с нормальным эталоном. Линия с крестообразной штриховкой соответствует значению сегмента в сравнении с первичной опухолью (анеуплоидной по отношению к диплоидной CHNT159). На фиг.6D показан участок хромосомы 2. Данные, показанные кружками, получены из сравнения SK-BR-3 с нормальным эталоном. Линия среднего сегмента для этого сравнения показана с вертикальной штриховкой. Линия среднего сегмента для сравнения пигмея с нормальным эталоном показана с диагональной штриховкой, а в случае первичной опухоли CHNT159 - с крестообразной штриховкой. Для фиг.6С и 6D вычисленное число копий для горизонтальных линий приведено справа от диаграммы.
На фиг.7 показана блок-схема иллюстративной системы согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.8 показана блок-схема последовательности операций иллюстративного предварительного этапа обработки для выполнения точных подсчетов «слов» согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.9А и 9В показана блок-схема последовательности операций иллюстративного алгоритма подсчета «слов» согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.10А и 10В показан иллюстративный пример алгоритма подсчета «слов» по фиг.9А и 9В согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.11 показан иллюстративный массив суффиксов, имеющий положения координат, соответствующие координатам генома согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.12А показано графическое представление переменных и структур данных, используемых в алгоритме, согласно некоторым вариантам осуществления настоящего изобретения.
На фиг.12В показано представление в виде псевдокода алгоритма согласно некоторым вариантам осуществления настоящего изобретения.
Описание изобретения
Настоящее изобретение относится к олигонуклеотидным зондам для анализа репрезентативной выборки ДНК-популяции (например, генома, хромосомы или смеси ДНК). Олигонуклеотидные зонды могут использоваться в растворе или могут быть иммобилизованы на твердой (включая полутвердую) поверхности, такой как матрица или микробусины (например, Lechner et al., Curr. Opin. Chem. Biol., 6:31-38 (2001); Kwok, Annu. Rev. Genomics Human Genet., 2:235-58 (2001); Aebersold et al., Nature, 422:198-207 (2003); и в патентах США 6355431 и 6429027). Репрезентативная выборка является репродуцируемым образцом ДНК-популяции, где результирующая ДНК обычно имеет новый формат или уменьшенную сложность или и то и другое (Lisitsyn et al., Science 258:946-51 (1993); Lucito et al., Proc. Natl. Acad. Sci. USA, 92:151-5(1998)). Например, репрезентативная выборка генома может состоять из последовательностей ДНК, которые получены только из небольшой части генома и, в основном, не имеют повторяющихся последовательностей. Анализ геномной репрезентативной выборки может выявить изменения в геноме, включая мутации, такие как делеции, амплификации, хромосомные перестройки и полиморфизмы. При проведении анализа в клинических целях с помощью него можно обеспечить понимание молекулярной основы заболевания, а также он может быть полезен для диагностики и лечения такого заболевания.
Композиции олигонуклеотидов по настоящему изобретению могут использоваться для гибридизации с репрезентативной выборкой ДНК-источника, где данные гибридизации обрабатывают для получения генетических профилей ДНК-источника (например, генетических нарушений и полиморфизмов, связанных с заболеваниями). Может быть предпочтительным, чтобы репрезентативные выборки (или в настоящем описании «тестируемая репрезентативная выборка») и по меньшей мере некоторую фракцию олигонуклеотидных зондов в композициях получают из одного и того же вида. Может быть использована ДНК любого вида, включая виды млекопитающих (например, ДНК свиньи, мыши, крысы, примата (например, человека), собаки и кошки), виды рыб, виды рептилий, виды растений и виды микроорганизмов.
I. ОЛИГОНУКЛЕОТИДНЫЕ ЗОНДЫ
Олигонуклеотидные зонды по настоящему изобретению, предпочтительно, разработаны с помощью виртуальной репрезентативной выборки ДНК-источника, такого как геномная ДНК указанного индивида. Репрезентативная выборка генома с получением фрагментов обычно, но не всегда, приводит в результате к упрощению его сложности. Сложность репрезентативной выборки соответствует сложности фракции генома, которая в нем представлена. Один из способов вычисления сложности представляет собой деление количества нуклеотидов во фрагменте на количество нуклеотидов в геноме. Сложность геномной репрезентативной выборки может находиться в пределах от менее 1% до 95% всего генома. Там, где используется ДНК с относительно простым геномом, репрезентативная выборка может иметь 100% сложность общего генома, например, репрезентативная выборка может быть получена путем рестрикционного расщепления всей ДНК без амплификации. Репрезентативная выборка, связанная с настоящим изобретением, обычно имеет сложность между 0,001% и 70%. Уменьшение сложности обеспечивает требуемые кинетические характеристики гибридизации.
«Реальное» рестриктирование ДНК с получением репрезентативной выборки включает в себя лабораторные процедуры («влажные работы»), с помощью которых получают репрезентативную выборку ДНК. «Виртуальное» рестриктирование с получением репрезентативной выборки, с другой стороны, имеет преимущество в том, что секвенированы полные геномы, например человеческий геном. С помощью вычислительного анализа доступных геномных последовательностей можно легко разработать большое количество олигонуклеотидных зондов, которые гибридизуются с картированными областями генома и имеют минимальную степень перекрывания последовательностей с остальным геномом.
В качестве примера для разработки набора олигонуклеотидных зондов для генетического анализа человека можно in silicо (т.е., виртуально) провести расщепление человеческого генома путем определения местоположения всех сайтов расщепления выбранной рестрикционной эндонуклеазы в секвенированном геноме. Затем можно провести анализ полученных фрагментов для идентификации тех, которые находятся в требуемой области (например, 200-1200 п.о., 100-400 п.о. и 400-600 п.о.), которые могут быть амплифицированы при помощи, например, ПЦР. Такие фрагменты определены в настоящем описании как «предсказанные, что они существуют» в репрезентативной выборке. Рестрикционная эндонуклеаза может быть выбрана исходя из сложности требуемой репрезентативной выборки. Например, рестрикционные эндонуклеазы, которые разрезают редко, например, такие как эндонуклеазы, которые «узнают» заданные последовательности из 6 п.о. или 8 п.о., будут продуцировать репрезентативную выборку более низкой сложности, в то время как рестрикционные эндонуклеазы, которые разрезают часто, например, такие как эндонуклеазы, которые «узнают» заданные последовательности из 4 п.о., будут продуцировать репрезентативные выборки более высокой сложности. Кроме того, факторы, такие как содержание G/C анализируемого генома, будут оказывать воздействие на частоту расщепления конкретными рестрикционными эндонуклеазами и, следовательно, влиять на выбор рестрикционных эндонуклеаз. Обычно используются надежные рестрикционные эндонуклеазы, которые не демонстрируют сильной активности. В качестве альтернативы также может применяться расщепление исходя из уровня метилирования заданного сайта, например, путем использования рестрикционного фермента, чувствительного к метилированию, или другого фермента, такого как McrBC, который «узнает» в ДНК метилированные цитозины.
Последовательности всех расщепленных фрагментов требуемой области (например, 200-1200 п.о., 100-400 п.о. и 400-600 п.о.) анализируют при помощи компьютера, где области некоторых из таких фрагментов, которые составляют по меньшей мере около 30 п.о. в длину и имеют минимальную гомологию с оставшимся геномом, могут быть выбраны как реперезентативные олигонуклеотидные зонды для генома человека. В примере 1 и секции IV ниже дополнительно проиллюстрированы способы идентификации олигонуклеотидов настоящего изобретения.
Олигонуклеотиды по настоящему изобретению могут быть длиной от примерно 30 нуклеотидов до примерно 1200 нуклеотидов. Точная длина выбранных олигонуклеотидов будет зависеть от предполагаемого использования, например, размера ДНК-источника, из которого получена репрезентативная выборка, и от того используются ли они как компоненты матрицы. Обычно олигонуклеотиды имеют длину более по меньшей мере 35 нуклеотидов, например по меньшей мере 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95 или 100 нуклеотидов, но они также могут быть короче, например, длиной 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 и 30 нуклеотидов. Обычно олигонуклеотиды имеют длину не более 600 нуклеотидов, например, не более 550, 500, 450, 400, 350, 300, 250, 200 или 150 нуклеотидов. Как очевидно специалистам в данной области техники, длина олигонуклеотидов будет зависеть от характеристик анализируемого генома, например, сложности и количества повторяющихся последовательностей.
II. МАТРИЦЫ ОЛИГОНУКЛЕОТИДОВ
Олигонуклеотидные зонды по настоящему изобретению могут использоваться в формате матрицы. Матрица содержит твердую подложку с зондами нуклеиновых кислот, прикрепленными к ней в определенных координатах или адресах. Каждый адрес содержит либо большое число копий единичного ДНК-зонда, либо смесь различных ДНК-зондов. Матрица нуклеиновых кислот, также называемая «микроматрицей» или «чипом», в общем случае описана, например, в патенте США 6361947 и ссылках, цитируемых в нем. Авторы назвали генетический анализ, использующий новые матрицы, «анализом с помощью микроматрицы репрезентативных олигонуклеотидов» (ROMA) или анализ, где расщепление зависит от метилирования в заданном сайте, «анализ с помощью микроматрицы олигонуклеотидов с визуализацией метилирования» («MOMA»).
Для производства микроматрицы по настоящему изобретению предварительно синтезированные олигонуклеотиды прикрепляют к твердой подложке, которая может быть изготовлена из стекла, пластика (например, полипропилена или нейлона), полиакриламида, нитроцеллюлозы или других материалов, и может быть пористой или непористой. В одном из способов прикрепление нуклеиновых кислот к поверхности происходит с помощью печати на стеклянные плашки, как в общем случае описано у Schena et al., Science 270:467-70 (1995); DeRisi et al., Nature Gen. 14:457-60 (1996); Shalon et al., Genome Res. 6:639-45 (1996); и Schena et al., Proc. Natl. Acad. Sci. USA, 93:10539-1286 (1995). Для матриц с низкой плотностью также могут использоваться дот-блоты на нейлоновой гибридизационной мембране. См., например, Sambrook et al., Molecular Cloning - A Laboratory Manual (2nd Ed.), vol.1-3, Cold Spring Harbor Laboratory, Cold Spring Harbor, New York, 1989.
В другом способе для изготовления микроматриц используют фотолитографические (или «фотопечатные») технологии для синтеза олигонуклеотидов непосредственно на подложке матрицы, т.е. in sutu. См. Fodor et al., Science 251:767-73 (1991); Pease et al., Proc. Natl. Acad. Sci. USA, 91:5022-6 (1994); Lipschutz et al., Nat. Genet., 21 (1 Suppl):20-46 (1999); Nuwaysir et al., Genome Res., 12(11):1749-55 (2002); Albert et al., Nucl. Acids Res., 31(7):e35 (2003); и патенты США 5578832, 5556752 и 5510270. Также могут использоваться другие способы быстрого синтеза и осаждения определенных олигонуклеотидов. См., например, Blanchard et al., Biosensors & Bioelectronics, 11:687-90 (1996); и Maskos and Southern, Nucl. Acids Res., 20:1679-1684.
Матрицы по настоящему изобретению обычно содержат по меньшей мере 100 (например, по меньшей мере 500, 1000, 5000 или 10000) олигонуклеотидных зондов, и могут содержать намного большее количество зондов, например, 25000, 50000, 75000, 85000, 100000, 200000, 250000, 500000 или 700000 зондов. Матрицы по настоящему изобретению обычно содержат не более 700000 зондов. Однако они могут содержать больше, например, более 800000, 900000 или 1000000 зондов. В некоторых вариантах осуществления матрицы представляют собой матрицы с высокой плотностью, с плотностями, превышающими примерно 60 различных зондов на 1 см2. Олигонуклеотиды в матрицах могут быть однонитевыми или двухнитевыми. Для простоты производства и использования матриц олигонуклеотидные зонды по настоящему изобретению могут быть модифицированы, например, путем введения в зонды пептидильной структуры и нуклеотид-аналогов.
III. ТЕСТИРУЕМЫЕ РЕПРЕЗЕНТАТИВНЫЕ ВЫБОРКИ
Олигонуклеотидные матрицы по настоящему изобретению могут использоваться для изучения любого образца нуклеиновой кислоты по выбору. Например, образец может быть библиотекой ДНК, библиотекой геномной ДНК или препаратом РНК. В других вариантах осуществления матрицы по настоящему изобретению могут использоваться для изучения образцов ДНК, которые являются репрезентативными выборками (или «тестируемыми репрезентативными выборками») популяции сложной ДНК, такой как геном более высокоразвитого организма.
Репрезентативные выборки и способы их получения описаны, например, у Lisitsyn et al., Proc. Natl. Acad. Sci. USA, 92:151(1995); Lucito et al., Proc. Natl. Acad. Sci. USA, 95:4487-4492(1998) и WO 99/23256. Один из подходов в создании репрезентативной выборки включает воспроизводимое расщепление ДНК-популяции на фрагменты. Воспроизводимое расщепление обычно выполняется расщеплением одной или несколькими рестрикционными эндонуклеазами (например, DpnI или BglII) или ферментом(ферментами), который выполняет расщепление в конкретных метилированных сайтах (например, McrBC), но может быть использован любой способ, который воспроизводимо расщепляет ДНК. Полученные фрагменты ДНК связывают с адаптером олигонуклеотидов. Затем эти фрагменты амплифицируют, например, с помощью полимеразной цепной реакции («ПЦР») или лигазной цепной реакции, используя праймеры, комплементарные адаптерам. Амплифицированные фрагменты представляют собой подгруппу начальной ДНК-популяции. Благодаря этапу амплификации репрезентативные выборки могут быть получены из очень небольших количеств начального материала (например, 5 нг ДНК). Репрезентативный дифференциальный анализ («RDA»), как описано у Lisitsyn et al., Science 258:946-51(1993) и патентах США 5436142 и 5501946, может быть использован для удаления любых известных нежелательных последовательностей из репрезентативной выборки, включая повторяющиеся последовательности.
Начальная популяция ДНК может представлять собой большие молекулы ДНК, такие как геном организма или его часть (например, хромосома или ее участок). Авторы называют репрезентативные выборки таких ДНК-популяций, как хромосомные или геномные репрезентативные выборки соответственно. Начальные ДНК-популяции могут быть получены, например, из образцов больных тканей, таких как образцы биопсии опухоли, образцов нормальных тканей, клеточных линий опухолей, нормальных клеточных линий, клеток, сохраненных в качестве фиксированных образцов, образцов аутопсии, образцов судебной экспертизы, образцов палео-ДНК, микропрепарированных образцов тканей, изолированных ядер, изолированных хромосом или участков хромосом и фракционированных клеток или образцов тканей. Также могут быть получены репрезентативные выборки из репрезентативной выборки (или «сложные репрезентативные выборки»). Сложные репрезентативные выборки могут использоваться для скрининга полиморфизма. См., например, WO 99/23256.
Для сравнительного анализа репрезентативных выборок двух источников ДНК, такого как сравнение геномных фрагментов из нормальной клетки с геномными фрагментами из раковой клетки или клетки с другим заболеванием, предпочтительным может быть параллельное получение двух репрезентативных выборок, например, изоляцией начальной ДНК из двух клеток в одно и то же время и одним и тем же способом, получением репрезентативных выборок из одинакового количества начальной ДНК и амплификацией фрагментов ДНК в одно и то же время в одинаковых условиях в одном и том же термальном циклере. Также предпочтительным может быть то, что нормальную клетку и больную клетку берут от одного и того же индивида, хотя возможно получение «нормальной» геномной ДНК путем комбинации, например, ДНК обоих родителей данного индивида.
Сложность репрезентативных выборок обычно бывает ниже сложности начальной ДНК-популяции, поскольку существуют последовательности, присутствующие в начальной популяции, которые не присутствуют в репрезентативной выборке. Сложность репрезентативной выборки связана с частотой разрезания рестрикционными эндонуклеазами в конкретной начальной популяции. Разрезания с большей частотой приводят к более сложной репрезентативной выборке. Поскольку фрагменты между 200-1200 парами оснований предпочтительно амплифицируют с помощью ПЦР в обычных условиях, могут быть получены наборы фрагментов высокой сложности путем расщепления начальной ДНК таким образом, что большинство фрагментов будут длиной 200-1200 пар оснований. Напротив, репрезентативные выборки низкой сложности могут быть получены расщеплением молекулы ДНК таким образом, чтобы меньшее количество фрагментов будут длиной 200-1200 пар оснований. Например, расщепление геномной ДНК человека с помощью DpnII может привести к набору фрагментов, имеющему около 70% сложности всего человеческого генома. С другой стороны, расщепление путем разрезания с меньшей частотой, такое как расщепление BamHI или BglII, может привести к репрезентативной выборке, имеющей только около 2% сложности человеческого генома. Репрезентативные выборки высокой сложности пригодны, например, для определения числа копий гена, картирования делеций, определения потери гетерозиготности, сравнительной геномной гибридизации и архивирования ДНК. Обычно репрезентативные выборки низкой сложности полезны для тех же целей, но дают лучшие кинетические характеристики гибридизации, чем репрезентативные выборки высокой сложности.
Сложность репрезентативной выборки может быть дополнительно уточнена путем использования более одного рестрикционного фермента для получения фрагментов перед лигированием с адаптерами, и/или путем использования одного или нескольких дополнительных рестрикционных ферментов для расщепления подгруппы фрагментов после лигирования с адаптерами, таким образом, укорачивая полученные нуклеотидные фрагменты из этих репрезентативных выборок фрагментов. Любой рестрикционный фермент, включая рестрикционные ферменты, чувствительные к метилированию, может