Способ расширенного скрининга предрасположенности к сердечно-сосудистым заболеваниям и биочип для осуществления этого способа
Иллюстрации
Показать всеИзобретение относится к области биотехнологии, а именно к способу обнаружения точечных нуклеотидных замен в генах человека, ответственных за предрасположенность и развитие сердечно-сосудистых заболеваний и биочипу, используемому в данном способе. Способ включает стадию выделения геномной ДНК из клинического образца, амплификацию участков генов АСЕ, АРОА4, АРОА5, АРОВ, EDN1, MEF2A, FGA, ADRB1, TNBS4, АРОЕ, FBN1, AGXT, MTHFR, CCR2, F5, F2, F7, АВСА1, ITGB3, AGTR2, B2R, DES, TLR4, NOS3, KDR, MMP9, THBS2, LPL, МРО, содержащих последовательности полиморфных локусов, перечисленные в SEQ ID 1-672 и получение одноцепочечного флюоресцентно меченого продукта методом ник-трансляции и рестрикции. Приготавливают биочип для скрининга предрасположенности к заболеваниям сердечно-сосудистой системы, содержащий набор иммобилизованных олигонуклеотидов SEQ ID 1-672. Осуществляют гибридизацию меченого амплифицированного продукта на указанном биочипе. Регистрируют результаты при длине волны 635 нм и 532 нм для красителей Cy5 и Cy3 соответственно. Интерпретируют результаты гибридизации путем сравнения интенсивности флюоресцентных сигналов, полученных при совершенной и несовершенной гибридизации. Предложенное изобретение позволяет получить комплексную оценку генетических предрасположенностей пациента к сердечно-сосудистым заболеваниям и оценить риски развития сердечно-сосудистых заболеваний. 2 н. и 4 з.п. ф-лы, 29 табл., 4 пр.
Реферат
Изобретение относится к молекулярной биологии, микробиологии и медицине, в частности, к способам выявления и идентификации известных полиморфизмов генов, ответственных за развитие сердечно-сосудистых заболеваний (ССЗ). Изобретение также относится к технологии изготовления биочипов, находящих применение в молекулярной биологии при секвенировании и картировании ДНК, детектировании мутаций и целого ряда медицинских приложений.
Сердечно-сосудистые заболевания в настоящее время остаются наиболее частой причиной смерти населения развитых стран, несмотря на достигнутые успехи в области диагностики, лечения данной патологии. Ведущее место в структуре смертности и инвалидности от заболеваний сердечно-сосудистой системы принадлежит ишемической болезни сердца. Острое нарушение коронарного кровообращения занимает ведущее место среди сердечно-сосудистых заболеваний, смертность от которых в Российской Федерации составляет 56% в структуре общей смертности (Лупанов В.П. Вторичная профилактика ишемической болезни сердца / Русский медицинский журнал. - 2005. - Т.13. - №.11. - С.747-750) [1]. Несмотря на проведенные обширные исследования по проблеме инфаркта миокарда и его последствий, все еще возникают затруднения в выделении категории больных с высоким риском развития сердечно-сосудистых заболеваний и их осложнений. Точная диагностика в сочетании с детальным анализом типа наследования того или иного заболевания имеет определяющее значение для формирования групп риска, то есть отбора пациентов, у которых вероятность ССЗ повышена. Своевременное обнаружение предрасположенности к данным заболеваниям и соответствующая терапия позволяют резко повысить качество жизни пациентов с сердечно-сосудистыми заболеваниями. В настоящее время известно несколько способов используемых для скрининга на сердечнососудистые заболевания.
Способ диагностики генетической предрасположенности к инфаркту миокарда у мужчин (RU №2182175, C12Q 1/68, 2002.05.10) [2], включает выявление генотипов геномного локуса, содержащего аллель гена. У исследуемых пациентов проводят генотипирование полиморфизма V64I гена CCR2 и при выявлении генотипа геномного локуса 3q21, содержащего аллель гена хемокинового рецептора CCR2 с заменой G на А в 190-й позиции кодирующей части, приводящей к замене V на I в 64-й позиции аминокислотной последовательности белка CCR2 641, диагностируют генетическую предрасположенность к инфаркту миокарда у мужчин.
Однако данный способ оценивает всего лишь один фактор из множества влияющих на риск развития сердечно-сосудистых заболеваний и поэтому не может дать полной информации о пациенте.
Известен способ определения предрасположенности к сердечно-сосудистым заболеваниям (RU №2304775, G01N 33/48, C12Q 1/68, 2007.08.20) [3], включающий выделение ДНК, проведение полимеразной цепной реакции для определения мутаций и/или полиморфизма генов, состоящей из первичной денатурации, цикла из денатурации, отжига и синтеза и заключительного синтеза, проведение гидролиза продуктов полимеразной цепной реакции эндонуклеазами рестрикции Msp I и Hinf I, и электрофорез в полиакриламидном геле, полимеразную цепную реакцию с использованием диагностического набора, при этом первичную денатурацию проводят в течение 7 мин при температуре 94°С, далее проводят цикл от 28 до 32 раз, включающий денатурацию при 94-96°С в течение от 1 мин до 1 мин 10 с, отжиг при температуре от 58 до 60°С в течение от 1 мин до 1 мин 10 с, синтез при температуре 72°С в течение от 1 мин до 1 мин 20 с, заключительный синтез в течение 5 мин при температуре 72°С, гидролиз продуктов полимеразной цепной реакции проводят смесью эндонуклеаз рестрикции, а электрофорез продуктов гидролиза проводят в 7,5% полиакриламидном геле до выхода маркера из геля, а затем определяют полиморфизм генов АСЕ, eNOS, MTHFR, GPIIIa.
Однако данный способ позволяет провести одновременный анализ лишь нескольких маркеров, что не дает возможности перекрыть спектр даже наиболее частых мутаций, оказывающих влияние на предрасположенность к сердечно-сосудистым заболеваниям. Кроме этого, известный способ требует до 3 суток на проведение анализа.
Известен способ генетической диагностики подверженности к сердечно-сосудистым заболеваниям (RU №2376372, C12N 15/00, 20.12.2009) [4]. Способ основан на определении генотипов по полиморфным вариантам А1166С гена AGTR1, А-240Т и A2350G гена АСЕ, С677Т гена MTHFR, Т174М гена AGT, C825T гена GNB3, VNTR4a/b и G894T гена NOS3, G1691A гена F5, PLA1/A2 гена ITGB3, G20210A гена F2 и оценке риска путем суммирования количества баллов, присвоенных каждому генотипу. При этом генотипам «низкого» риска сердечно-сосудистой патологии присваивается 0 баллов, к ним относятся генотипы 1166АА гена AGTR1, -240АА, 2350АА гена АСЕ, 677СС гена MTHFR, 174TT гена AGT, 825CC гена GNB3, VNTR4bb и 894GG гена NOS3, 1691GG гена F5, PLA1/A1 гена ITGB3, 20210GG гена F2. Генотипам «среднего» риска присваивается 0,5 баллов, к ним относятся генотипы 1166АС гена AGTR1, -240АТ и 2350AG гена АСЕ, 677СТ гена MTHFR, 174TM гена AGT, 825CT гена GNB3, VNTR4ab и 894GT гена NOS3. Генотипам «высокого» риска присваивается 1 балл, к ним относятся генотипы 1166СС гена AGTR1, -240ТТ и 2350GG гена АСЕ, 677ТТ гена MTHFR, 174MM гена AGT, 825TT гена GNB3, VNTR4aa и 894GT гена NOS3, 1691GA и 1691АА гена F5, PLA1/A2 и PLA2/A2 гена ITGB3, 20210GA и 20210АА гена F2. Риск сердечно-сосудистых болезней считается «низким» при сумме баллов от 0 до 3, средним - от 3,5 до 6, высоким - от 6,5 до 11 баллов.
Известен способ изучения полиморфной области гена PAR1, для оценки риска сердечно-сосудистых заболеваний (RU №2380422, C12Q 1/68, С12Р 19/34, C12N 15/12, 27.01.2010) [5], которая может содержать замену Т на С в положении 3090 и/или замену А на С в положении 3329 полинуклеотидной последовательности гена дикого типа (NM_001992), с применением пары специфических праймеров, а также способ определения в названной области, полученной из ДНК-содержащего биологического образца, наличия или отсутствия указанных замен. Предложены полные наборы компонентов, применение которых обеспечивает как амплификацию полиморфной области гена PAR1 по изобретению, так и в случае необходимости, проведение дальнейшего анализа ее на наличие генетических модификаций в положениях 3090 и/или 3329.
Ограничение информативности данного способа приводит к невозможности предсказать риски развития различных патологий сердечно-сосудистой системы, независимых от гена PAR1.
Все вышеописанные способы нецелесообразно применять для массового скрининга, т.к. они трудоемки и исследуют недостаточное количество полиморфных локусов, что особенно важно для оценки мультифакторных патологий, какими являются патологии сердечно-сосудистой системы.
Известны способы изготовления биочипов на основе гидрогелей, в которых технологический цикл состоит из этапов: (1) подготовки подложки, (2) формирования на ней матрицы ячеек геля, (3) нанесения на ячейки растворов биологических макромолекул в соответствии с заранее составленной схемой биочипа, (4) химической обработки ячеек с целью иммобилизации молекул-зондов, (5) отмывки и просушки полученных биочипов.
Известен способ приготовления биочипа (US №5574142, A61K 47/48, A61K 47/48, 1996.11.12) [6], согласно которому используют специальную тонкослойную (5 мкм) камеру с реакционным объемом, ограниченным с одной стороны подложкой будущего биочипа, а с другой - окном, прозрачным в УФ-области. Ячейки биочипа формируют одну за другой путем циклического выполнения следующих операций: (1) заполнения камеры композицией с соответствующим зондом, (2) полимеризации композиции в месте нахождения будущей ячейки под действием УФ-излучения, сфокусированным в квадратное пятно необходимого размера, (3) промывки камеры перед заполнением ее очередным раствором.
Известен «Способ изготовления микрочипа на основе олигонуклеотидов» (RU №2157385, C08F 220/56, G01N 33/50, C12Q 1/68, С07Н 21/00, 2000.10.10) [7], согласно которому изготовляют микрочип на основе олигонуклеотидов, иммобилизованных в органическом геле, приготовленном путем сополимеризации непредельных производных олигонуклеотидов с ненасыщенными мономерами, причем водно-солевые растворы, содержащие ненасыщенные мономеры, модифицированные непредельными фрагментами олигонуклеотиды и компонент каталитической системы, индуцирующей полимеризацию, растворимый только в воде, наносят на стеклянную подложку в виде микрокапель и проводят сополимеризацию мономеров погружением сформированной матрицы в не смешивающийся с водой органический растворитель, содержащий растворенный другой компонент каталитической системы.
Известно изобретение «Композиция (К) для иммобилизации биологических макромолекул в гидрогелях методом сополимеризации, способ приготовления композиции и биочип» (RU №2206575, C07K 17/08, С07Н 21/00, C08F 220/56, C12Q 1/68, 2003.06.20) [8], которое относится к композициям (К) для полимеризационной иммобилизации биологических макромолекул в гидрогелях при формировании биочипов К=аА+bB+cC+dD+eE, включающим А - мономер на основе производных акриловой и метакриловой кислот; В - водорастворимый сшивающий агент; С - модифицированную биологическую макромолекулу, содержащую ненасыщенную группу; D - водорастворимое соединение как компонент среды проведения сополимеризации; Е - воду, где а, b, с, d, e - процентное содержание (X) каждого компонента в композиции (Х=m/v100% для твердых веществ и X=v/v100% для жидких веществ), в которой общее содержание мономера и сшивающего агента лежит в интервале 3-40% (3(a+b)(40)), соотношение мономера и сшивающего агента находится в пределах 97: 3-60:40, а процентное содержание компонентов С, D и Е находится в пределах (с) от 0,0001% до 10%; (d) от 0% до 90%; (e) от 5% до 95%, способу их приготовления, к модифицированным биологически значимым соединениям ДНК и белкам, биочипу, в котором сформированный на подложке слой ила разделен пустыми промежутками на несколько ячеек, причем каждая из ячеек может содержать либо не содержать иммобилизованные макромолекулы, а макромолекулы, иммобилизованные в разных ячейках, могут различаться по своей природе и свойствам, и двум способам проведения полимеризационно-цепной реакции на биочипе.
Однако известные композиции для иммобилизации биологических макромолекул в гидрогелях при формировании биочипов, биочипы и способы их изготовления имеют ряд недостатков.
1. Использование только акриламида как гелеобразующего компонента и N,N'-метиленбисакриламида как сшивающего агента при изготовлении гелей сильно ограничивает спектр получаемых гидрогелей по структуре и пористости.
2. Модифицированные олигонуклеотиды, используемые для изготовления биочипов, содержат только 5'-концевую метакриламидную группу, связанную с молекулой олигонуклеотида кислотолабильной фосфорамидной связью, или концевую аллильную группу, обладающую низким сродством к акриламиду N,N'-метиленбисакриламиду в реакции сополимеризации.
3. Известные биочипы отличаются низкой воспроизводимостью свойств гелевых ячеек и неоднородностью распределения в объеме ячейки иммобилизуемого соединения.
4. Известный способ проведения химической полимеризации компонентов композиции при изготовлении биочипа осуществляют в атмосфере влажного азота, что снижает эффективность полимеризации.
5. Известный способ фотоиндуцируемой полимеризации использует УФ-излучение с длиной волны 254 нм, что приводит к деструкции олигонуклеотидов.
6. Известные способы изготовления биочипов технически сложны и используют процедуры, не обеспечивающие необходимой однородности и воспроизводимости свойств гелевых ячеек и плохо поддающиеся автоматизации.
Выше перечисленные недостатки методов диагностики сердечно-сосудистых заболеваний и биочипов для их осуществления устраняются заявляемым изобретением.
Наиболее близким по выполнению и достигаемому результату к заявляемому изобретению является «Способ скрининга сердечно-сосудистых заболеваний и биочип для осуществления этого способа» (RU №2008140355, G01N 33/48, 20.04.2010) [9], принимаемый за прототип.
Способ включает: (а) выделение геномной ДНК из клинического образца и амплификацию гена и получение одноцепочечного флюоресцентно-меченого продукта; (б) приготовление биочипа с иммобилизованными нуклеотидами; (в) гибридизацию амплифицированного меченого продукта на биочипе; (г) регистрацию и (д) интерпретацию результатов гибридизации путем сравнения интенсивности флюоресцентных сигналов, полученных при совершенной и несовершенной гибридизации. Согласно известному изобретнию на стадии (а) используют амплификацию генов из списка SEQ ID 1-432 и получение одноцепочечного флюоресцентно-меченого продукта методом ник-трансляции и рестрикции, при этом на стадии (а) применяется рестриктаза DPN2; на стадии (б) приготавливают биочип для скрининга предрасположенности к заболеваниям сердечно-сосудистой системы, содержащий набор иммобилизованных олигонуклеотидов SEQ ID 1-432; на стадии (в) осуществляют гибридизацию меченого амплифицированного продукта на указанном биочипе; на стадии (г) регистрацию результатов проводят на длине волны 635 нм и 532 нм для красителей Cy5 и Cy3 соответственно; на стадии (д) интерпретацию результатов гибридизации осуществляют путем сравнения интенсивности флюоресцентных сигналов, полученных при совершенной и несовершенной гибридизации: если в ячейке первого ряда такого участка интенсивность сигнала выше, чем в остальных рядах, и выше порогового уровня, определяемого отрицательным контролем, образец относят к дикому типу по данному нуклеотиду (не имеющему мутации в данном положении); если интенсивность сигнала в ячейке 2, 3 или 4 ряда отдельно или попарно (например, ячейки 2 и 3 ряда) выше, чем в остальных рядах, и выше порогового уровня, определяемого отрицательным контролем, образец относят к мутантному типу по данному нуклеотиду; если интенсивность сигнала в ячейке первого ряда и одной из ячеек 2, 3 или 4 ряда выше, чем в остальных ячейках участка, и выше порогового уровня, определяемого отрицательным контролем, то образец относят к гетерозиготному типу. Биочип для скрининга сердечно-сосудистых заболеваний, содержащий набор иммобилизованных олигонуклеотидов SEQ ID 1-432 и 10 неспецифичных олигонуклеотидов в качестве контроля, применение которого позволяет детектировать точечные мутации и делеции в генах FGA, MEF2A, EDN1, АРОВ, АРОА5, АРОА4, АСЕ, AGXT, FBN1, MTHFR, TNBS4, ADRB1, АРОЕ, CCR2 с высокой специфичностью, а порядок размещения олигонуклеотидов обеспечивает возможность наиболее достоверно оценить наличие или отсутствие мутации и ее точную локализацию.
Авторами настоящего изобретения накоплен клинический материал, позволяющий сделать вывод о необходимости значительного расширения списка исследуемых генов и полиморфизмов, необходимых для максимально надежного и полного диагностирования пациента. При проведении обследования пациентов с проявлениями тяжелой сердечной недостаточности известный способ-прототип не всегда позволял обнаружить отклонения в генотипе. На основании дальнейшего углубленного обследования и обнаружения мутаций, не входящих в число определяемых предыдущим биочипом, был сделан вывод о необходимости включения в число идентифицируемых мутаций дополнительно 30 полиморфных сайтов в 15 генах.
Заявляемый способ позволяет получить более полную комплексную оценку генетических предрасположенностей пациента к ССЗ, и с большей достоверностью оценить риски развития сердечно-сосудистых заболеваний.
Задача настоящего изобретения - создание нового ускоренного метода обнаружения точечных нуклеотидных замен в генах человека, ответственных за предрасположенность и развитие сердечно-сосудистых заболеваний. В основу изобретения положена также задача создания биочипа, обеспечивающего оптимальное функционирование иммобилизованных в нем биологически значимых макромолекул.
Поставленная задача решается путем достижения нового технического результата - обнаружения большего количества мутаций в ДНК обследуемого за один анализ биологической жидкости, при использовании одноцепочечных меченых фрагментов ДНК генома человека непосредственно из клинического образца (цельной крови, волос и т.п.). Присутствие или отсутствие мутаций ДНК определяют с помощью гибридизации полученных фрагментов на специализированном олигонуклеотидном биочипе. Способ включает также способы регистрации и интерпретации результатов анализа.
Существенные признаки способа:
(а) - выделение геномной ДНК из клинического образца и амплификацию гена и получение одноцепочечного флюоресцентно меченого продукта; (б) - приготовление биочипа с иммобилизованными нуклеотидами; (в) - гибридизацию амплифицированного меченого продукта на биочипе; (г) - регистрацию и (д) - интерпретацию результатов гибридизации путем сравнения интенсивности флюоресцентных сигналов, полученных при совершенной и несовершенной гибридизации.
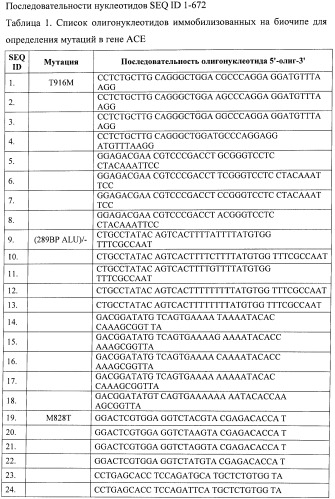
Согласно изобретению, на стадии (а) используют амплификацию генов из списка SEQ ID 1-672 и получение одноцепочечного флюоресцентно меченого продукта методом ник-трансляции и рестрикции, при этом на стадии (а) применяется рестриктаза DPN2; на стадии (б) приготавливают биочип для скрининга предрасположенности к заболеваниям сердечно сосудистой системы содержащий набор иммобилизованных олигонуклеотидов SEQ ID 1-672 на стадии (в) осуществляют гибридизацию меченого амплифицированного продукта на указанном биочипе; на стадии (г) регистрацию результатов проводят на длине волны 635 нм и 532 нм для красителей Cy5 и Cy3 соответственно; на стадии (д) - интерпретацию результатов гибридизации осуществляют путем сравнения интенсивности флюоресцентных сигналов, полученных при совершенной и несовершенной гибридизации: если в ячейке первого ряда такого участка интенсивность сигнала выше, чем в остальных рядах, и выше порогового уровня, определяемого отрицательным контролем, образец относят к дикому типу по данному нуклеотиду (не имеющему мутации в данном положении); если интенсивность сигнала в ячейке 2, 3 или 4 ряда отдельно или попарно (например, ячейки 2 и 3 ряда) выше, чем в остальных рядах и выше порогового уровня, определяемого отрицательным контролем, образец относят к мутантному типу по данному нуклеотиду; если интенсивность сигнала в ячейке первого ряда и одной из ячеек 2, 3 или 4 ряда выше, чем в остальных ячейках участка, и выше порогового уровня определяемого отрицательным контролем, то образец относят к гетерозиготному типу.
Размер амплифицированного одноцепочечного флюоресцентно меченого фрагмента составляет около 60 пар оснований (п.о.).
В качестве клинического образца используют цельную кровь, волосы, слюну сперму, кусочки кожи.
Биочип для осуществления способа расширенного скрининга предрасположенности к сердечно-сосудистым заболеваниям содержит набор иммобилизованных олигонуклеотидов SEQ ID 1-672 и 10 неспецифичных олигонуклеотидов в качестве контроля, применение которого позволяет детектировать точечные мутации и делеции в генах АСЕ, АРОА4, АРОА5, АРОВ, EDN1, MEF2A, FGA, ADRB1, TNBS4, АРОЕ, FBN1, AGXT, MTHFR, CCR2, F5, F2, F7, АВСА1, ITGB3, AGTR2, B2R, DES, TLR4, NOS3, KDR, ММР9, THBS2, LPL, МРО с высокой специфичностью, а порядок размещения олигонуклеотидов обеспечивает возможность наиболее достоверно оценить наличие или отсутствие мутации и ее точную локализацию. При этом биочип содержит набор олигонуклеотидов SEQ ID 1-672 и 10 неспецифичных олигонуклеотидов в качестве контроля, а также олигонуклеотиды, комплементарные олигонуклеотидам SEQ ID 1-672. А также он содержит от 1 до 4 копий набора олигонуклеотидов SEQ ID 1-672.
Новый технический результат изобретения состоит в том, что разработанные нами существенные признаки изобретения позволяют быстро и с высокой точностью провести анализ ДНК на наличие предрасположенности к сердечно-сосудистым заболеваниям, при этом для анализа достаточно одной пробы крови /слюны, волос и т.п./.
Достижение нового технического результата обусловлено следующими положениями.
Применение рестрикции и ник-трансляции позволяет получать меченые фрагменты небольшого размера - около 60 п.о. - геномной ДНК. Получаемые одноцепочечные меченые фрагменты ДНК способны вступать в специфическое гибридизационное взаимодействие с олигонуклеотидами, иммобилизованными на биочипе. Известный порядок расположения олигонуклеотидов на биочипе дает возможность установить наличие или отсутствие мутаций генов, ответственных за развитие сердечно-сосудистых заболеваний в образце ДНК. Разработанные условия подготовки образца и гибридизации обеспечивают высокую степень дифференциации между теми ячейками биочипа, где прошла гибридизация, и теми, где не возникло специфических гибридизационных комплексов. Дизайн олигонуклеотидов обеспечивает абсолютную специфичность их взаимодействия с участками генов, ответственных за развитие сердечно-сосудистых заболеваний, содержащими исследуемую мутацию. При этом разработанный нами уникальный алгоритм формирования олигонуклеотидов биочипа позволяет создавать индивидуальные биочипы с заданными группами генов, мутаций в них и температурой плавления ДНК. Регистрация и обработка результатов осуществляется специализированной программой, позволяющей достоверно определить наличие сигнала по отношению к фоновому уровню.
К выделенной из образца ДНК добавляют рестриктазу DPN2 для разделения геномной ДНК на более мелкие фрагменты. Затем осуществляют ник-трансляцию с помощью ДНК-полимеразы (Klenow Fragment), в результате которой накапливаются флуоресцентно меченые одноцепочечные продукты. Полученные продукты гибридизуют на биочипе с иммобилизованными зондами, комплементарными последовательностям фрагментов исследуемых участков генов.
Подробное описание способа и клинические примеры его выполнения
Обработка клинического образца
Целевая нуклеиновая кислота, в которой хотят определить интересующие полиморфизмы, обычно изолируется от образца ткани. Если цель - геномная ДНК, образец может быть получен из любой ткани (за исключением эритроцитов). Например, цельная кровь, лимфоциты периферической крови, кожа, волосы или сперма - подходят для получения клинических образцов. Данные источники также подходят, если для анализа используется РНК. Если для анализа используется иРНК, образец должен выделяться из тканей, в которых экспрессируется иРНК. Если нуклеиновая кислота в образце - РНК, она обычно подвергается обратной транскрипции в ДНК. Если необходимо, ДНК образца или кДНК, полученная в результате обратной транскрипции, амплифицируется, например, с помощью ПЦР.
Набор олигонуклеотидов (ячейки биочипа), приведенных в SEQ ID 1-672, наносят на стеклянную подложку в виде капель с максимальной аккуратностью и воспроизводимостью с помощью роботов (например, BioOdyssey, Bio-Rad, США). Каждая сформированная ячейка представляет собой полусферу диаметром 1 мкм и периодом около 8 мкм и содержит иммобилизованный олигонуклеотид - зонд, комплементарный последовательности участка гена, связанного с наличием или отсутствием предрасположенности к сердечно-сосудистым заболеваниям.
Олигонуклеотиды для иммобилизации на биочипе синтезированы на автоматическом синтезаторе (типа 394 DNA/RNA synthesizer, Applied Biosystems, США) и содержат спейсер со свободной аминогруппой 3'-Amino-Modifier C7 CPG 500 (Glen Research) для последующей иммобилизации на подложку биочипа.
Из образца выделяют геномную ДНК с помощью коммерческого набора DNA Box 500 (Protrans).
Полученную геномную ДНК фрагментируют с помощью рестриктазы DPN2. После фрагментации ДНК проводят реакцию ник-трансляции с помощью Klenow Fragment (Sigma, 40 ед/мкл) в присутствии Cy5-dCTP или Cy3-dCTP (Amersham, 1 мМ раствор). Затем очищают ДНК с помощью фильтра microcon 30 (Amicon/Millipore) и проводят гибридизацию на биочипе.
Гибридизацию меченого образца на биочипе проводят в буфере следующего состава: 1М GuSCN; 50 мМ HEPES, рН 7,5; 5 мМ ЭДТА, рН 7.0. Гибридизацию выполняют в коммерчески производимой гибиридизационной камере объемом 30 мкл (Sigma) в течение ночи (16-18 часов) при 65°С. Отмывку проводят трижды в следующих буферах.
Первая отмывка: 2Х SSC, 0.03% SDS, 5 мин 70°С (для точного отделения специфичного от неспецифичного гибридизационного сигнала).
Вторая отмывка: 1X SSC, 5 мин при комнатной температуре.
Третья отмывка: 0.2Х SSC, 5 мин при комнатной температуре.
Регистрация и интерпретация результатов гибридизации - стадия (д)
Регистрацию и фиксирование гибридизационной картины проводят с помощью автоматического комплекса, состоящего из сканера биочипов типа Innoscan 700 (Inopsys) и компьютера на длине волны 635 нм и 532 нм для красителей Cy5 и Cy3 соответственно. Обработку интенсивности флуоресцентного сигнала проводят визуально или с помощью программы MapixSoftware (Inopsys). Сигналы обрабатываются программой в соответствии с порядком нанесения олигонуклеотидов на чип.
Олигонуклеотиды синтезируют на автоматическом синтезаторе ДНК и наносят на биочип таким образом, что олигонуклеотиды, комплементарные участку гена прямой цепи ДНК («прямые» олигонуклеотиды), содержащему 1 положение мутации, расположены в столбик один за другим, при этом верхнее положение занимает олигонуклеотид, комплементарный дикому типу гена (т.е. не содержащий мутации). Олигонуклеотиды, комплементарные участку гена обратной цепи ДНК, содержащему соответствующее положение мутации, располагают таким же образом, как и «прямые». Олигонуклеотиды располагают на биочипе случайным образом с учетом выше указанных условий. Для увеличения чувствительности метода биочип может содержать от 2-х до нескольких копий набора олигонуклеотидов. Кроме того, на биочипе расположены 10 ячеек, содержащих неспецифичные к исследуемому геному олигонуклеотиды, выполняющие роль отрицательного контроля.
Интерпретацию результатов проводят следующим образом.
Интерпретацию результатов гибридизации осуществляют путем сравнения интенсивности флюоресцентных сигналов, полученных при совершенной и несовершенной гибридизации. Внутри каждого участка биочипа размером 1 на 4 ячейки визуально или с помощью программного обеспечения сравнивают интенсивности флуоресцентного сигнала. Если в ячейке первого ряда такого участка интенсивность сигнала выше, чем в остальных рядах, и выше порогового уровня, определяемого отрицательным контролем, образец относят к дикому типу по данному нуклеотиду (не имеющему мутации в данном положении). Если интенсивность сигнала в ячейке 2-го, 3-го или 4-го ряда отдельно или попарно (например, ячейки 2-го и 3-го ряда) выше, чем в остальных рядах, и выше порогового уровня, определяемого отрицательным контролем, образец относят к мутантному типу по данному нуклеотиду (тип и расположение мутации устанавливают путем сравнения со схемой расположения олигонуклеотидов на биочипе). Если интенсивность сигнала в ячейке первого ряда и одной из ячеек 2-го, 3-го или 4-го ряда выше, чем в остальных ячейках участка, и выше порогового уровня, определяемого отрицательным контролем, то образец относят к гетерозиготному типу (одна аллель - дикий тип, вторая аллель - мутантный тип). В результате выдается заключение о наличии или отсутствии мутаций в анализируемых положениях соответствующих генов.
Наличие или отсутствие мутаций генов, ответственных за развитие СС заболеваний, свидетельствует о предрасположенности к развитию СС заболеваний или отсутствии данной предрасположенности.
Олигонуклеотидный биочип для скрининга на сердечно-сосудистые заболевания
Олигонуклеотиды для иммобилизации на биочипе синтезируют на автоматическом синтезаторе (типа 394 DNA/RNA synthesizer, Applied Biosystems, США) и содержат спейсер со свободной аминогруппой 3'-Amino-Modifier C7 CPG 500 (Glen Researh) для последующей иммобилизации на подложку биочипа или 5'-Amino-Modifier C6 (Glen Researh) для введения флуоресцентной метки. Введение флуоресцентной метки Cy5 осуществляют в соответствии с рекомендациями производителя (Amersham).
Синтезированный набор олигонуклеотидов (ячейки биочипа) наносят на стеклянную подложку в виде капель с максимальной аккуратностью и воспроизводимостью с помощью роботов (BioOdyssey, Bio-Rad, США). Каждая сформированная ячейка представляет собой полусферу диаметром 1 мкм и периодом около 8 мкм и содержит иммобилизованный олигонуклеотид - зонд, комплементарный последовательности участка гена, связанного с наличием или отсутствием предрасположенности к сердечно-сосудистым заболеваниям.
Структура биочипа
Биочип представляет собой набор олигонуклеотидов, иммобилизованных на твердой подложке.
Данный набор олигонуклеотидов формируется согласно следующему алгоритму:
1. Формируют базу данных из последовательностей генов и их мутаций популяционных частотах (этих мутаций), связанных с развитием сердечно-сосудистых заболеваний.
2. Задают группу генов, выбранную по медицинским показаниям для отдельного пациента.
3. Выбирают для указанных выше генов мутации:
популяционная частота встречаемости которых при заболевании более 70%;
популяционная частота встречаемости которых при заболевании более 25%;
все встречающиеся мутации при заболевании.
4. Задают температуру плавления ДНК Тпл и концентрацию ионов натрия [Na+], при которых планируется проводить диагностику, и запоминают.
5. Для каждой выбранной мутации запоминают номер позиции замены нуклеотида в цепи ДНК - n1, n2, n3…ni и осуществляют следующий цикл обработки.
6. Задают номера нуклеотидов K=n-30, L=n+30 и запоминают.
7. Выбирают отрезок гена a, ограниченный нуклеотидами К и L, и запоминают.
8. Рассчитывают температуру плавления Тпл1 ДНК для отрезка ДНК a, ограниченного нуклеотидами К и L, по известной формуле (Good, N.Е., Biochemistry 5, 467-476, 1986).
Tпл1=81.5+Log[Na+]+0.41*(G+C)/N-600/N, где:
[Na+] - заданная концентрация ионов натрия,
G - количество оснований гуанина в отрезке a,
С - количество оснований цитозина в отрезке a,
N - количество всех оснований в отрезке a,
и запоминают.
9. Сравнивают запомненные значения Тпл и Тпл1.
10. При Тпл<Tпл1-0,5 переходят к пункту 11, при Тпл>Tпл1+0,5 переходят к пункту 16, при Тпл=Тпл1±0.5 переходят к пункту 21.
11. Определяют номер олигонуклеотида L1 в отрезке ДНК a по формуле L1=L-1 и присваивают номеру олигонуклеотида L значение L1, что соответствует уменьшению отрезка a на один нуклеотид справа, и запоминают.
12. Повторяют последовательность действий указанных выше пунктов 7-9 для этого отрезка ДНК a и при Тпл<Тпл1-0,5 переходят к пункту 13, при Тпл=Tпл1±0,5 переходят к пункту 21.
13. Определяют номер олигонуклеотида К1 в отрезке ДНК a по формуле К1=К+1 и присваивают номеру олигонуклеотида К значение К1, что соответствует уменьшению отрезка a на один нуклеотид слева, и запоминают.
14. Повторяют последовательность действий указанных выше пунктов 7-9 для этого отрезка ДНК a и при Тпл<Tпл1+0,5 переходят к пункту 11, при Тпл=Тпл1±0.5 переходят к пункту 21.
15. Продолжают цикл обработки, указанный в пунктах 7-14, поочередно с уменьшением отрезка ДНК a то справа, то слева, до тех пор, пока температура плавления отрезка ДНК Тпл1 не станет равной заданной температуре плавления ДНК Тпл. В результате получен отрезок ДНК, содержащий мутацию, характерную для одного заболевания.
16. Определяют номер олигонуклеотида L1 в отрезке ДНК a по формуле L1=L+1 и присваивают номеру олигонуклеотида L значение L1, что соответствует увеличению отрезка a на один нуклеотид справа.
17. Повторяют последовательность действий указанных выше пунктов 7-9 для этого отрезка ДНК a и при Тпл>Tпл1+0,5 переходят к пункту 18, при Тпл=Тпл1±0.5 переходят к пункту 21.
18. Определяют номер олигонуклеотида К1 в отрезке ДНК a по формуле К1=К-1 и присваивают номеру олигонуклеотида К значение К1, что соответствует увеличению отрезка a на один нуклеотид слева.
19. Повторяют последовательность действий указанных выше пунктов 7-9 для этого отрезка ДНК a и при Тпл>Тпл1+0,5 переходят к пункту 18, при Тпл=Tпл1±0.5 переходят к пункту 21.
20. Продолжают цикл обработки, указанный в пунктах 7-9 и 16-19, поочередно с увеличением отрезка ДНК a то справа, то слева, до тех пор, пока температура плавления отрезка ДНК Тпл1 не станет равной заданной температуре плавления ДНК Тпл. В результате получен отрезок ДНК, содержащий мутацию, характерную для одного заболевания.
21. Получен отрезок ДНК a с заданной температурой плавления, содержащий мутацию, характерную для одного заболевания.
22. Формируют 3 отрезка a 1, a 2 и a 3, идентичных отрезку гена a, за исключением нуклеотида в положении n, таким образом, что все четыре отрезка a-a 3 имеют различные нуклеотиды (А, Т, G и С) в положении n.
23. Формируют 4 олигонуклеотида b, b1, b2, b3, комплементарные соответствующим олигонуклеотидам a, a 1, a 2, a 3. В результате получают 8 олигонуклеотидов для одной мутации: 4 прямых и 4 обратных, содержащих все варианты точечных замен в позиции n.
24. Повторяют весь цикл обработки, указанный в п.п.7-23 для каждой выбранной мутации. В результате получают сформированный набор последовательностей олигонуклеотидов, содержащих мутации генов, характерных для всех заданных групп сердечно-сосудистых заболеваний.
Для детекции мутаций типа SNP используют набор олигонуклеотидов, состоящий из двух частей. Первая часть представляет собой множество олигонуклеотидов, каждый олигонуклеотид точно комплементарен участку ДНК гена, содержащему исследуемую позицию. Вторая часть набора состоит из олигонуклеотидов, точно комплементарных участку ДНК гена, содержащему исследуемую позицию, за исключением нуклеотида в исследуемой позиции таким образом, что на одну исследуемую позицию приходится 3 олигонуклеотида с разными нуклеотидами в исследуемом положении. В итоге на одно исследуемое положение для одной цепи ДНК приходится 4 олигонуклеотида, отличающиеся нуклеотидом в исследуемом положении. Такой подход позволяет определить, какой из нуклеотидов А, С, G или Т находится в исследуемой позиции и, соответственно, сделать заключение о наличии или отсутствии мутации в данном положении. В набор олигонуклеотидов биочипа входят олигонуклеотиды, комплементарные как прямой цепи ДНК гена, так и обратной. Кроме того, набор олигонуклеотидов может быть нанесен на биочип в нескольких копиях (от 1 до 4 копий). Это позволяет повысить чувствительность анализа.
Для детекции делеций используют набор олигонуклеотидов, состоящий из двух частей. Первая часть набора олигонуклеотидов такая же, как и для детекции мутаций типа SNP. Вторая часть состоит из множества олигонуклеотидов, точно комплементарных участку ДНК гена, за исключением того, что в данных олигонуклеотидах отсутствует нуклеотид(ы) в исследуемом положении. В результате для исследования одной делеций в одной цепи ДНК используется 2 олигонуклеотида. В набор олигонуклеотидов биочипа входят олигонуклеотиды, комплементарные как прямой цепи ДНК гена, так и обратной. Кроме того, набор олигонуклеотидов может быть нанесен на биочип в нескольких копиях (от 1 до 4 копий).
Олигонуклеотиды синтезируют на автоматическом синтезаторе ДНК и наносят на биочип таким образом, что олигонуклеотиды, комплементарные участку гена прямой цепи ДНК («прямые» олигонуклеотиды), содержащему 1 положение мутации, расположены в столбик один за другим, при этом верхнее положение занимает олигонуклеотид, комплементарный дикому типу гена (т.е. не содержащий мутации). Олигонуклеотиды, комплементарные участку гена обратной цепи ДНК, содержащему соответствующее положение мутации, располагают таким же образом, как и «прямые». Олигонуклеотиды располагают на биочипе случайным образом с учетом выше указанных условий. Для увеличения чувствительности метода биочип может содержать от 2-х до нескольких копий набора олигонуклеотидов. Кроме того, на биочипе расположены 10 ячеек содержащих неспецифичные к исследуемому геному олигонуклеотиды, выполняющие роль отрицательного контроля.
Клинический пример 1
Женщина, 48 лет. Легкая гипертензия. Артериальное давление находится в пределах 150-160/90-100 мм рт.ст. В анамнезе проявления гипертензии после 40 лет. В семье, кроме пациентки, диагноз гипертензии поставлен у родственников первой степени родства, у отца ишемическая болезнь сердца, у матери гипертензия.
Было проведено молекулярно-генетическое типирование по заявляемому методу с помощью биочипа на наиболее частые мутаций в генах АСЕ, АРОА4, АР