Способ выбора днк-зондов для микрочиповой диагностики, биочип и способ типирования гена нейраминидазы и гемагглютинина вируса гриппа а
Иллюстрации
Показать всеИзобретение относится к молекулярной биологии, генетической инженерии и медицине. Предлагаемый способ позволяет определять структуру представительных зондов для такого рода микрочипов. Например, предлагаемый способ позволяет определять различные типы гена нейраминидазы вируса гриппа A. Способ основан на проведении двуэтапной полимеразной цепной реакции (ПЦР) кДНК вируса с последующей гибридизацией полученных флуоресцентно-меченых ампликонов с ДНК-микрочипом, содержащим соответствующие дискриминирующие гибридизационные зонды. Использование этого способа позволяет определять все 9 субтипов гена нейраминидазы вируса гриппа A. Изобретение предназначено для использования при конструировании микрочипов, создаваемых для выявления целевых микроорганизмов с высоковариабельным геномом. 3 н.п. ф-лы, 9 ил., 6 пр.
Реферат
Изобретение относится к молекулярной биологии, генетической инженерии, медицине и предназначено для использования при конструировании микрочипов, создаваемых для выявления целевых микроорганизмов с высоковариабельным геномом. Предлагаемый способ позволяет определять структуру представительных зондов для такого рода микрочипов. Например, предлагаемый способ позволяет находить зонды для выявления различных типов генов нейраминидазы и гемагглютинина вируса гриппа A. Предлагаемый способ типирования вируса гриппа основан на проведении двуэтапной полимеразной цепной реакции (ПЦР) кДНК вируса с последующей гибридизацией полученных флуоресцентно-меченых ампликонов с ДНК-микрочипом, содержащим соответствующие дискриминирующие гибридизационные зонды. Использование этого способа позволяет определять все 9 субтипов гена нейраминидазы вируса.
Биологические микрочипы в настоящее время используются для параллельного анализа специфических взаимодействий биологических макромолекул. За короткое время биологические микрочипы выделились в самостоятельную область анализа с приложениями от исследования фундаментальных проблем молекулярной биологии и молекулярной эволюции до практических применений в медицине, фармакологии, экологии, судебно-медицинской экспертизе и других областях. Возможность параллельного анализа одного образца на множестве типирующих зондов чрезвычайно важна для диагностики и типирования исследуемого микроорганизма. Это позволяет снизить время анализа от нескольких дней или даже недель до нескольких часов. Биологические микрочипы были успешно использованы для диагностики: вируса папилломы (An, H.J., Cho, N.H., Lee, S.Y., Kim, I.H., Lee, C., Kim, S.J., Mun, M.S., Kim, S.H. and Jeong, J.K. (2003) Correlation of cervical carcinoma and precancerous lesions with human papillomavirus (HPV) genotypes detected with the HPV DNA chip microarray method. Cancer, 97, 1672-1680), вируса атипичной пневмонии (SARS) (Wang, D., Urisman, A., Liu, Y.T., Springer, M., Ksiazek, Т.G., Erdman, D.D., Mardis, E.R., Hickenbotham, M., Magrini, V. et al. (2003) Viral discovery and sequence recovery using DNA microarrays. PLoS Biol., 1, E2), корона- и риновирусов (Kistler, A., Avila, P.C., Rouskin, S., Wang, D., Ward, Т., Yagi, S., Schnurr, D., Ganem, D., DeRisi, J.L. et al. (2007) Pan-viral screening of respiratory tract infections in adults with and without asthma reveals unexpected human coronavirus and human rhinovirus diversity. J. Infect. Dis., 196, 817-825), филовирусов и малярийного паразита (Palacios, G., Quan, P.L., Jabado, O.J., Conlan, S., Hirschberg, D.L., Liu, Y., Zhai, J., Renwick, N., Hui, J. et al. (2007) Panmicrobial oligonucleotide array for diagnosis of infectious diseases. Emerg. Infect. Dis., 13, 73-81), ортопоксвирусов (Ryabinin VA, Shundrin LA, Kostina EB, Laassri M, Chizhikov V, Shchelkunov SN, Chumakov K, Sinyakov AN. Microarray assay for detection and discrimination of Orthopoxvirus species. 2006. J. Med. Virol. V.78, No 10, p.1325-1340), ротавирусов (Chizhikov, V., Wagner, M., Ivshina, A., Hoshino, Y., Kapikian, A.Z. and Chumakov, K. (2002) Detection and genotyping of human group A rotaviruses by oligonucleotide microarray hybridization. J. Clin. Microbiol., 40, 2398-2407) и ряда других микроогранизмов.
Успех разработки микрочиповой диагностики, основанной на гибридизации типирующих зондов с нуклеиновой кислотой образца патогена, зависит от правильного выбора дискриминирующих олигонуклеотидных зондов микрочипа. Задача поиска типирующих зондов решается достаточно просто для сильно различающихся по структуре геномных последовательностей, например, при нахождении родоспецифичных зондов и в большинстве случаев не представляет каких-либо проблем. Так отличить вирус оспы от вируса герпеса достаточно легко (Ryabinin VA, Shundrin LA, Kostina EB, Laassri M, Chizhikov V, Shchelkunov SN, Chumakov K, Sinyakov AN. Microarray assay for detection and discrimination of Orthopoxvirus species. 2006. J. Med. Virol. V.78, No 10, pp.1325-1340). Задача существенно осложняется при поиске зондов в высоковариабельных геномах.
Известен способ (Wernersson, R., and Н.В. Nielsen. 2005. Oligo Wiz 2.0 - integrating sequence feature annotation into the design of microarray probes. Nucleic Acids Res. 33: W611-W615, прототип) для выбора микрочиповых зондов. Метод основан на попарном анализе нуклеотидных последовательностей с целью выявления консервативных районов в этих последовательностях. Затем в выявленных консервативных районах выбираются микрочиповые ДНК-зонды. При этом подбираются зонды, образующие близкие температуры плавления с мишенью, отсеиваются зонды, имеющие внутренние шпильки. Успех выбора зондов определяется наличием консервативных районов в анализируемых последовательностях. Если консервативных последовательностей программа в анализируемых последовательностях не находит, ДНК-зонды не могут быть выбраны.
В случае высоковариабельных геномов данный подход очень трудно, если вообще возможно, реализовать, например, в случае вируса гриппа типа A. При попытке создать микрочип, выявляющий субтипы вируса гриппа A по генам нейраминидазы и геммаглютинина, выясняется, что невозможно найти одни и те же типирующие зонды для всех известных штаммов. Это обусловлено с одной стороны, высокой вариабельностью генома, а с другой стороны, большим числом (несколько тысяч) отсеквенированных геномных последовательностей вируса гриппа. Поэтому стратегия выбора зондов в таких случаях должна быть модифицирована.
Для выбора дискриминирующих зондов в случае высоковариабельных геномов мы предлагаем использовать новый подход. При разработке модифицированной стратегии выбора зондов мы исходили из предположения, что при типировании микроорганизмов более важны сходные пептидные элементы белков исследуемого микроорганизма, а не кодирующие их нуклеиновые кислоты, поскольку именно белки и пептиды ответственны за биологические эффекты. Поэтому первым этапом поиска микрочиповых зондов в нашем методе является анализ аминокислотных последовательностей выбранных генов микроорганизма и выявление консервативных пептидов, характерных только для определенного субтипа микроогранизма. На следующем этапе для выявленных отобранных пептидов с помощью таблицы кодонов аминокислот определяются все возможные кодирующие их нуклеотидные последовательности. Из полученных последовательностей с помощью описанной ниже процедуры отбирают набор зондов, обеспечивающих типирование целевого гена.
Предлагаемый подход опробован на примере типирования вируса гриппа A по вирусным маркерам - генам нейраминидазы и гемагглютинина.
Всего известно 9 субтипов нейраминидазы и учитывая, что протяженность гена нейраминидазы составляет примерно 1500 пар оснований, нахождение по 15-25 зондов, специфичных по отношению к каждому из 9 подтипов нейраминидазы, является сложной задачей. Отбор олигонуклеотидных зондов для определения подтипа нейраминидазы проводился с использованием разработанного программного обеспечения в два этапа. Первый этап включает в себя отбор специфичных аминокислотных последовательностей, характерных для определяемого подтипа, но отсутствующих в других белковых последовательностях анализируемого набора белков. На втором этапе проводится отбор олигонуклеотидных зондов, исходя из структуры пептидов.
Работа по расчету зондов включает следующие стадии:
- отбор белковых и нуклеиновых последовательностей нейраминидазы;
- отбор пептидов, общих для определяемого субтипа нейраминидазы;
- удаление из полученного набора тех пептидов, которые присутствуют в белковых последовательностях других подтипов нейраминидазы;
- расчет олигонуклеотидных зондов исходя из структуры пептидов;
- выбор наиболее представительных олигонуклеотидных зондов;
- удаление из полученного набора тех олигонуклеотидных зондов, введение в которые одной или двух точечных замен дают зонды, характерные для других подтипов нейраминидазы;
- выбор олигонуклеотидных зондов с оптимальной температурой плавления.
В работе использовали данные GENBANK (http://www.ncbi.nlm.nih.gov) по нейраминидазе вируса гриппа A по март 2008 г.
Как видно из Таблицы 1, различные подтипы нейраминидазы представлены различным количеством последовательностей. Наиболее многочисленны подтипы N1 и N2; остальные подтипы присутствуют в существенно меньшей степени. Внутри определенного подтипа нейраминидазы имеется дифференциация, связанная с подтипом гемагглютинина. Так, нейраминидаза N1 в наибольшей степени представлена подтипом гемагглютинина H1 и H5, тогда как некоторые из подтипов гемагглютинина проявляются незначительно (H4, H10, H12) или не представлены вовсе (H8, H13, H14, H15).
В полученной базе данных для каждого серотипа нейраминидазы (набор для всех субтипов геммагглютинина) выявляют общие пептиды, длиной в 7 аминокислотных остатков. Предварительный анализ показал, что выбор более длинных пептидов снижает число зондов, общих для определяемого подтипа, а более короткие являются менее специфичными. При анализе рассматривались только последовательности длиной не менее 200 аминокислотных остатков. Поскольку найти общие пептиды для абсолютно всех белков данного серотипа не удается как в силу вариабельности белковой последовательности, так из-за того, что в GenBank представлены частично секвененированные последовательности, то отбирались пептиды, содержащиеся не менее чем в 90% белков. Полученный набор пептидов для каждого серотипа нейраминидазы вируса гриппа определяет практически 100% всех штаммов и изолятов анализируемого типа.
На следующем этапе анализа производят селекцию пептидных наборов, характеризующих серотипы нейраминидазы. Из полученных наборов удаляют пептиды одной и той же структуры, но принадлежащие к наборам разных серотипов. В результате получают набор 7-членных пептидов, характеризующих определенный подтип нейраминидазы.
На следующей стадии набор 7-членных пептидов переводят в набор 21-членных олигонуклеотидов, кодирующих эти пептиды.
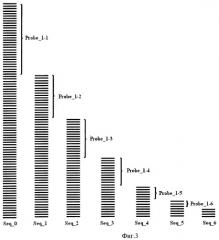
Очевидно, что общее число олигонуклеотидных зондов, представляющих весь набор пептидов, слишком велико и может достигать нескольких тысяч. Поскольку многие из них определяют лишь несколько последовательностей ДНК, то следующим этапом является отбор из исходного полного набора зондов наиболее оптимальной по размеру выборки олигонуклеотидов (20-24 зонда), позволяющей с наибольшей вероятностью определять субтип нейраминидазы. На первый взгляд достаточно выбрать наиболее часто встречающиеся зонды. Но при таком подходе могут быть селективно отобраны зонды только для нейраминидазы с наиболее представленным субтипом гемагглютина. В случае субтипов нейраминидазы первого типа (H_X_N1) это будут последовательности H1N1 - 1140 последовательности из 2696 (42%) и H5N1 - 1295 последовательностей (48%). Для уменьшения влияния «фактора разной представленности» нейраминидазы первого типа различными подтипами гемагглютинина используется следующий прием (Фиг.2). Из полного набора зондов (Probe_Total) отбирается наиболее представительный (Probe_1-1) для полного набора ДНК последовательностей (Seq_0). На следующей стадии из полного набора последовательностей ДНК отбрасываются все те, которые содержат первый зонд (Probe_1-1), и получают набор Seq_1. Для этого набора находят наиболее представительный олигонуклеотидный зонд (Probe_1-2). Из набора Seq_1 отбрасывают последовательности, содержащие зонд Probe_1-2, и получают набор Seq_2. Операции повторяют до получения зондов Probe_1-3, Probe_1-4, Probe_1-5, Probe_1-6.
На следующей стадии из набора Probe_Total отбрасывают первые шесть зондов (Probe_1-1) - (Probe_1-6) и из оставшихся олигонуклеотидных зондов отбирают по той же схеме еще шесть зондов, используя в качестве стартового полный набор ДНК (Seq_0). Операцию повторяют еще 3 раза и получают 30 зондов.
Следует отметить, что в случае малого количества последовательностей ДНК (например, для нейраминидазы четвертого типа, представленной 31 последовательностью) уже Probe_1-1 - Probe_1-3 определяли исходный набор ДНК. Тогда как, для нахождения большего количества зондов (20-24) расчет проводится не 5, а большее число раз. По мере увеличения числа итераций наиболее представительные зонды выявляют меньше штаммов целевого субтипа, а наименее представительные - больше.
Возможны и другие варианты селекции зондов. Например, для более полного учета минорных последовательностей ДНК (таких как H8N1, H10N1 и т.д.) можно модифицировать описанный выше подход. В процессе расчета из полного набора ДНК-последовательностей (Seq_0) можно исключить те, которые содержат больше заданного числа зондов (N_tr). Варьируя значение этого параметра можно изменять долю зондов, определяющих нейраминидазу первого типа в минорных наборах ДНК. Чем меньше значение N_tr, тем большее число последовательностей ДНК из полного набора будет иметь не менее N_tr зондов в своем составе, тогда как при больших значениях этого параметра расчет будет проводиться фактически по первому варианту. В рассматриваемом нами случае для всех оставшихся субтипов нейраминидазы используется набор из 30 олигонуклеотидных зондов.
Для того чтобы уменьшить число ложноположительных результатов, олигонуклеотидные зонды, образующие ДНК-дуплексы с кДНК нецелевых субтипов с одним или двумя мисматчами, не используются в разрабатываемом микрочипе.
Следующей стадией модификации зондов является их укорачивание или удлинение для выравнивания температур плавления ДНК-гибридов. Следует отметить, что для этого не ставились слишком жесткие условия. Разброс интервала температур плавления ДНК-гибридов был выбран равным 10°C. Последовательность зонда укорачивали либо удлиняли на 1-2 нуклеотидных основания. Модифицированные зонды подвергали проверке на наличие в последовательностях других подтипов.
Подобным же образом были рассчитаны зонды и для других серотипов нейраминидазы. Количество зондов для определения конкретного серотипа нейраминидазы варьировалось от 19 до 24. Всего был отобран 191 зонд (Фиг.3).
Для объективного представления результатов гибридизации на микрочипе важно проводить инструментальный анализ результатов сканирования чипа. Для этого после проведения сканирования проводился анализ флуоресценции спотов с использованием программы ScanArray Express (Perkin-Elmer).
При определения субтипа нейраминидазы вируса гриппа использовали подход, основанный на определении средней флуоресценции спота каждого подтипа (Yµ)
,
где µ - подтип нейраминидазы,
i - номер спота,
N - число спотов на чипе для используемых для определения подтипа нейраминидазы.
Такой подход был выбран потому, что даже в случае несовершенных дуплексов, имеет место связывание зондов с анализируемой ДНК и степень соответствия структуры зонда структуре ДНК всегда соответствует уровню флуоресценции спотов. Т.е. в большей степени будут флуоресцировать споты, характерные для определяемого подтипа нейраминидазы, чем таковые для всех других подтипов.
Аналогично определяли субтип гемагглютинина вируса гриппа. Всего существует 16 субтипов вируса гриппа A. Как и для нейраминидазы, представленность подтипов гемагглютинина в базах данных была различной - максимальна для подтипов H1, H3, H5 и существенно более низкой для остальных подтипов. Наименее представлен подтип H14 - всего две последовательности, в связи с чем расчет зондов для данного подтипа не проводился. Для определения каждого подтипа гемагглютинина использовалось от 13 (пандемический свиной грипп H1N1) до 28 (гемагглютинин третьего типа) олигонуклеотидных зондов. Общее число зондов, использованных в микрочипе, составило 325.
Принципиальная схема типирования гена нейраминидазы вируса гриппа A на микрочипе включает следующие стадии:
Из образца слизистой ткани выделяют суммарную РНК;
с помощью универсального праймера 5'-GACTAATACGACTCACTATAGGGAGCAAAAGCAGG-3', специфичного к РНК вируса гриппа A, получают кДНК;
3. с помощью праймеров 5'-CTATAGGGAGCAAAAGCAGGAGT-3' и 5'-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3', специфичных к гену нейраминидазы, из кДНК путем проведения ПЦР получают полноразмерный ампликон гена нейраминидазы вируса гриппа A;
4. полученный ампликон реамплифицируют в асимметричном режим с праймерами:
5'-CTATAGGGAGCAAAAGCAGGAGT-3',
5'-TAACAGGA(G/A)CA(C/T)TCCTC(A/G)TA(G/A)TG-3' и
5'-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3',
5'-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3'
и метят путем введения флуоресцентной метки в его состав;
5. на следующей стадии проводят гибридизацию полученных ампликонов с дискриминирующими зондами, входящими в состав разработанного микрочипа;
6. анализ результатов гибридизации проводят с помощью сканера микрочипов и программы Scanarray Express. Результаты анализа представляются в графической форме с помощью программы Microsoft Office Excel.
Принципиальная схема типирования гена гемагглютинина вируса гриппа A на микрочипе включает следующие стадии:
1. Из образца слизистой ткани выделяют суммарную РНК;
2. с помощью универсального праймера 5'-GACTAATACGACTCACTATAGGGAGCAAAAGCAGG-3', специфичного к РНК вируса гриппа A, получают кДНК;
3. с помощью праймеров 5'-GACTCACTATAGGGAGCAAAAGCAGGGG-3' и 5'-GTGACACTATAGAAGTAGAAACAAGGGTGTTTT-3', специфичных к гену гемагглютинина, из кДНК путем проведения ПЦР получают полноразмерный ампликон гена гемагглютинина вируса гриппа A;
4. полученный ампликон реамплифицируют в асимметричном режим с праймерами:
5'-GACTCACTATAGGGAGCAAAAGCAGGGG-3'
5'-59-TCWATRAANCCNGCDATDGCHCC-3'
5'-GGDGCHATHGCNGGNTTYATWGA-3'
5'-GGTGACACTATAGAAGTAGAAACAAGGGTGTTTT-3' и метят путем введения флуоресцентной метки в его состав;
5. на следующей стадии проводят гибридизацию полученных ампликонов с дискриминирующими зондами, входящими в состав разработанного микрочипа;
6. анализ результатов гибридизации проводят с помощью сканера микрочипов и программы Scanarray Express. Результаты анализа представляются в графической форме с помощью программы Microsoft Office Excel.
Изобретение иллюстрируется следующими графическими материалами:
Фиг.1. Таблица 1. Количество анализированных серотипов нейраминидазы вируса гриппа типа А.
Фиг.2. Таблица 2. Количество анализированных серотипов гемагглютинина вируса гриппа типа A.
Фиг.3. Схема селекции олигонуклеотидных зондов для типирования гена нейраминидазы первого типа вируса гриппа A.
Фиг.4. Таблица 3. Структура олигонуклеотидных зондов для выявления серотипов нейраминидазы вируса гриппа A.
Фиг.5. Таблица 4. Структура олигонуклеотидных зондов для выявления серотипов гемагглютинина вируса гриппа A.
Фиг.6. Структура микромассива микрочипа, выявляющего субтип гена нейраминидазы вируса гриппа A.
Фиг.7. Результат типирования ампликонов гена нейраминидазы вируса гриппа A на микрочипе. Вертикальной чертой указана относительная ошибка измерения значения флуоресценции.
Фиг.8. Структура микромассива микрочипа, выявляющего субтип гена гемагглютинина вируса гриппа A.
Фиг.9. Результат типирования ампликонов гена гемагглютинина вируса гриппа A на микрочипе. Вертикальной чертой указана относительная ошибка измерения значения флуоресценции.
Для лучшего понимания сущности изобретения ниже приведены примеры его конкретного выполнения.
Пример 1. Изготовление микрочипов для типирования генов нейраминидазы геммаглютинина вируса гриппа A
Печать микрочипов осуществляли на стеклянных, содержащих изотиоцианатную группу слайдах методом контактной печати на споттере BioOdyssey Calligrapher miniarrayer («Bio-Rad») одним пином. Каждый слайд содержал 4 эквивалентных микромассивов зондов (микроэрреея), пригодных для одновременной гибридизации 4 образцов. Средний диаметр спотов (ячеек микрочипа с иммобилизованными зондами) был ~360 µm, расстояние между спотами - 380 µm.
Формат микроэррея составлял 24×25 спотов. Микромассив микрочипа для типирования гена нейраминидазы содержал 24×9 спотов (фиг.6), а микромассив микрочипа для типирования гена гемагглютинина - 24×9 слотов (фиг.8). Печать проводилась из 384-луночных планшетов (24×16). Каждому подтипу нейраминидазы или гемагглютинина отводилась одна строка. На фиг.6 и 8 приведены расположение зондов в планшетах и, соответственно, в микромассивах микрочипа.
Для удобства работы и нормирования сигнала в крайние ячейки плашки добавлялся маркерный зонд, несущий на 3'-конце аминогексильную группу, необходимый для иммобилизации на поверхности слайда. На 5'-конце зонда находится флуорофор на основе тетраметилродамина (Tamra), позволяющий визуализировать границы субэррея, определять качество печати чипа и проводить нормировку сигнала флуоресценции.
В каждую ячейку планшета при печати добавлял 60 мкл раствора А (135 мМ NaHCO3) и 20 мкл раствора зонда (С=200 нмоль/мл). Т - 80 мкл 6 нМ раствор Tamra (dT8)~NH2 в 100 мМ NaHCO3. В буфер - 60 мкл раствора А (135 мМ NaHCO3) и 10 мкл воды.
Пример 2. Синтез кДНК вируса гриппа A
Геномную РНК выделяли из 140 мкл клеточной культуры (титр вируса 105-107 pfu/мл) с использованием набора QIAquick Viral RNA Kit (QIAGEN, Valencia, Ca).
Обратную транскрипцию проводили следующим образом: смесь, содержащую 1 мкл праймера 5'-GACTAATACGACTCACTATAGGGAGCAAAAGCAGG-3' (20 мкМ), 9 мкл РНК (из экстрагированного объема 80 мкл) и 2 мкл dNTP (10 мМ) нагревали 10 мин при 65°C, охлаждали во льду. Добавляли 4 мкл 5х кДНК буфера, 1 мкл 0.1 М DTT, 1 мкл РНКзина (40 ед), 1 мкл воды, 1 мкл обратной транскриптазы ThermoScript (Invitrogen, Carlsbad, CA). Смесь центрифугировали и инкубировали 55°C - 5 мин, 60°C - 55 мин.
Пример 3. Получение флуоресцентных ампликонов гена нейраминидазы для анализа
Для амплификации фрагментов гена нейраминидазы использовали следующие пары праймеров - внешний и внутренний:
NA_F1 5'-CTATAGGGAGCAAAAGCAGGAGT-3' и
NA_R1 5'-TAACAGGA(G/A)CA(C/T)TCCTC(A/G)TA(G/A)TG-3';
NA_F2 5'-CA(C/T)TA(C/T)GAGGA(G/A)TG(C/T)TCCTGTTA-3' и
NA_R2 5'-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3'.
Длина нарабатываемых ампликонов составляла ~830 и ~600 пар нуклеотидов, соответственно. Для получения преимущественно одноцепочечной ДНК проводили ПЦР в асимметричном режиме. Состав реакционной смеси (50 мкл): буфер для ПЦР «Qiagen» (1.5 мМ MgCl2, KCl, (NH4)2SO4, Трис-HCl, pH 8.7), MgCl2 - 1.5 мМ, 200 нМ dATP, dCTP dGTP, 70 нМ dTTP, 100 нМ dUTP-Су3, 80 нМ прямого праймера, 1 мкМ обратного праймера, 1.25 ед. Hot Start Taq-полимеразы («Qiagen») и 2 мкл раствора ДНК. ПЦР проводили в термоциклере iCycler («BioRad»), используя следующий протокол: 95°C - 15 мин, затем 35 циклов 94°C - 30 сек, 52°C - 55°C - 30 сек, 72°C - 1 мин 10 сек. Контроль наработки ампликона осуществляли электрофорезом в 1% агарозном геле с окрашиванием этидий бромидом. Флуоресцентно-меченые ампликоны очищали от избытка меченых трифосфатов на гель-фильтрующих колонках «Qiagen», высушивали досуха на вакуумной центрифуге при 60° и использовали для гибридизации.
Пример 4. Получение флуоресцентных ампликонов гена гемагглютинина для анализа
Для амплификации фрагментов гена гемагглютинина использовали следующие пары праймеров - внешний и внутренний:
HA_F1: 5'-GACTCACTATAGGGAGCAAAAGCAGGGG-3'
HA_R1: 5'-59-TCWATRAANCCNGCDATDGCHCC-3'
HA_F2: 5'-GGDGCHATHGCNGGNTTYATWGA-3'
HA_R1: 5'-GGTGACACTATAGAAGTAGAAACAAGGGTGTTTT-3'.
Протяженность ампликонов составляла 600 и 800 нт соответственно.. Для получения преимущественно одноцепочечной ДНК проводили ПЦР в асимметричном режиме. Состав реакционной смеси (50 мкл): буфер для ПЦР «Qiagen» (1.5 мМ MgCl2, KCl, (NH4)2SO4, Трис-HCl, pH 8.7), MgCl2 - 1.5 мМ, 200 нМ dATP, dCTP dGTP, 70 нМ dTTP, 100 нМ dUTP-Су3, 80 нМ прямого праймера, 1 мкМ обратного праймера, 1.25 ед. Hot Start Taq-полимеразы («Qiagen») и 2 мкл раствора ДНК. ПЦР проводили в термоциклере iCycler («BioRad»), используя следующий протокол: 95°C - 15 мин, затем 35 циклов 94°C - 30 сек, 52°C - 55°C - 30 сек, 72°C - 1 мин 10 сек. Контроль наработки ампликона осуществляли электрофорезом в 1% агарозном геле с окрашиванием этидий бромидом. Флуоресцентно-меченые ампликоны очищали от избытка меченых трифосфатов на гель-фильтрующих колонках «Qiagen», высушивали досуха на вакуумной центрифуге при 60° и использовали для гибридизации.
Пример 5. Гибридизация анализируемых ампликонов
Высушенный образец ампликона растворяли в 10 мкл воды, добавляли 10 мкл 2х гибридизационного буфера (1х гибридизационный буфер: 6х SSC, 5х раствор Денхардта, 0.1% Tween 20) и прогревали перед нанесением на слайд 2 мин при 97°C, охлаждали во льду. Гибридизацию проводили в гибридизационной камере фирмы ArrayIt, Sunnyvale при температуре 55° - 60°C в течение 2 час. Затем слайд последовательно отмывали в растворах 6х SSC, 2х SSC + 0,1% SDS, 2х SSC и 1х SSC, центрифугировали и сканировали на сканере Scan Array Express 2.0 («Perkin Elmer») при длинах волны возбуждающего лазера λ=543 нм и λ=633 нм. Изображение анализировали с использованием программы ScanArray Express, входящих в математическое обеспечение сканера.
Пример 6. Анализ результатов гибридизации
Сканирование слайда проводили на сканере ScanArray Express 2.0 (Perkin Elmer) последовательно с использованием двух возбуждающих лазеров с длинами волн λ1=543 нм для Су3 и λ2=633 нм для Су5. Изображение, полученное по каналу возбуждения флуоресценции с длиной волны λ2, служило показателем качества печати чипа, а интегральная величина флуоресценции каждого спота рассматривалась в качестве нормировочной для флуоресценции того же спота, измеренной по каналу возбуждения с λ1. Анализ изображения осуществляли с использованием программы Scanarray Express, входящих в математическое обеспечение сканера. Для i-го спота нормированную флуоресценцию рассчитывали по формуле:
Ii=Ii(Су3)/Ii(Cy5),
где Ii(Су3)=Fi(Су3)-Bi(Су3), Fi(Су3) - интегральная интенсивность флуоресценции в пределах спота, Bi(Су3) - средняя фоновая интенсивность флуоресценции в окружении спота. Аналогичным образом рассчитывали величину Ii(Су5).
Для анализа результатов гибридизации рассчитывали среднюю флуоресценцию спотов по формуле:
,
где µ - подтип нейраминидазы,
i - номер спота,
N - число спотов на чипе для используемых для определения подтипа нейраминидазы.
Субтип определяли по столбцу гистограммы с максимальным значением средней флуоресценции спотов. Указанием на однозначность отнесения является отсутствие перекрывания интервалов ошибок субтипов. Интервал ошибок для каждого из субтипов рассчитывался по формуле
Sµ=SxYµ,
где Sx - относительная ошибка флуоресценции.
Относительная ошибка измерения значения флуоресценции определялась исходя из вариации в интенсивности флуоресценции маркерных спотов субэррея (xi, расположены в левой и правой колонках микрочипа).
Sx=σ/Хср, где
xi - флуоресценция i-го маркерного спота;
n - число маркерных спотов.
Эта ошибка является интегральной и включает в себя все ошибки, связанные с приготовлением раствора олигонуклеотида для печати и печатью микрочипа.
Результаты анализа представляются в графической форме с помощью программы Microsoft Office Excel.
Результаты анализа вируса гриппа A, содержащих различные субтипы гена нейраминидазы, приведены на Фиг.7.
Результаты анализа вируса гриппа A, содержащих различные субтипы гена гемагглютинина, приведены на Фиг.9.
Во всех проанализированных случаях типирование генов гемагглютинина и нейраминидазы вируса гриппа A с помощью заявляемого микрочипа осуществлялось корректно.
1. Способ выбора ДНК-зондов для микрочиповой диагностики высоковариабельных геномов, отличающийся тем, что выбор таких ДНК-зондов осуществляется на основе анализа белковых последовательностей и проводится в два этапа:а) на первом этапе проводят анализ аминокислотных последовательностей белков микроорганизма, выявляют консервативные олигопептиды и определяют внутри них короткие дискриминирующие микроорганизм пептиды;б) с помощью генетического кода определяют структуру ДНК-зондов, кодирующих дискриминирующие микроорганизм пептиды.
2. Биочип для типирования гена нейраминидазы вируса гриппа А, содержащий типирующие гибридизационные зонды, ковалентно присоединенные непосредственно к стеклянной микроматрице, имеющие структуру дискриминирующих ДНК-полинуклеотидов, приведенных в Таблице 3.
3. Способ типирования гена нейраминидазы вируса гриппа А, включающий выделение суммарной РНК из природного образца, проведение ОТ-ПЦР для амплификации сегмента генома вируса гриппа, кодирующего ген нейраминидазы в асимметричном режиме с использованием специфичных олигонуклеотидов-праймеров для получения флуоресцентно меченых преимущественно одноцепочечных ДНК, проведение ДНК-ДНК гибридизации полученных ампликонов с дискриминирующими зондами, входящими в состав биочипа, регистрацию флуоресцентного сигнала меченого образца с помощью регистрирующего устройства и интерпретацию полученных результатов, отличающийся тем, что при амплификации гена нейроминидазы используют набор олигонуклеотидов-праймеровNA-F1 5'-CTATAGGGAGCAAAAGCAGGAGT-3' иNA_R1 5'-TAACAGGA(G/A)CA(C/T)TCCTC(A/G)TA(G/A)TG-3';NA _F2 5'-CA(C/T)TA(C/T)GAGGA(G/A)TG(C/T)TCCTGTTA-3' иNA-R2 5'-CACTATAGAAGTAGAAACAAGGAGTTTTTT-3'), в качестве биочипа - микроматрицу, охарактеризованную в п.2, в качестве регистрирующего устройства - флуоресцентный сканер, а интерпретацию полученных результатов проводят с помощью программы Scanarray Express.