Улавливание и характеристика совместно локализованного хроматина
Иллюстрации
Показать всеИзобретение относится к области биотехнологии, в частности к способу анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими представляющими интерес нуклеотидными последовательностями. Способ предусматривает (а) получение образца поперечно сшитой ДНК и (b) расщепление поперечно сшитой ДНК первым ферментом рестрикции. Далее проводят (с) лигирование поперечно сшитых нуклеотидных последовательностей, (d) удаление поперечных сшивок; (е) расщепление нуклеотидных последовательностей вторым ферментом рестрикции (f) лигирование одной или нескольких последовательностей ДНК, имеющих известный состав нуклеотидов, с доступным сайтом(ами) расщепления вторым ферментом рестрикции, который фланкирует одну или несколько представляющих интерес нуклеотидных последовательностей. Проводят (g) амплификацию одной или нескольких представляющих интерес нуклеотидных последовательностей с использованием по меньшей мере двух олигонуклеотидных праймеров, причем каждый праймер гибридизуется с последовательностями ДНК, которые фланкируют представляющие интерес нуклеотидные последовательности. Далее осуществляют (h) гибридизацию амплифицированной последовательности(ей) с чипом; и (i) определения частоты взаимодействия между последовательностями ДНК. 9 н. и 20 з.п. ф-лы, 19 ил., 2 табл., 8 пр.
Реферат
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к анализу частоты взаимодействий двух или более нуклеотидных последовательностей в ядерном пространстве.
УРОВЕНЬ ТЕХНИКИ
Целью исследований архитектуры ядра клеток млекопитающих является понимание того, как ДНК длиной 2 метра укладывается в ядре размером 10 мкм, обеспечивая при этом точную экспрессию генов, которые определяют тип клетки, и каким образом она точно реплицируется во время каждого клеточного цикла. Значительный прогресс в данной области был достигнут в результате микроскопических исследований, которые выявили, что геномы распределены в ядерном пространстве не случайно. Например, плотно упакованный гетерохроматин расположен отдельно от более открытого эухроматина, и хромосомы занимают разные территории в ядерном пространстве. Существует сложная взаимосвязь между положением в ядре и транскрипционной активностью. Хотя транскрипция происходит во всем внутреннем пространстве ядра, активные гены, которые образуют кластеры на хромосомах, преимущественно локализованы на краю или на внешней стороне территории своей хромосомы. Отдельные гены могут перемещаться при изменениях их транскрипционного состояния, как было измерено относительно сравнительно крупных ориентиров в ядре, таких как территории хромосом, центромеры или периферия ядра. Кроме того, активно транскрибируемые гены, находящиеся на расстоянии десятков миллионов оснований друг от друга на хромосоме, могут находиться рядом в ядре, как было недавно показано посредством флуоресцентной гибридизации in situ (FISH) для локуса β-глобина и нескольких других выбранных генов. Кроме транскрипции геномная организация связана с координацией репликации, рекомбинации и вероятности транслокации локусов (что может приводить к злокачественным перерождениям) и установкой и переустановкой эпигенетических программ. На основании указанных наблюдений полагают, что архитектурная организация ДНК в клеточном ядре вносит основной вклад в функционирование генома.
Разработаны различные анализы, позволяющие проникнуть в пространственную организацию геномных локусов in vivo. Разработан один анализ, называемый «RNA-TRAP» (Carter et al. (2002) Nat. Genet. 32, 623), который заключается в направлении пероксидазы хрена (HRP) к мишени - образующимся РНК-транскриптам с последующим количественным анализом катализируемого HRP отложения биотина в области хроматина.
Другой анализ, который был разработан, назван способом улавливания конформации хромосом (chromosome conformation capture, 3C), которая является средством исследования структурной организации области генома. Способ 3C заключается в количественном ПЦР-анализе частот поперечного связывания между двумя данными рестрикционными фрагментами ДНК, что является мерой их близости в ядерном пространстве (см. фиг.1). Исходно разработанный для анализа конформации хромосом дрожжей (Dekker et al., 2002), указанный способ был адаптирован для исследования взаимосвязи между экспрессией генов и укладкой хроматина в сложных кластерах генов млекопитающих (см., например, Tolhuis et al., 2002; Palstra et al., 2003; and Drissen et al., 2004). Коротко, способ 3C заключается в поперечном сшивании клеток формальдегидом in vivo и расщеплении хроматина в ядре ферментом рестрикции с последующим лигированием фрагментов ДНК, которые были поперечно сшиты в один комплекс. Затем продукты лигирования количественно оценивают в ПЦР. Стадия ПЦР-амплификации требует наличия информации о последовательности для каждого из фрагментов ДНК, которые необходимо амплифицировать. Таким образом, способ 3C обеспечивает меру частоты взаимодействия между выбранными фрагментами ДНК.
Существует большая потребность в высокопроизводительном способе, посредством которого можно систематически проводить объективный скрининг целого генома в отношении локусов ДНК, которые контактируют друг с другом в ядерном пространстве.
В настоящем изобретении предпринята попытка усовершенствовать способ 3C.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Способ 3C, который применяют в настоящее время, дает возможность анализировать только ограниченное количество выбранных взаимодействий ДНК-ДНК вследствие ограничений стадии ПЦР-амплификации, которая требует знания информации о конкретной последовательности для каждого анализируемого фрагмента. Кроме того, отбор фрагментов рестрикции в качестве кандидатов для взаимодействий ДНК дальнего действия требует значительного количества предварительных сведений (например, положение гиперчувствительных сайтов) о представляющем интерес локусе, которые обычно не доступны. Учитывая функциональную значимость многих ДНК-ДНК-взаимодействий дальнего действия, описанных до настоящего времени, возможность случайным образом проводить скрининг элементов ДНК, которые за счет образования петли приближаются к представляющей интерес последовательности - такой как генный промотор, энхансер, инсулятор, сайленсер, начало репликации или MAR/SAR - или к представляющей интерес области генома - такой как область с высокой плотностью генов или область с низкой плотностью генов или повторяющийся элемент - может значительно облегчить картирование последовательностей в регуляторной сети.
Настоящее изобретение относится к способу 4C [т.е. улавливанию и характеристике совместно локализованного хроматина «capture and characterise co-localised chromatin), который обеспечивает высокопроизводительный анализ частоты взаимодействий двух или более нуклеотидных последовательностей в ядерном пространстве.
Способ 4C (улавливание и характеристика совместно локализованного хроматина) является модифицированным вариантом способа 3C, который обеспечивает возможность объективного широкого поиска в геноме фрагментов ДНК, которые взаимодействуют с выбранным локусом. Коротко, анализ 3C осуществляют как обычно, но минуя стадию ПЦР. Матрица 3C содержит «приманку» (например, выбранный фрагмент рестрикции, который охватывает представляющий интерес ген), лигированную со многими разными представляющими интерес нуклеотидными последовательностями (представляющими собой геномное окружение данного гена). Матрицу расщепляют другим, вторым ферментом рестрикции и лигируют. Преимущественно амплифицируют одну или несколько представляющих интерес нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью, используя по меньшей мере один (предпочтительно по меньшей мере два) олигонуклеотидный праймер, при этом по меньшей мере один праймер гибридизуется с последовательностью ДНК, которая фланкирует представляющие интерес нуклеотидные последовательности. Обычно это дает картину фрагментов ПЦР, которая является высоко воспроизводимой в независимых реакциях амплификации и специфичной для данной ткани. В одном варианте используют HindIII и DpnII в качестве первого и второго ферментов рестрикции. Затем амплифицированные фрагменты можно пометить и необязательно гибридизовать с чипом, обычно против контрольного образца, содержащего геномную ДНК, расщепленную такой же комбинацией ферментов рестрикции.
В одном предпочтительном варианте осуществления настоящего изобретения лигированные фрагменты, которые расщепляют вторым ферментом рестрикции, затем повторно лигируют с образованием небольших колец ДНК.
Следовательно, способ 3C был модифицирован так, чтобы амплифицировать все представляющие интерес нуклеотидные последовательности, которые взаимодействуют с нуклеотидной последовательностью-мишенью. На практике это означает, что вместо осуществления реакции амплификации с праймерами, которые являются специфичными по отношению к фрагментам, которые хотят проанализировать, амплификацию осуществляют, используя олигонуклеотидный праймер(ры), который гибридизуется с последовательностью ДНК, которая фланкирует представляющие интерес нуклеотидные последовательности. Преимуществом является то, что 4C не смещен в сторону конструирования праймеров ПЦР, которые включены в стадию ПЦР-амплификации и поэтому могут быть использованы для поиска во всем геноме взаимодействующих элементов ДНК.
СУЩНОСТЬ АСПЕКТОВ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
Аспекты настоящего изобретения представлены в прилагаемой формуле изобретения.
В первом аспекте предлагается способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими представляющими интерес нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий стадии (a) получения образца поперечно сшитой ДНК; (b) расщепления поперечно сшитой ДНК первым ферментом рестрикции; (c) лигирования поперечно сшитых нуклеотидных последовательностей; (d) удаления поперечных сшивок; (e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции; (f) лигирования одной или нескольких последовательностей ДНК, имеющих известный состав нуклеотидов, с доступным сайтом(ами) расщепления вторым ферментом рестрикции, который фланкирует одну или несколько представляющих интерес нуклеотидных последовательностей; (g) амплификации одной или нескольких представляющих интерес нуклеотидных последовательностей с использованием по меньшей мере двух олигонуклеотидных праймеров, причем каждый праймер гибридизуется с последовательностями ДНК, которые фланкируют представляющие интерес нуклеотидные последовательности; (h) гибридизации амплифицированной последовательности(ей) с чипом; и (i) определения частоты взаимодействия между последовательностями ДНК.
Во втором аспекте предлагается способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий стадии (a) получения образца поперечно сшитой ДНК; (b) расщепления поперечно сшитой ДНК первым ферментом рестрикции; (c) лигирования поперечно сшитых нуклеотидных последовательностей; (d) удаления поперечных сшивок; (e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции; (f) образования кольцевых нуклеотидных последовательностей; (g) амплификации одной или нескольких нуклеотидных последовательностей, которые лигированы с нуклеотидной последовательностью-мишенью; (h) необязательно гибридизации амплифицированных последовательностей с чипом; и (i) определения частоты взаимодействия между последовательностями ДНК.
В третьем аспекте предлагается кольцевая нуклеотидная последовательность, содержащая первую и вторую нуклеотидные последовательности, в которой концы первой и второй нуклеотидных последовательностей разделены разными сайтами узнавания ферментами рестрикции и в которой указанная первая нуклеотидная последовательность является нуклеотидной последовательностью-мишенью, а указанная вторая нуклеотидная последовательность может быть получена поперечным сшиванием геномной ДНК.
В четвертом аспекте предлагается способ получения кольцевой нуклеотидной последовательности, включающий стадии (a) получения образца поперечно сшитой ДНК; (b) расщепления поперечно сшитой ДНК первым ферментом рестрикции; (c) лигирования поперечно сшитых нуклеотидных последовательностей; (d) удаления поперечных сшивок; (e) расщепления нуклеотидных последовательностей вторым ферментом рестрикции; и (f) образования кольцевых нуклеотидных последовательностей.
В пятом аспекте предлагается способ анализа частоты взаимодействия нуклеотидной последовательности-мишени с одной или несколькими нуклеотидными последовательностями (например, одним или несколькими геномными локусами), включающий применение кольцевой нуклеотидной последовательности.
В шестом аспекте предлагается чип с зондами, иммобилизованными на подложке, содержащий один или несколько зондов, которые гибридизуются или способны гибридизоваться с кольцевой нуклеотидной последовательностью.
В седьмом аспекте предлагается набор зондов с последовательностями, комплементарными последовательностям нуклеиновой кислоты, граничащей с каждым из первых сайтов узнавания рестрикционными ферментами первого фермента рестрикции в геномной ДНК.
В восьмом аспекте предлагается способ получения набора зондов, включающий стадии (a) идентификации каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК; (b) конструирования зондов, которые способны гибридизоваться с последовательностями, граничащими с каждым из первых сайтов узнавания ферментом рестрикции в геномной ДНК; (c) синтеза зондов; и (d) объединения зондов с образованием набора зондов или по существу набора зондов.
В девятом аспекте предлагается набор зондов или по существу набор зондов, который получен или может быть получен способом, описанным в данной публикации.
В десятом аспекте предлагается чип, содержащий матрицу зондов, или по существу набор зондов, описанных в данной публикации.
В одиннадцатом аспекте предлагается чип, содержащий набор зондов согласно настоящему описанию.
В двенадцатом аспекте предлагается способ получения чипа, включающий в себя стадию иммобилизации на твердой подложке по существу матрицы зондов или по существу набора зондов, описанных в данной публикации.
В тринадцатом аспекте предлагается способ получения чипа, включающий в себя стадию иммобилизации на твердой подложке матрицы зондов или набора зондов, описанных в данной публикации.
В четырнадцатом аспекте предлагается чип, который получен или может быть получен способом, описанным в данной публикации.
В пятнадцатом аспекте предлагается способ идентификации одного или нескольких ДНК-ДНК-взаимодействий, которые являются показателем конкретного патологического состояния, включающий в себя стадию осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом на стадии (a) образец поперечно сшитой ДНК получают из пораженной заболеванием и не пораженной заболеванием клетки, и при этом разница между частотой взаимодействия между последовательностями ДНК из пораженной заболеванием клетки и не пораженной заболеванием клетки свидетельствует о том, что ДНК-ДНК-взаимодействие является показателем конкретного патологического состояния.
В шестнадцатом аспекте предлагается способ диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия, включающий в себя стадию осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом стадия (a) заключается в получении образца поперечно сшитой ДНК от субъекта; и стадия (i) заключается в сравнении частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в непораженном контроле; при этом различие между значением, полученным для контроля, и значением, полученным для субъекта, является показателем того, что субъект имеет данное заболевание или синдром, или является показателем того, что субъект будет иметь данное заболевание или синдром.
В семнадцатом аспекте предлагается способ диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия, включающий в себя стадию осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом стадия (a) заключается в получении образца поперечно сшитой ДНК от субъекта; и при этом указанный способ включает в себя дополнительную стадию (j) идентификации одного или нескольких локусов, которые были подвергнуты перестройке в геноме, которая связана с заболеванием.
В восемнадцатом аспекте предлагается способ анализа для идентификации одного или нескольких агентов, которые модулируют ДНК-ДНК-взаимодействие, включающий в себя стадии (a) контактирования образца с одним или несколькими агентами; и (b) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения, при этом стадия (a) заключается в получении поперечно сшитой ДНК из образца; при этом разница между (i) частотой взаимодействия между последовательностями ДНК в присутствии агента и (ii) частотой взаимодействия между последовательностями ДНК в отсутствие агента являются показателем того, что агент модулирует ДНК-ДНК-взаимодействие.
В девятнадцатом аспекте предлагается способ определения положения сбалансированного и/или несбалансированного точечного разрыва (например, в случае транслокации), включающий в себя стадию (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; и (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом переход от низкой к высокой частоте ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем положения точки разрыва.
В двадцатом аспекте предлагается способ определения положения сбалансированной и/или несбалансированной инверсии, включающий в себя стадии (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; и (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом обратная картина частот ДНК-ДНК-взаимодействий для образца по сравнению с контролем является показателем инверсии.
В двадцать первом аспекте предлагается способ определения положения делеции, включающий в себя стадии (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом снижение частоты ДНК-ДНК-взаимодействия в образце по сравнению с контролем является показателем делеции.
В двадцать втором аспекте предлагается способ определения положения дупликации, включающий в себя стадии (a) осуществления стадий (a)-(i) согласно первому и второму аспектам настоящего изобретения; и (b) сравнения частоты взаимодействия между последовательностями ДНК с частотой взаимодействия в контроле; при этом увеличение или уменьшение частоты ДНК-ДНК-взаимодействия в случае образца от субъекта по сравнению с контролем является показателем дупликации или инсерции.
В двадцать третьем аспекте предлагается агент, который получен или может быть получен с применением способа анализа, описанного в данной публикации.
В двадцать четвертом аспекте предлагается применение кольцевой нуклеотидной последовательности для идентификация одного или нескольких ДНК-ДНК-взаимодействий в образце.
В двадцать пятом аспекте предлагается применение кольцевой нуклеотидной последовательности для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
В двадцать шестом аспекте предлагается применение матрицы зондов или набора зондов, описанных в данной публикации, для идентификации одного или нескольких ДНК-ДНК-взаимодействий в образце.
В двадцать седьмом аспекте предлагается применение матрицы зондов или набора зондов, описанных в данной публикации, для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
В двадцать восьмом аспекте предлагается применение чипа, описанного в данной публикации, для идентификация одного или нескольких ДНК-ДНК-взаимодействий в образце.
В двадцать девятом аспекте предлагается применение чипа, описанного в данной публикации, для диагностики или прогнозирования заболевания или синдрома, вызванного или связанного с изменением ДНК-ДНК-взаимодействия.
В тридцатом аспекте предлагается способ, матрица зондов, набор зондов, способ, чип, способ анализа, агент или применение, которые по существу описаны в данной публикации со ссылками на примеры и фигуры.
ПРЕДПОЧТИТЕЛЬНЫЕ ВАРИАНТЫ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Предпочтительно реакция лигирования на стадии (f) приводит к образованию колец ДНК.
Предпочтительно нуклеотидная последовательность-мишень выбрана из группы, состоящей из геномной перестройки, промотора, энхансера, сайленсера, инсулятора, связанной с матриксом области, области регуляции локуса, транскрипционной единицы, начала репликации, горячей точки рекомбинации, точки разрыва при транслокации, центромеры, теломеры, области с высокой плотностью генов, области с низкой плотностью генов, повторяющегося элемента и сайта интеграции (вируса).
Предпочтительно нуклеотидной последовательностью-мишенью является нуклеотидная последовательность, которая связана или вызывает заболевание, или расположена на расстоянии вплоть до 15 млн.п.о. или более на линейной ДНК-матрице от локуса, который ассоциирован с заболеванием или вызывает заболевание.
Предпочтительно нуклеотидная последовательность-мишень выбрана из группы, состоящей из AML1, MLL, MYC, BCL, BCR, ABL1, IGH, LYL1, TAL1, TAL2, LMO2, TCRα/δ, TCRβ и HOX или других локусов, связанных с заболеванием, которые описаны в «Catalogue of Unbalanced Chromosome Aberrations in Man» 2nd edition. Albert Schinzel. Berlin: Walter de Gxuyter, 2001. ISBN.3-11-011607-3.
Предпочтительно первым ферментом рестрикции является фермент рестрикции, который узнает сайт узнавания, состоящий из 6-8 п.о.
Предпочтительно первый фермент рестрикции выбран из группы, состоящей из BglII, HindIII, EcoRI, BamHI, SpeI, PstI и NdeI.
Предпочтительно вторым ферментом рестрикции является фермент рестрикции, который узнает сайт узнавания нуклеотидной последовательности, состоящий из 4 или 5 п.о.
Предпочтительно второй сайт узнавания ферментом рестрикции расположен на расстоянии более чем примерно 350 п.о. от первого сайта рестрикции в нуклеотидной последовательности-мишени.
Предпочтительно нуклеотидная последовательность является меченой.
Предпочтительно последовательности зондов комплементарны последовательности нуклеиновой кислоты, граничащей с каждой стороны с каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно последовательности зондов комплементарны последовательности нуклеиновой кислоты, которая расположена в пределах менее чем 300 пар оснований от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды комплементарны последовательности, которая расположена в пределах менее чем 300 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды комплементарны последовательности, которая расположена в пределах от 200 до 300 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды комплементарны последовательности, которая расположена в пределах от 100 до 200 п.о. или от 0 до 100 п.о. от каждого из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно два или более зондов способны гибридизоваться с последовательностью, граничащей с каждым первым сайтом узнавания рестрикционными ферментами для первого фермента рестрикции в геномной ДНК.
Предпочтительно зонды перекрываются или частично перекрываются.
Предпочтительно перекрывание составляет менее 10 нуклеотидов.
Предпочтительно последовательность зонда соответствует всей или части последовательности между каждым из первых сайтов узнавания рестрикционными ферментами для первого фермента рестрикции и каждым из первых ближайших вторых сайтов узнавания рестрикционными ферментами для второго фермента рестрикции.
Предпочтительно каждый зонд является по меньшей мере 25-мером.
Предпочтительно каждый зонд является 25-60-мером.
Предпочтительно зонды являются продуктами ПЦР-амплификации.
Предпочтительно чип содержит примерно 300000-400000 зондов.
Предпочтительно чип содержит примерно 385000 или более зондов, предпочтительно примерно 750000 зондов, более предпочтительно 6×750000 зондов.
Предпочтительно чип содержит или представляет полный геном данного вида при более низком разрешении.
Предпочтительно чип содержит один из каждых 2, 3, 4, 5, 6, 7, 8, 9 или 10 зондов, которые располагаются по порядку на линейной хромосомной матрице.
Предпочтительно переход от более низкой к более высокой частоте взаимодействия является показателем положения сбалансированного и/или несбалансированного точечного разрыва.
Предпочтительно обратная картина частоты ДНК-ДНК-взаимодействия в образце от субъекта по сравнению с контролем является показателем сбалансированной и/или несбалансированной инверсии.
Предпочтительно уменьшение частоты ДНК-ДНК-взаимодействия в случае образца от субъекта по сравнению с контролем в сочетании с увеличением частоты ДНК-ДНК-взаимодействия для более отдаленных областей является показателем сбалансированной и/или несбалансированной делеции.
Предпочтительно увеличение или уменьшение частоты ДНК-ДНК-взаимодействия в случае образца от субъекта по сравнению с контролем является показателем сбалансированной и/или несбалансированной дупликации или инсерции.
Предпочтительно перед осуществлением указанного способа используют спектральное кариотипирование и/или FISH.
Предпочтительно заболевание является наследственным заболеванием.
Предпочтительно заболевание представляет собой злокачественную опухоль.
Предпочтительно две или более амплифицированных последовательностей метят по-разному.
Предпочтительно две или более амплифицированных последовательностей метят идентично в том случае, когда последовательности находятся на разных хромосомах.
Предпочтительно две или более амплифицированных последовательностей метят идентично в том случае, когда последовательности находятся на одной и той же хромосоме на расстоянии, которое является достаточно большим, чтобы было минимальное перекрывание между сигналами ДНК-ДНК-взаимодействия.
Предпочтительно диагностика или прогноз являются пренатальной диагностикой или прогнозом.
ПРЕИМУЩЕСТВА
Настоящее изобретение имеет ряд преимуществ. Указанные преимущества будут понятны из следующего описания.
В качестве примера настоящее изобретение имеет преимущество, так как помимо прочего относится к коммерчески применимым нуклеотидным последовательностям, способам, зондам и чипам.
В качестве следующего примера настоящее изобретение имеет преимущество, так как оно относится к высокопроизводительному анализу частоты взаимодействия двух или более нуклеотидных последовательностей в ядерном пространстве.
В качестве следующего примера настоящее изобретение имеет преимущество, так как при использовании обычного способа 3C каждое отдельное ДНК-ДНК-взаимодействие необходимо анализировать в отдельной особой реакции ПЦР с использованием уникальной пары праймеров. Поэтому высокопроизводительный анализ возможен только в том случае, если ПЦР автоматизирована, но расходы на такое большое количество праймеров будут слишком велики. Соответственно высокопроизводительный (широко охватывающий геном) анализ ДНК-ДНК-взаимодействий не осуществим в случае обычного способа 3C. В отличие от этого, настоящее изобретение дает возможность одновременного скрининга тысяч ДНК-ДНК-взаимодействий. Высокопроизводительный анализ ДНК-ДНК-взаимодействий согласно настоящему изобретению существенно увеличит масштаб и разрешающую способность анализа.
В качестве следующего примера настоящее изобретение имеет преимущество, так как при использовании обычного способа 3C скрининг смещен к таким последовательностям ДНК, для которых сконструированы, упорядочены и включены в анализ олигонуклеотидные праймеры. Выбор таких олигонуклеотидных праймеров обычно основан на данных, касающихся, например, положения (отдаленных) энхансеров и/или других регуляторных элементов/гиперчувствительных сайтов, которые, как полагают, будут поперечно связываться с исследуемой нуклеотидной последовательностью. Таким образом, обычный 3C смещен в сторону конструирования ПЦР-праймеров, которые включают на стадии ПЦР-амплификации, тогда как 4C не смещен и может быть использован для поиска во всем геноме в отношении взаимодействующих элементов ДНК. Это связано с тем, что амплификация поперечно сшитых последовательностей в 4C не основана на рассчитанных данных о последовательностях, которые поперечно связываются с исследуемой нуклеотидной последовательностью. Точнее, в одном варианте осуществления 4C последовательности, которые перекрестно связываются с первой нуклеотидной последовательностью (мишенью), могут быть амплифицированы с использованием ПЦР-праймеров, которые гибридизуются с данной нуклеотидной последовательностью. Таким образом, настоящее изобретение обеспечивает объективный широко охватывающий геном скрининг ДНК-ДНК-взаимодействий.
В качестве следующего примера настоящее изобретение имеет преимущество, так как использование обычного способа 3C обеспечивает возможность только избирательной амплификации одного ДНК-ДНК-взаимодействия. Это не информативно при гибридизации с чипом. Способ был усовершенствован так, что все фрагменты, которые взаимодействуют с первой нуклеотидной последовательностью (мишенью) теперь амплифицируют, например, избирательно амплифицируют.
В качестве следующего примера настоящее изобретение имеет преимущество, так как способ 4C можно применять для выявления сбалансированных или несбалансированных генетических аберраций - таких как все типы транслокаций, делеций, инверсий, дупликаций и других генетических перестроек - в нуклеиновой кислоте, например, в хромосомах. Способом 4C (в котором измеряют близость фрагментов ДНК) даже можно определять предрасположенность субъекта к возникновению некоторых транслокаций, делеций, инверсий, дупликаций и других перестроек в геноме (например, сбалансированных или несбалансированных транслокаций, делеций, инверсий, дупликаций и других перестроек в геноме). Преимущество по сравнению с имеющимся в настоящее время способом заключается в том, что он не требует знания точного положения изменения, так как разрешающая способность способа 4C является такой, что его можно применять для выявления перестроек даже тогда, когда «4C-приманка» (которую определяют по сайтам узнавания первым и вторым ферментами рестрикции, которые анализируют) расположена далеко (например, до одного миллиона оснований или еще больше) от изменения. Другое преимущество заключается в том, что способ 4C позволяет точно картировать изменения, так как его можно использовать для определения двух (первых) сайтов рестрикции, между которыми происходит изменение. Другое преимущество состоит в том, что клетки не надо культивировать перед фиксацией. Таким образом, также можно анализировать, например, твердые опухоли в отношении геномных перестроек.
В качестве следующего примера настоящее изобретение имеет преимущество, так как способом 4C также можно выявлять изменения (например, перестройки) в предзлокачественном состоянии, т.е. до того, как все клетки будут содержать указанные изменения. Таким образом, способ можно применять не только для диагностики заболевания, но также для прогнозирования заболевания.
В качестве следующего примера конструкция чипа согласно настоящему изобретению особенно предпочтительна по сравнению с существующими мозаичными геномными чипами - такими как мозаичные геномные чипы Nimblegen - так как конструкция позволяет представлять намного большую часть генома на одном чипе. В качестве примера в случае узнавания ферментом рестрикции гекса-нуклеотидной последовательности будет достаточно 3 чипа примерно с 385000 зондами каждый, чтобы охватить, например, полный геном человека или мыши. В случае узнавания ферментом рестрикции более чем 6 п.о. можно использовать один чип примерно с 385000 зондами, чтобы охватить, например, полный геном человека или мыши. Преимущества конструкции чипа заключаются в том, что (1) каждый зонд является информативным, так как анализирует независимое событие лигирования, что значительно облегчает интерпретацию результатов; и (2) можно осуществить большое представление генома в виде пятен на одном чипе, что является эффективным с точки зрения затрат.
Способ 4C предпочтительно можно использовать для тонкого картирования плохо охарактеризованных перестроек, исходно выявленных цитогенетическими способами (световая микроскопия, FISH, SKY и т.д.).
Способ 4C предпочтительно можно использовать для одновременного скрининга на одном чипе в отношении комбинаций перестроек, которые произошли вблизи нескольких локусов.
КРАТКОЕ ОПИСАНИЕ ФИГУР
Фиг.1
Принцип способа 3C
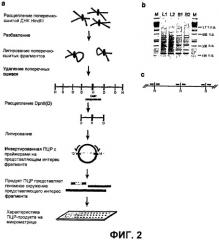
Фиг.2
(a) Принцип варианта способа 4C. Анализ 3C осуществляют как обычно, например, используя HindIII (H) в качестве фермента рестрикции. После удаления поперечных сшивок смесь ДНК будет содержать первую нуклеотидную последовательность (мишень), лигированную со многими разными фрагментами. Указанные фрагменты будут амплифицированы и помечены с использованием способов амплификации - таких как инвертированная ПЦР - например, на DpnII-кольцах, используя праймеры, специфичные для первой нуклеотидной последовательности (мишени). Меченые продукты амплификации могут быть гибридизованы с чипами, как описано в данной публикации. В качестве примеров приведены HindIII и DpnII, но также могут быть использованы другие комбинации ферментов рестрикции - например, рестриктазы, узнающие 6 или 8 нуклеотидов и рестриктазы, узнающие 4 или 5 нуклеотидов, (b) результаты ПЦР после разделения продуктов гель-электрофорезом из двух независимых образцов фетальной печени (L1, L2) и головного мозга (B1, B2), (c) схематичное представление положения зондов на микроматрице. Зонды конструировали в пределах 100 п.о. HindIII-сайтов. Таким образом, каждый зонд позволяет проанализировать одного возможного партнера при лигировании.
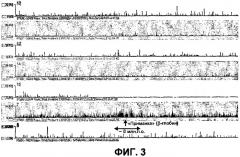
Фиг.3
Способ 4C выявляет геномное окружение гена β-глобина (хромосома 7). Показаны необработанные отношения (сигналы 4C для HS2-β-глобина, деленные на сигналы, полученные для контрольного образца) для зондов, локализованных в областях генома размером ~35 млн.п.о. в хромосомах мыши 10, 11, 12, 14, 15, 7 и 8 (сверху вниз; показанные области находятся на одинаковом расстоянии от каждой соответствующей центромеры). Следует обратить внимание на большой кластер сильных сигналов вокруг «приманки» (глобина) в хромосоме 7 (ряд 6), который показывает, что способ 4C выявляет геномные фрагменты, находящиеся вблизи линейной хромосомной матрицы (в соответствии с тем фактом, что частоты взаимодействия обратно пропорциональны расстоянию между сайтами в геноме). Следует обратить внимание, что область, связанная в цис-положении вокруг «приманки», где наблюдаются высокие интенсивности сигнала, является большой (>5 млн.п.о.), свидетельствуя, например, что транслокации могут быть выявлены даже в случае «приманок», находящихся на расстоянии более 1 млн.п.о. от точки разрыва.
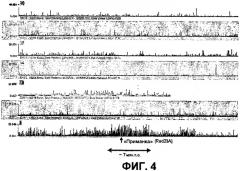
Фиг.4
Способ 4C выявляет геномное окружение Rad23A (хромосома 8). Показаны необработанные отношения (сигналы 4C для Rad23A, деленные на сигнал, полученный для контрольного образца) для зондов, локализованных в геномных областях ~15 млн.п.о. или более в хромосомах мыши 10, 11, 12, 14, 15, 7 и 8 (сверху вниз; показанные области находятся на одинаковом расстоянии от каждой соответствующей центромеры). Следует обратить внимание на большой кластер сильных сигналов вокруг «приманки» (Rad23A) в хромосоме 8 (ряд 7), который показывает, что способ 4C выявляет геномные фрагменты, находящиеся вблизи линейной хромосомной матрицы (в соответствии с тем фактом, что частоты взаимодействия обратно пропорциональны расстоянию между сайтами в геноме). Следует обратить внимание, что область, связанная в цис-положении вокруг «приманки», где наблюдаются высокие интенсивности сигнала, является большой (>5 млн.п.о.), свидетельствуя, например, что транслокации могут быть выявлены даже в случае «приманок», находящихся на расстоянии более 1 млн.п.о. от точки разрыва.
Фиг.5
4C-взаимодействия β-глобина в хромосоме 7 (~135 млн.п.о.) д