Способ выявления устойчивых к рифампицину изолятов mycobacterium tuberculosis
Иллюстрации
Показать всеИзобретение относится к области молекулярной биологии и медицины. Предложен способ выявления мутаций гена rpoB посредством проведения ПЦР, где в режиме «реального времени» нарабатывается фрагмент гена rpoB с использованием праймеров Rpo11 (5'-GACGTGGAGGCGATCACAC-3') и Rpo12 (5'-GGCACGCTCACTTGACAGAC-3'), после чего проводится HRM-анализ. Для увеличения надежности HRM-анализа мутаций в rpoB гене смешивают равное количество тестируемой ДНК и ДНК H37Rv. Проводят уравнивание концентрации с помощью количественной ПЦР с использованием той же пары праймеров с построением калибровочной кривой и использованием регрессионного уравнения. Способ благодаря высокой специфичности и чувствительности позволяет надежно и быстро определить тип мутаций в гене rpoB, ассоциированных с возникновением устойчивости к рифампицину. 2 ил.
Реферат
Предлагаемое изобретение относится к молекулярной биологии и медицине и может быть использовано в лабораторной диагностике туберкулеза для быстрого выявления мутаций в гене rpoB, ассоциированных с формированием устойчивости к рифампицину.
Антибиотикоустойчивость у микобактерий туберкулезного комплекса - достаточно хорошо изученное явление с точки зрения молекулярно-генетических механизмов. Описаны мутации, ассоциированные с устойчивостью к изониазиду, этамбутолу, пиразинамиду, рифампицину и другим противотуберкулезным препаратам [1]. Показано, что 95% устойчивых к рифампицину изолятов имеют мутации в «коровом» регионе гена rpoB, кодирующем β-субъединицу РНК-полимеразы размером 81 п.н. Наиболее часто мутации возникают в 531 (Ser->Leu) и 526 (His->Tyr) кодонах. С возникновением устойчивости к рифампицину также ассоциированы другие нуклеотидные замены: в 511, 515, 516, 526 и других кодонах, встречающиеся значительно реже.
Определение устойчивости микобактерий классическими бактериологическими методами занимает 1-2 мес., которые можно считать «потерянными» для больного. Автоматизированные системы с использованием жидких сред, в основе которых лежит метод пропорций, позволяют получить результат в течение 14 дней, при этом стоимость его весьма велика. Гибридизационные методы ПЦР-анализа лекарственной устойчивости дают результат в течение 4-5 часов, также затратны и требуют дополнительного оборудования.
В настоящее время разработан ряд методов, позволяющих диагностировать лекарственную устойчивость микобактерий к рифампицину посредством прямого выявления мутаций в «коровом» регионе гена rpoB с использованием различных молекулярно-генетических подходов [1]. Методы выявления лекарственной резистентности, основанные на анализе ДНК микобактерий, выполняются в течение нескольких дней, показывают достаточную специфичность и чувствительность. Однако у данных методов есть существенный недостаток, как то высокая вероятность контаминации при большом потоке анализов. С появлением метода ПЦР в режиме «реального времени» указанный недостаток можно преодолеть, при этом существенно сократив время анализа. Помимо этого стоит отметить простоту метода для выполняемого персонала.
В 1997 г. предложен новый вид анализа продуктов ПЦР, основанный на использовании интеркалирующих флуоресцентных красителей ДНК и возможностей оборудования для проведения ПЦР в режиме «реального времени» [2]. После проведения стандартной ПЦР реакционная смесь, содержащая амплифицированный фрагмент ДНК, подвергается нагреванию, что приводит к переходу фрагмента ДНК из двухцепочечной формы в одноцепочечную, диссоциации красителя и снижению уровня флуоресценции. В результате получается характерная кривая плавления, специфичная для конкретного ПЦР-фрагмента. Попытки увеличения информативности флуоресцентного мониторинга плавления ДНК привели к появлению метода анализа кривых плавления с высоким разрешением (high resolution melting curve analysis - HRM, HRMA, HRMCA) [3]. Мутации в пределах анализируемого ПЦР-фрагмента ДНК приводят к небольшим изменениям температуры плавления, что сдвигает кривую по оси значения температуры или, в отдельных случаях, меняет ее форму. Ранее описано применение HRM-анализа для быстрой детекции мутаций, вызывающих лекарственную устойчивость у микобактерий туберкулеза [4-6]. Однако описанные подходы не позволяют достичь высоковоспроизводимых результатов, что особенно важно в клинической практике.
Наиболее близким к заявленному способу - прототипом является способ типирования, предложенный K.G.Hoek и соавт. [4], где впервые применили HRM-анализ для выявления мутаций в rpoB гене. Известно, что при анализе однонуклеотидных замен (SNP) в геноме человека методом HRM наиболее надежно выявляются гетерозиготы. Это обусловлено тем, что после плавления ампликонов двух типов и их дальнейшей реассоциации образуются молекулы четырех типов - две исходные и две несущие неспаренные основания (мис-матчи) в области SNP, обладающие значительно более низкой температурой плавления. Суперпозиция кривых плавления этих фрагментов ДНК дает характеристическую кривую, которая часто содержит два перегиба и значительно отличается от кривой плавления ампликона дикого типа. Этот факт использован в целом ряде исследований по скринингу высокопенетрантных мутаций для моногенных заболеваний человека [7]. В данной работе Хоек и соавт. амплифицировали участок rpoB гена дикого типа (штамм H37Rv) и анализируемый клинический образец, затем смешивали ампликоны, проводили еще 10 раундов ПЦР и уже после проводили анализ кривых плавления. Недостатком данного метода является снижение гомогенности и высокая вероятность кросс-контаминации ампликонами любых типов.
Предлагаемый способ выявления устойчивых к рифампицину изолятов М.tuberculosis путем определения мутаций в гене rpoB, ассоциированных с формированием устойчивости к рифампицину, заключается в проведении ПЦР в режиме «реального времени» с использованием HRM-анализа, отличается, тем, что в процессе амплификации формируются «искусственные» гетеродуплексы за счет одновременной коамплификации ДНК дикого типа и тестируемой ДНК, что может увеличить надежность анализа. В ПЦР используются праймеры следующей структуры: Rpo11 5'-GACGTGGAGGCGATCACAC-3' и Rpo12 5'-GGCACGCTCACTTGACAGAC-3'. При этом главный принцип ПЦР в режиме «реального времени», а именно анализ продуктов ПЦР с закрытой крышкой, сохраняется, что нивелирует возможность контаминации ампликонами, полученными в процессе реакции.
Способ заключается в в следующем
Выделение ДНК из культуры М.tuberculosis проводят согласно описанной методике [8].
Реакции амплификации выполняют при помощи термоциклера с оптическим блоком для детекции флуоресценции CFX96 (Bio-Rad). Для регистрации флуоресценции в режиме реального времени в реакционную смесь добавляют интеркалирующий краситель SYTO9. Качество реакции амплификации считают приемлемым при выполнении следующих условий:
- эффективность реакции амплификации больше 80% (Е>0,8);
- коэффициент корреляции между ожидаемым и эмпирическим уравнениями калибровочной кривой больше 0,99 (r2>99);
- разница между значениями Ct для повторяющихся точек калибровочной кривой не превышает 0,5 цикла;
- отсутствие неспецифичных продуктов амплификации.
Эффективность ПЦР рассчитывали при помощи уравнения (1)
log(R0)=log(RN)-log(1+E)xNt,
где
- N - независимая переменная, соответствующая номеру амплификационного цикла, на котором проводятся измерения;
- RN - количество амплифицированных молекул после N-го цикла амплификации (прямо пропорционально значению флуоресценции амплификационной смеси);
- R0 - исходное число амплифицируемых молекул;
- Е - эффективность амплификации.
Для этого с парой праймеров (Rpo11/Rpo12) амплифицируются образцы калибровочной кривой (четыре последовательных 4-кратных разведения анализируемой и референтной (H37Rv) ДНК). Используя калибровочную прямую и регрессионное уравнение (1), проводят уравнивание концентраций анализируемых образцов ДНК до концентрации наименее концентрированной ДНК.
Непосредственно ПЦР в режиме «реального времени» с последующим HRM-анализом проводят при смешивании равного количества тестируемой ДНК и ДНК H37Rv. ПЦР проводят в объеме 20 мкл, содержащем 67 мМ трис-HCl (pH 8,9), 16 мМ сульфат аммония, 1,5 мМ MgCl2, 0,01% Твин 20, 0,2 мМ дНТФ, 0,5 мкМ растворы олигонуклеотидных праймеров (Rpo11 5'-GACGTGGAGGCGATCACAC-3' и Rpo12 5'-GGCACGCTCACTTGACAGAC-3') и 1 ЕД/акт Taq-полимеразы. Реакцию проводят на амплификаторе CFX96 («Bio-Rad», США) с начальной денатурацией при 96°С в течение 2 мин, далее 35 циклов с денатурацией при 95°С в течение 5 с, отжигом праймеров при 63°С в течение 5 с и синтезом при 72°С в течение 5 с.
Для анализа кривой плавления ПЦР смесь нагревают до 96°С 5 мин, далее инкубируют минуту при температуре 63°С и проводят плавление со скоростью 0,2°С на один шаг снятия флуоресцентного сигнала, вплоть до 96°С. Для оценки качества ПЦР-аликвоты реакции фракционируют в 6% полиакриламидном геле. Для HRM-анализа используют «Precision Melt AnalysisTM Software» («Bio-Rad», США). Специфичность реакции амплификации подтверждают по отсутствию дополнительных пиков на кривой плавления и дополнительных бэндов на электрофореграмме.
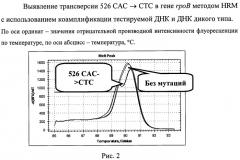
Характерные кривые плавления представлены на рисунках 1 и 2. Представлено графическое представление кривых плавления продуктов амплификации участка гена rpoB. Замена в кодоне 531 TCG->TTG гена rpoB горизонтально сдвигает пики кривых плавления влево из-за уменьшения температуры плавления (см. рис.1). Использование интеркаллирующего красителя SYBRGreen I (более дешевого) надежно выявляло данную мутацию. Нуклеотидная замена в кодоне 526 САС->СТС в гене rpoB изменяет характер кривых плавления, образуя характерный перегиб (см. рис.2), выявление данной мутации проводится с использованием SYTO9
Таким образом, предложенный подход обладает высокой специфичностью и чувствительностью при прямом анализе ДНК, выделенных из мокроты. Скрининг устойчивости к рифампицину с использованием предложенного нами подхода, учитывая его быстроту и низкую себестоимость, может быть эффективен для установления MDR-туберкулеза.
Разработанный способ выявления мутаций в гене rpoB Mycobacterium tuberculosis обладает высокой чувствительностью и специфичностью (100%), а также наиболее низкой стоимостью среди описанных к настоящему времени альтернативных подходов, имеет высокий уровень надежности и воспроизводимости. Способ апробирован на представительной выборке из 200 образцов ДНК M.tuberculosis. Суммарное время проведения анализа, начиная с биологического образца (бактериальные клетки или мокрота), не превышал 5,5 ч. Стоит отметить, что разработанный подход к анализу точковых мутаций может подходить и для анализа других нуклеотидных замен, представляющих научный и практический интерес. Разработанный метод хорошо адаптируем к инструментальным возможностям современных клинико-диагностических лабораторий и, таким образом, уже в настоящее время может быть использован во фтизиатрической практике.
Способ выявления устойчивых к рифампицину изолятов Mycobacterium tuberculosis путем определения мутаций в гене rpoB, ассоциированных с формированием устойчивости к рифампицину, посредством проведения ПЦР в режиме «реального времени» с использованием HRM-анализа, отличающийся тем, что проводят амплификацию на смеси равного количества тестируемой ДНК и ДНК дикого типа с использованием праймеровRpo11: 5'-GACGTGGAGGCGATCACAC-3';Rpo12: 5'-GGCACGCTCACTTGACAGAC-3',формируют гетеродуплексы за счет одновременной коамплификации ДНК дикого типа и тестируемой ДНК, при этом на первом этапе ПЦР проводят уравнивание концентрации с помощью количественной ПЦР с использованием той же пары праймеров (Rpo11/Rpo12) с построением калибровочной кривой и использованием регрессионного уравнения.