Способ видовой идентификации лактобацилл l.casei/paracasei, l.fermentum, l.plantarum и l.rhamnosus
Иллюстрации
Показать всеИзобретение относится к области биотехнологии и касается способа видовой идентификации лактобацилл L.casei/paracasei, L.fermentum, L.plantarum, L.rhamnosus. Предлагаемый способ включает постановку реакции ПЦР с видоспецифическими праймерами, причем конструируют праймеры, специфичные к первому гену оперона F1F0 АТФ синтазы (гену субъединицы а) и предшествующего ему гена урацилфосфорибозилтрансферазы для L.casei/paracasei и L.rhamnosus и гена урацилтранспортного белка для L.plantarum и L. fermentum. Представленное решение может быть использовано для идентификации видов лактобацилл в различных образцах микрофлоры человека и животных, в фармацевтических препаратах и в продуктах питания. 1 з.п. ф-лы, 1 ил., 3 табл.
Реферат
Область техники, к которой относится изобретение
Изобретение относится к биотехнологии, в частности к медицинской микробиологии, фармакологии и пищевой промышленности и может быть использовано для идентификации видов и штаммов лактобацилл в различных образцах микрофлоры человека и животных, в фармацевтических препаратах, в продуктах питания.
Уровень техники
Род Lactobacillus объединяет грам-положительные не образующие споры кислотоустойчивые аэротолерантные или анаэробные микроорганизмы с низким GC-составом ДНК (ниже 54%). Лактобациллы являются хемоорганотрофами и растут на богатых средах, каталазонегативны и образуют молочную кислоту как конечный продукт метаболизма углеводов. В настоящее время идентифицировано более 140 видов лактобацилл (http://www.bacterio.cict.fr/1/lactobacillus.html): ввиду обширности рода для удобства систематизации выделяют несколько филогенетических групп, каждая из которых объединяет от нескольких до двух десятков видов. Наиболее многочисленны группы L.brevis, L.buchneri, L.casei, L.delbrueckii, L.plantarum, L.reuteri, L.salivarius, L.sakei (Felis G., Dellaglio F. Taxonomy of Lactobacilli and Bifidobacteria. Curr. Issues Intestinal Microbiol., 2007, 8, 44-61). Места обитания лактобацилл чрезвычайно многообразны: различные растения, организмы животных и человека, продукты питания. Лактобациллы издавна используются для ферментации продуктов - молочных (простокваша, йогурты, сыры, сметана), мясных (колбасы, вяленое мясо, сосиски), растительных (квашение овощей, силосование). В организме человека Лактобациллы составляют важную часть микробиоты, встречаются во всех местах обитания микроорганизмов (генитальном тракте, ротовой полости, толстом и тонком кишечнике) и являются обязательными представителями нормальной микрофлоры здоровых людей. Лактобациллы широко используются в качестве лекарственных пробиотических препаратов для профилактики и лечения диареи и воспалительных заболеваний, а также в качестве биологически активных добавок и продуктов функционального питания для человека и кормовых добавок для животных (Lebeer S., Vanderleyden J., De Keersmaecker C.J. Genes and molecules of Lactobacilli supporting probiotic action. Microbiol. Molec. Biol. Rev., 2008, 72, 4, 738-764). Обнаружена корреляция между состоянием пробиотического компонента микрофлоры человека и некоторыми заболеваниями человека (Larsen N.. Vogensen F.K., van der Berg F.W.J., Nielesen D.S., Andreasen A.S., Pedersen B.K., Al-Soud W.A., Sorensen S.J., Yansen L.H., Jakobsen M. Gut microbiota in human adults with type 2 diabets differs from non-diabetic adults. PLoS ONE, 2010, 5, 2, e9085). Перечисленные свойства не принадлежат всем представителям рода лактобацилл, они являются видо- и штаммоспецифичными (MacKenzie D.A., Jeffers F., Parker M.L., Vibert-Vallet A., Bogaerts R.J., Roos S., Walter J., Juge N. Strain-specific diversity of mucus-binding proteins in the adhesion and aggregation properties of Lactobacillus reuteri. Microbiology, 2010, 156, 3368-3378). Проблема быстрой и однозначной видовой и штаммовой идентификации лактобацилл встает при изучении состава микробиоты человека, при поиске и длительном сохранении пробиотически ценных штаммов, при сравнении видов и штаммов, полученных в разных лабораториях и т.д.
Для начальной идентификации рода, филогенетической группы и вида Lactobacillus используют микробиологические и биохимические методы, однако они дают лишь предварительные сведения о систематическом положении данного микроорганизма и часто не позволяют отнести его к определенному виду. Для более точной идентификации микроорганизмов используют разнообразные молекулярно-генетические методы (Singh S., Goswami P., Singh R., Heller K.J. Application of molecular identification tools for Lactobacillus, with a focus on discrimination between closely related species: a review. LWT - Food science and technologie, 2009, 42, 448-457; Ботина С.Г., Климина К.М., Коробан Н.В., Амерханова A.M., Зинченко В.В., Даниленко В.Н. Реклассификация отечественных пробиотических культур бактерий рода Lactobacillus. Генетика, 2010, 46, 11, 1485-1492). Эти методы можно подразделить на несколько групп.
1. Методы, не связанные с ПЦР: анализ рестрикционных фрагментов хромосомной ДНК (RLFP); разделение суммарного белка клеток в SDS-PAGE электрофорезе; ДНК-ДНК гибридизация, в том числе с использованием чипов (comparative genomic hybridization, CGH) (Markiewicz L.H., Biedrzycka E., Wasilewska E., Bielecka M. Rapid molecular identification and characteristics of Lactobacillus strains. Folia Microbiol., 2010, 55, 5 481-488).
2. Методы, основанные на реакции ПЦР (Saito S., Kobayashi M., Kimoto-Nira H., Aoki R., Mizumachi K., Miyata S., Yamamoto K., Kitagawa Y., Suzuki C. Intraspecies discrimination of Lactobacillus paraplantarum by PCR. FEMS Microbiology Letters, 2011, 316, 70-76). Используются как случайные праймеры (RAPD), так и праймеры для повторяющихся последовательностей ДНК (REP-PCR, ERIC-PCR) и праймеры для определенных генов. В качестве таких генов чаще других используются гены 16S и 23S рибосомальных РНК и спейсерные районы между ними. Используются также некоторые белок-кодирующие гены: tuf (ген фактора элогации Tu), recA, hsp60 (ген белка теплового шока), rpoA (ген α-субъединицы РНК-полимеразы), dnaK (ген белка теплового шока 70 kDa) (Huang C.-H., Lee F.-L. The dnaK gene as a molecular marker for the classification and discrimination of the Lactobacillus casei group. Antonie van Leeuwenhoek, 2011, 99, 319-327), β-субъединицы F1F0-АТФ синтазы (Sievers M, Uermösi С, Fehlmann M, Krieger S. Cloning, sequence analysis and expression of the F1F0-ATPase beta-subunit from wine lactic acid bacteria. Syst Appl Microbiol., 2003, 26, 3, 350-6). Широко распространен метод, объединяющий анализ рестрикционных фрагментов и ПЦР - т.н. AFLP.
3. Методы, основанные на определении нуклеотидной последовательности (НП) ДНК: определение НП отдельных генов (или их фрагментов) рибосомальной РНК и белок-кодирующих генов, перечисленных в п.2; определение одиночных нуклеотидных замен в таких генах (SNP) (Huang C.-H., Chang M.-T., Huang M.-C., Lee F.-L. Rapid identification of Lactobacillus plantarum group using the SNaPshort minisequencing. Systematic and applied microbiology, 2011, 34, 586-589); одновременное определение НП фрагментов нескольких белок-кодирующих генов (MLST) (Raftis E., Salvetti E., Torriani S., Felis G.E., O'Toole P.W. Genomic diversity of Lactobacillus salivarius. Appl. Environ. Microbiol., 2011, 77, 3, 954-965).
Для идентификации вида чаще используется ПЦР с родо- и видоспецифичными праймерами, созданными по генам и межгенным спейсерам рибосомальных и белок-кодирующих генов, с последующим анализом продуктов реакции в электрофорезе и определением их НП. Для идентификации штаммовой принадлежности лактобацилл чаще используются рестрикционный анализ ДНК, ПЦР с неспецифическими праймерами (RAPD, REP-PCR, ERIC-PCR), определение НП ряда генов (MLST), гибридизация с чипами (CGH). Однако ни один из перечисленных методов не является универсальным, каждый имеет свои достоинства, недостатки, область применения и используется для анализа конкретных видов или групп видов и для решения конкретных задач. Считается, что для корректной идентификации вида и штамма следует использовать несколько молекулярных методов или несколько генов (Singh S., Goswami P., Singh R., Heller K.J. Application of molecular identification tools for Lactobacillus, with a focus on discrimination between closely related species: a review. LWT - Food science and technologie, 2009, 42, 448-457). Поэтому важно включение в молекулярно-генетическую идентификацию лактобацилл новых генов.
Наиболее близким к предлагаемому нами подходу является метод, разработанный Markiewicz L.H., Biedrzycka E., Wasilewska E., Bielecka M. (Rapid molecular identification and characteristics of Lactobacillus strains, Folia Microbiol., 2010, 55, 5481-488). Для идентификации видов предлагаются видоспецифичные праймеры, сделанные по нуклеотидной последовательности гена 16S РНК и примыкающего к нему межгенного района, а для идентификации штаммов - рестрикционный анализ с использованием пульс-электрофореза и метод ПЦР с неспецифическими праймерами (RAPD, REP-PCR). Т.е. идентификация вида и штамма проводилась различными методами (в нашем случае - одним). Методы, использованные авторами для идентификации штаммов, основаны на анализе многих фрагментов, получаемых при электрофорезе в агарозном геле. Они дают хорошие результаты при сравнении штаммов в одной лаборатории, однако трудно воспроизводимы и мало применимы для сравнения штаммов из разных лабораторий. Предлагаемый нами метод идентификации штаммов может быть использован для сравнения как конкретных штаммов, так и тех, чья нуклеотидная последовательность известна только из базы данных.
Раскрытие изобретения
Задачей настоящего изобретения является создание метода универсальной, дешевой, быстрой и точной молекулярно-генетической идентификации видов и штаммов лактобацилл, который может быть применен как для идентификации отдельных штаммов, так и для изучения состава микробиологических сообществ в микробиоте человека, пищевых цепях и других экосистемах.
Метод основан на использовании для видовой и штаммовой идентификации лактобацилл нового генетического маркера - межгенного района, предшествующего оперону F1F0 АТФ синтазы - и включает в себя постановку реакции ПЦР с видоспецифическими праймерами и определение нуклеотидной последовательности данного района. FIFO АТФ синтаза - важнейший фермент энергетического метаболизма клетки, присутствующий в клетках подавляющего большинства микроорганизмов. Фермент состоит из двух основных частей: растворимой F1 части, катализирующей синтез (гидролиз) АТФ, и погруженной в мембрану FO части, обеспечивающей транспорт протонов через мембрану. Фермент осуществляет синтез АТФ за счет энергии трансмембранного протонного градиента, обеспечивая клетку универсальным источником энергии (Capaldi R.A., Aggeler R. Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor. Trends Biochem Sci. 2002, 27, 3, 154-60). Для некоторых организмов - в том числе лактобацилл - показано, что фермент может осуществлять обратный процесс экспорта протонов из клетки, используя энергию АТФ; это поддерживает стабильность внутриклеточного значения рН. Гены субъединиц фермента (у лактобацилл их 8) объединены в оперон. Перед первым геном оперона находится межгенный район (109-340 пн), содержащий промотор и сайты связывания с регуляторами транскрипции (Kullen M.J, Klenhammer T.R. Identification of pH-inducible, proton-translocating F1F0-ATPase (atpBEFHAGDC) operon of Lactobacillus acidophilus by different display: gene structure, cloning and characterization. Molec. Microbiol., 1999, 33, 1152-1161).
1. Биоинформатический анализ межгенного района, предшествующего оперону F1F0 АТФ-синтазы у лактобацилл.
Используя имеющиеся в GenBank данные, мы выделили межгенные районы, предшествующие оперону F1F0 АТФ-синтазы, у лактобацилл, чьи геномы полностью секвенированы и аннотированы (39 штаммов 19-ти видов; данные на 30.12.2011 г; http://www.ncbi.nlm.nih.gov/sutils/genom_table.cgi; табл.1). Проведенный нами анализ показал, что нуклеотидные последовательности межгенного района перед опероном F0F1 АТФ-синтазы, включающего промотор, видоспецифичны. Попарное сравнение межгенных районов разных видов (алгоритм blatn, http://blast.ncbi.nlm.nih.gov/Blast.cgi) дает для большей части последовательностей 33-50% идентичности, т.е. практически сходство отсутствует. Для некоторых пар видов, принадлежащих к одной группе, процент идентичности несколько больше, однако он не превышает 91% (табл.2). Сходство нуклеотидных последовательностей межгенного района перечисленных видов совпадает с родством видов, выявленным по другим генетическим маркерам, в том числе и по нуклеотидной последовательности гена 16S РНК. Данный метод позволяет разделить близкие виды L.rhamnosus и L.casei, но не дает различий для штаммов L.casei и L.paracasei.
Попарное сравнение межгенных районов разных штаммов одного вида (L.acidophilus, L.casei, L.delbrueckii, L.fermentum, L.gasserie, L.johnsonii, L.plantarun, L.salivariuas, L.rhamnosus) дает значительно большее сходство, 99-100% идентичности. Нуклеотидные последовательности межгенных районов штаммов одного вида отличаются на 1-3 нуклеотида. У видов L. acidophilus, L.reuteri, L.salivarius идентичность НП различных штаммов меньше и составляет 94-97%. У всех видов подобные различия штаммоспецифичны и могут быть генетическими маркерами штаммов (табл.1).
| Таблица 1 | ||||
| Характеристика межгенного района, предшествующего оперону F1F0 АТФ-синтазы, у аннотированных штаммов лактобацилл из GenBank (http://www.ncbi.nlm.nih.gov/sutils/genom table.cgi). | ||||
| Вид лактобацилл | Филогенетическая группа | ГЦ-состав, % | Величина межгенного р-на перед опероном F1F0-АТФ-синтазы, пн | Сравнение идентичности нуклеотидной последовательности с аналогичным межгенным районом других штаммов данного вида |
| L.acidophilus NCFM | L.delbrueckii | 34 | 122 | L.acidophilus ATCC 4796 - 122/122=100% |
| L.acidophilus 30SC 115/122=94% | ||||
| L.amylovorus GRL1112 | L.delbrueckii | 38 | 123 | |
| L.brevis ATCC 361 | L.brevis | 46 | 208 | |
| L. buchnerie NRRL B-30929 | L.buchneri | 44 | 333 | |
| L.casei BL23 | L.casei | 46 | 302 | L.casei Zhung - 302/302-100% |
| L.casei LC2W - 302/302=100% | ||||
| L. casei BDII - 302/302=100% | ||||
| L.casei ATCC 334- 301/302=99%; | ||||
| L.crispatus ST1 | L.delbrueckii | 37 | 122 | |
| L.delbrueckii s.bulgaricus | L.delbrueckii | 49 | 109 | L.delbrueckii CNCM 1-1519 - 109/109=100%; |
| ATCC BAA-365 | L.delbrueckii 2038 - 109/109=100%; | |||
| L.delbrueckii ATCC 11842 -108/109=99%; | ||||
| L.delbrueckii CNCM 1-1632 - 106/109=99%; | ||||
| L.delbrueckii PB2003/044-T3-4 - 108/109=99% | ||||
| L.delbrueckii ND02 - 106/109=97%; | ||||
| L.fermentum IFO 3956 | L.reuteri | 51 | 326 | L.fermentum CECT 5716 - 326/326=100% |
| L.fermentum 28-3-CHN - 325/326=99%; | ||||
| L.fermentum ATCC 14931 - 325/326=99% | ||||
| L.gasseri ATCC33323 | L. delbrueckii | 35 | 343 | L.gasseri SV-16A-US - 343/343=100%; |
| L.gasseri SJGE US - 343/343=100%; | ||||
| L.gasseri 224-1 - 343/343=100%; | ||||
| L.gasseri 202-4 - 343/343-100%; | ||||
| L.gasseri JV-V03 - 339/343-99% | ||||
| L.helveticus DCP 4571 | L.delbrueckii | 37 | 123 | L.helveticus H10 - 122/123=99% |
| L.johnsonii NC 533 | L.delbrueckii | 34 | 110 | L.johnsonii ATCC33200 - 109/110=99%; |
| L.johnsonii FI 9785 - 109/110=99% | ||||
| L.johnsonii DPC 6026 - 110/110=100% | ||||
| L.kefiranofaciensis ZW3 | L.delbrueckii | 38 | 123 | |
| L.plantarum WCFS1 | L.plantarum | 44 | 277 | L.plantarum ATCC 14917 - 276/277=99%; |
| L.plantarum ST-III - 276/277=99%; | ||||
| L.plantarum JDM1 - 275/277=99% | ||||
| L.reuteri DSM 20016 F275 | L.reuteri | 38 | 314 | L.reuteri MM2-3 - 314/314=100%; |
| L.reuteri VV4-1A - 314/314=100%; | ||||
| L.reuteri JSM 112- 314/314=100%; | ||||
| L.reuteri mlc - 300/316=95%; | ||||
| L.reuleri SD212 - 296/315=94%; | ||||
| L.reureti CF 48-3A - 296/315=94%; | ||||
| L.reuteri lpuph 290/308=95%; | ||||
| L.rhamnosus GG | L.casei | 46 | 298 | L.rhamnosus ATCC 8530 - 298/298=100%; |
| L.rhamnosus Lc 705 - 298/298=100%; | ||||
| L.rhamnosus LMS2-1 - 298/298=100%; | ||||
| L.rhamnosus HN001 - 297/298=99% | ||||
| L.ruminis ATCC 27782 | L.salivarius | 44 | 288 | |
| L.sakei 23K | L.sakei | 41 | 308 | |
| L.salivarius UCC 118 | L.salivarius | 32 | 230 | L.salivarius CECT 5713 - 230/230=100%; |
| L.salivarius ATCC 11741- 230/230=100%; | ||||
| L.salivarius ACS-116-4- | ||||
| Col5a-225/230-98% | ||||
| L.sanfranciscansis TMW 1.1304 | L.fructivorans | 35 | 119 |
| Таблица 2 | ||
| Попарное сравнение идентичности нуклеотидных последовательностей межгенных районов, предшествующих оперону F1F0 АТФ-синтазы, у различных видов лактобацилл. Случаи, дающие более 50% идентичности. | ||
| Виды лактобацилл | Филогенетическая группа | Идентичность нуклеотидных последовательностей межгенных районов, предшествующих оперону F1F0 АТФ-синтазы (%) |
| L.helveticus - L.acidophilus | L.delbrueckii | 85% |
| L.helveticus - L.delbrueckii | L.delbrueckii | 66% |
| L.helveticus - L.johnsonii | L.delbrueckii | 64% |
| L.helveticus- L.gasseri | L.delbrueckii | 65% |
| L.johnsonu - L.delbrueckii | L.delbrueckii | 58% |
| L.johnsonii-L.gasseri | L.delbrueckii | 91% |
| L.rhamnosus - L.casei | L.casei | 88% |
| L.fermentum- L reuteri | L. reuteri | 54% |
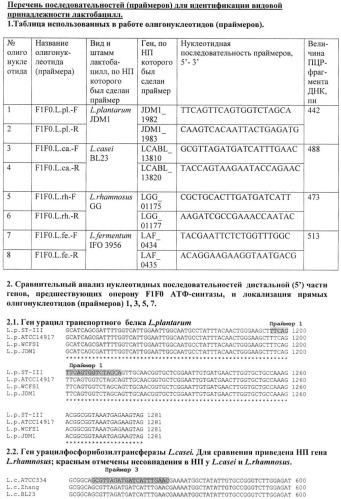
2. Конструирование праймеров для видоспецифичной ПЦР-идентификации межгенных районов, предшествующих оперону F1F0 АТФ-синтазы лактобацилл.
Для создания видоспецифичных праймеров мы выбрали четыре вида лактобацилл, которые наиболее часто встречаются в организме здоровых людей центрального района России. Это виды L.case/paracasei. L.rhamnosus, L.fermentum, L.plantarum. Мы сконструировали праймеры по нуклеотидной последовательности не самого межгенного района, а окружающих его генов, предшествующего оперону гена и первого гена оперона FIFO АТФ-синтазы. Первым геном оперона у всех проанализированных штаммов лактобацилл является ген субъединицы А мембранного комплекса F0 (atpB). Геном, предшествующим оперону, у L.casei и L.rhamnosus является ген урацилфосфорибозилтрансферазы, у L.plantarum и L.fermentum - ген урацил-транспортного белка. Как показали результаты биоинформативных исследований с использованием алгоритма blatn (http://blast.ncbi.nlm.nih.gov/Blast.cgi) (см. «Перечень последовательностей для идентификации видовой принадлежности лактобацилл»), нуклеотидная последовательность этих генов, как и межгенного района, видоспецифична, однако могут встречаться отдельные нуклеотидные замены. Праймеры были сконструированы по НП, идентичной у всех штаммов данного вида. Характеристика праймеров приведена в разделе «Перечень последовательностей для идентификации видовой принадлежности лактобацилл». Биоинформативный анализ показал отсутствие значительной идентичности НП праймеров с НП хромосом других видов лактобацилл. В качестве материала для ПЦР-реакции могли быть использованы как очищенная ДНК, так и отдельные колонии и пробы образцов, содержащих клетки исследуемого штамма; это значительно упрощает исследование. Праймеры были проверены при видовой идентификации более 70 штаммов различных лактобацилл. Продукты ПЦР образовывались и идентифицировались в электрофорезе в агарозном геле только с праймерами, свойственными данному виду. Если исследуемый штамм не принадлежал ни к одному из 4-х изучаемых видов лактобацилл, то ПЦР-продукты не обнаруживались ни с одним из праймеров. Если анализировалась смесь штаммов, то продукты ПЦР обнаруживались с праймерами, специфичными для всех компонентов смеси. Для всех проверенных штаммов результаты идентификации совпали с результатами идентификации по данным нуклеотидной последовательности гена 16S РНК.
3. Определение нуклеотидной последовательности межгенных районов, предшествующих оперону F1F0 АТФ-синтазы, для штаммоспецифичной идентификации лактобацилл.
Определение НП межгенных районов, предшествующих оперону FIFO АТФ-синтазы у лактобацилл, выявило различия между штаммами, заключающиеся в замене, делеции или вставке одного, реже большего числа нуклеотидов (табл.1, 4). Данные различия могут быть использованы для типирования штаммов - либо сами по себе, либо в сочетании с другими штаммоспецифическими маркерами.
| Таблица 3 | |||
| Нуклеотидные замены в межгенном районе перед опероном F1F0 АТФ-синтазы у различных штаммов 4-х видов лактобацилл. Жирным шрифтом отмечены штаммы из GenBank, остальные штаммы - из лабораторной коллекции. | |||
| Вид лактобацилл | Штамм | Штамм сравнения | Нуклеотидные замены |
| L.rhamnosus | LMS2-1 | GG | нет изменений |
| Lc705 | нет изменений | ||
| АТСС 8530 | нет изменений | ||
| CASL | нет изменений | ||
| АТСС 21052 | нет изменений | ||
| R0011 | нет изменений | ||
| U101 | нет изменений | ||
| 421-2 | нет изменений | ||
| 32k | нет изменений | ||
| 51зв | нет изменений | ||
| MTCC 5462 | N108+A | ||
| HN001 | N 22 G→A | ||
| 24ст | N 22 G→A | ||
| 7дст | N 22 G→A | ||
| 50 зв | N 22 G→A | ||
| 30зв | N 22 G→A | ||
| 38к | N 22 G→A | ||
| 72 зв | N 263 C→Т | ||
| L.plantarum | АТСС14917 | ST-III | нет изменений |
| 46к | нет изменений | ||
| 106зв | нет изменений | ||
| 8РАЗ | N 45 G→A | ||
| CS396 | N 45 G→A | ||
| 90ТС4 | N 45 G→A | ||
| К3 | N 45 G→A | ||
| JDM1 | N 74 А→G | ||
| 36ст | N 74 A→G | ||
| WCFS1 | N1 26 G→C | ||
| L.fermentum | СЕСТ 5716 | IFO 3956 | нет изменений |
| DSM25371 | нет изменений | ||
| 526 | нет изменений | ||
| 39зв | нет изменений | ||
| 11 зв | нет изменений | ||
| №24 ΔА; | |||
| 2 пр | №98 G→А | ||
| АТСС 14931 | №24 ΔА | ||
| 28-3-CN | №159 С→G | ||
| 11дст | №269 С→Т | ||
| 291ч | №269 С→Т | ||
| L.casei/paracasei | Zhang | BL23 | нет изменений |
| АТСС25302 | нет изменений | ||
| 8700-2 | нет изменений | ||
| BD-II | нет изменений | ||
| LC2W | нет изменений | ||
| 577 | нет изменений | ||
| 17k | нет изменений | ||
| ATCC334 | N 75 G→A | ||
| БТ 24-88 | N 86 С→Т | ||
| 23л | N 149 G→T | ||
| К3Ш24 | N 149 G→T |
Таким образом, предложенный нами способ идентификации лактобацилл позволяет определять не только видовую, но, в ряде случаев, и штаммовую принадлежность лактобацилл. Кроме того, он значительно проще метода, основанного на анализе гена 16S РНК: для L.casei, L.plantarum, L.fermentum, L.rhamnosus вид микроорганизмов может быть установлен по результатам ПЦР, без секвенирования продуктов ПЦР; в данном методе могут быть использованы микроколичества биомассы бактерий, без выделения ДНК. Метод позволяет разделять близкие виды L.casei - L.rhamnosus.
Осуществление изобретения
Культуру лактобацилл выращивают на среде MFC ("HiMedia", Индия) в течение 48 часов при 37°С при анаэробных условиях в анаэростате HiAnaerobicTM ("HiMedia", Индия). Бактериальная масса с чашки может быть суспендирована в минимальном объеме стерильной воды, и 1 мкл суспензии может быть использован в реакции ПЦР.
В реакции ПЦР может быть использована и геномная ДНК. Для ее выделения клетки из 20 мл ночной культуры осаждают центрифугированием при 4000g в течение 10 мин, промывают 2 мл буфера (10 мМ Трис-HCl, 10 мМ ЭДТА-Na2, рН 8,0) и ресуспендируют в 500 мкл выше указанного буфера, затем суспензию переносят в 2-мл центрифужную пробирку и добавляют 50 мкл хлороформа. Смесь энергично встряхивают с помощью вортекса (5 раз по 10 с), вносят 100 мкл раствора лизоцима (60 мг/мл) и инкубируют 30 мин при 37°С. Добавляют 6 мкл РНКазы А (10 мг/мл) и инкубируют еще 30 мин при 37°С. Далее к суспензии добавляют 200 мкл 10% SDS и 200 мкл 5М NaCl, осторожно перемешивают и инкубируют 16 ч при 65°С. Полученный лизат клеток остужают до комнатной температуры, добавляют 1 мл смеси фенол/хлороформ (1:1) и перемешивают путем переворачивания пробирки в течение 5 мин до состояния гомогенной эмульсии. Затем смесь центрифугируют при 12000g в течение 15 мин. Водную фазу, содержащую ДНК, отбирают в новую 1,5-мл пробирку и смешивают с 600 мкл изопропанола. Смесь инкубируют 30 мин при комнатной температуре и центрифугируют при 12000 g в течение 20 мин. Осадок ДНК трижды промывают порциями по 0,5 мл 75% этанола с центрифугированием по 5 мин при 12000 g и растворяют в 100 мкл воды.
Амплификацию ДНК проводят с использованием набора «Амплификация» фирмы «Dialat Ltd» на приборе «Терцик» («ДНК-технология»). Состав смеси для ПЦР (на 100 мкл): 10 мкл 10×ПЦР буфера, 10 мкл смеси 2,5 mM ΣdNTPs, 4 мкл 50 mM MgCl2, 0,3 мкг геномной ДНК и 0,8 мкл фермента Taq-полимеразы. Олигонуклеотидные праймеры добавляют в концентрации 20 пмоль на 100 мкл смеси.
Параметры ПЦР реакции: 94°С в течение 5 мин (лизис клеток и денатурация геномной ДНК); затем 30 циклов амплификации - 94°С - 20 с (денатурация), 56°С в течение 20 с (отжиг олигонуклеотидов), 72°С - 30 с (достройка нити ДНК); финальная элонгация фрагментов при 72°С - 5 мин.
Результаты исследования учитывают путем анализа продуктов амплификации исследуемых образцов методом электрофореза в 1% агарозном геле. В пробирки с исследуемыми образцами после завершения амплификации вносят под масло 1/5 объема раствора 6Х DNA Loading Dye («Fermentas»), перемешивают и 8 мкл полученного образца вносят в лунки агарозного геля. Электрофорез проводят в камере для горизонтального электрофореза "SE-2" (Компания «Хеликон») с источником питания "Эльф-4" («ДНК-технология») при напряжении 120 вольт в течение 60 мин. Результаты электрофореза учитывают в ультрафиолетовом свете с длиной волны 254 нм на трансиллюминаторе TCP-20 MC («Vilber Lourmat», Франция). В качестве контроля размера полученного фрагмента используют ДНК маркер GeneRuler™ 100+ п.н. («Fermentas»).
Для определения нуклеотидной последовательности фрагменты ПЦР вырезают из геля и очищают от агарозы с помощью набора Fermentas K0691. Для этого добавляют Binding Buffer (на 100 мг агарозы - 100 мкл буфера); инкубируют гель в буфере 10-15 мин при 50-60°С до полного растворения геля; добавляют изопропанол (на 100 мг геля и 100 мкл буфера - 100 мкл изопропанола); помещают растворенный гель на колонку и откручивают 1 мин; промывают колонку 100 мкл Binding Buffer, затем 700 мкл Wash Buffer; откручивают пустую колонку еще раз 1 мин для полного удаления буфера и содержащегося в нем спирта; помещают колонку в чистую пробирку на 1,5 мл и смывают ДНК с колонки 20-50 мкл Elution Buffer.
Определение нуклеотидной последовательности фрагментов производилось на приборе 3730х1 DNA Analyzer (Applied Biosystems,USA) с использованием набора BigDye® Terminator v3.1 Cycle Sequencing Kit (Applied Biosystems, USA) в НИИ ФХМ ФМБА РФ.
Примеры идентификации по настоящему изобретению.
1. Определение вида штаммов лактобацилл 24 ст и 2 пр.
Клетки штаммов 24 ст и 2 пр были использованы в реакции ПЦР в качестве матрицы с видоспецифичными праймерами, перечисленными в «Перечне последовательностей». Продукты ПЦР для клеток штамма 24 ст обнаруживаются только с праймерами 5-6, а для клеток штамма 2 пр - только с праймерами 7-8 (рис.1), следовательно, штамм 24 ст принадлежит к виду L.rhamnosus, штамм 2пр - к виду L.fermentum.
2. Определение специфических штаммовых характеристик штаммов 24 ст и 2 пр.
ПЦР-фрагменты штаммов L.rhamnosus 24 ст и L.fermentum 2 пр (см. рис.1) были выделены из агарозы, секвенированы и их нуклеотидная последовательностей была сопоставлена с нуклеотидной последовательностью штаммов из GenBank L.rhamnosus GG и L.fermentum IFO 3956 соответственно. Установлено, что штамм 24 ст отличается от штамма GG заменой одного нуклеотида в межгенном районе, предшествующем оперону F1F0 АТФ-синтазы (N 22 G→А); штамм 24 ст по данному показателю идентичен штамму HN001 из GenBank и ряду лабораторных штаммов (табл.3). Штамм 2 пр отличается от штамма IFO 3956 одной делецией (№24 ΔА) и одной заменой нуклеотида (№98 G→А) и отличен от всех описанных штаммов L.fermentum.
1. Способ видовой идентификации лактобацилл L.casei/paracasei, L.fermentum, L.plantarum, L.rhamnosus, включающий постановку реакции ПЦР с видоспецифическими праймерами, отличающийся тем, что конструируют праймеры, специфичные к первому гену оперона F1F0 АТФ синтазы (гену субъединицы а) и предшествующего ему гена урацилфосфорибозилтрансферазы для L.casei/paracasei и L.rhamnosus и гена урацилтранспортного белка для L.plantarum и L. fermentum.
2. Способ по п.1, отличающийся тем, что включает в себя определение нуклеотидной последовательности данного межгенного района.