Способ идентификации лактобацилл
Иллюстрации
Показать всеИзобретение относится к области биотехнологии и касается способа идентификации лактобацилл. Представленный способ основан на комбинации и полиморфизме генов токсин-антитоксин суперсемейств RelBE и MazEF и характеризуется тем, что для идентификации проводят амплификацию геномных ДНК с использованием набора олегонуклеотидов определенной структуры, ПЦР-продукты анализируют в агарозном геле, а размер полученного фрагмента определяют с помощью ДНК-маркера. Исследуемый штамм относят к определенной группе, виду или штамму в случае наработки фрагментов при использовании определенных олигонуклеотидов. Представленное изобретение может быть использовано для идентификации штаммов лактобацилл в микробиоте человека, продуктах питания, а также для определения исследуемого штамма в клинических пробах или молекулярного отслеживания в коммерческих препаратах. 3 табл., 3 пр.
Реферат
Область техники, к которой относится изобретение
Изобретение относится к биотехнологии, в частности к медицинской микробиологии, а также к производству заквасок для кисломолочных продуктов питания, и может быть использовано для идентификации штаммов лактобацилл в микробиоте человека, продуктах питания и в цепочке их изготовления.
Уровень техники
Бактерии рода Lactobacillus составляют незначительную часть микробиоты кишечника взрослого человека, приблизительно 0,01-0,6% от всех обитателей желудочно-кишечного тракта, однако активно осуществляют регуляторные функции внутри популяции кишечных бактерий и являются представителями нормальной микрофлоры кишечника здоровых людей (Jones B.V., Sun F., Marches J.R. Comparative metagenomic analysis of plasmid encoded functions in the human gut microbiome BMC Genomics 2010, 11:46; Saulnier DM, Kolida S, Gibson GR. Microbiology of the human intestinal tract and approaches for its dietary modulation. Curr Pharm Des. 2009,15 (13), 1403-1414; Ботина С.Г., Коробан Н.В., Климина К.М., Глазова А.А., Захарович Н.В., Зинченко В.В., Даниленко В.Н. Генетическое разнообразие бактерий рода Lactobacillus из гастроинтестинальной микробиомы людей. Генетика, 2010, том 46, №12, с.1-9).
Лактобациллы являются важным биотехнологическим объектом, они используются в качестве заквасок при производстве кисломолочных продуктов питания (йогурт, простокваша, ряженка и сыры). Лактобациллы широко применяются в составе лекарственных пробиотических препаратов для профилактики кишечных расстройств и вирусной диареи, лечения воспалительных заболеваний кишечника, а также в качестве биологически активных добавок, продуктов функционального питания для человека и кормовых добавок для животных (Gupta V, Garg R. Probiotics. Indian J Med Microbiol. 2009, 27 (3), 202-9; Kaur IP, Kuhad A, Garg A, Chopra K. Probiotics: delineation of prophylactic and therapeutic benefits. J Med Food. 2009, 12 (2), 219-35; Guarino A, Lo Vecchio A, Canani RB. Probiotics as prevention and treatment for diarrhea. Curr Opin Gastroenterol. 2009, 25 (1), 18-23.)
В последние годы обнаружена корреляция между состоянием пробиотического компонента микрофлоры человека и некоторыми заболеваниями, в том числе болезнью Крона и онкозаболеваниями (Edwards LA, Lucas M, Edwards EA, Torrente F, Heuschkel RB, Klein NJ. Murch SH, Bajaj-Elliott M, Phillips AD. Aberrant response to commensal Bacteroides thetaiotaomicron in Crohn's disease: an ex vivo human organ culture study. Inflamm Bowel Dis. 2011 May; 17(5):1201-8; Cain AM, Karpa KD. Clinical utility of probiotics in inflammatory bowel disease. Altern Ther Health Med. 2011 Jan-Feb; 17(1):72-9).
Поэтому актуальной задачей является использование лактобацилл в качестве биомаркеров для тестирования ранних стадий различных заболеваний человека. Установлено, что пробиотические свойства лактобацилл являются штаммоспецифическими.
Род Lactobacillus насчитывает около 140 видов, многие из которых являются комменсалами в составе микробиоты человека и имеют сходные фенотипические и физиологические характеристики, но их геномы сильно различаются по GC-составу, что усложняет идентификацию данного рода. Выделяют 4 наиболее распространенные и имеющие практическое значение филогенетические группы лактобацилл, различающихся по GC-составу: группа L.acidophilus (32-37%), группа L. plantarum (44%), группа L.casei (46%), группа подвидов L.delbrueckii (49-51%). (Singh S, Goswami P, Singh R, Heller K.J. Application of molecular identification tools for Lactobacillus, with a focus on discrimination between closely related species: A review LWT - Food Science and Technology, 2009, 42, 448-457; Felis GE, Dellaglio F. Taxonomy of Lactobacilli and Bifidobacteria. Curr Issues Intest Microbiol. 2007 Sep; 8(2):44-61).
В настоящее время для идентификации вида, филогенетической группы и рода Lactobacillus применяются следующие методы:
1. Амплификация специфических для конкретного вида лактобацилл фрагментов генов 16S и 23S рРНК, также и с последующим DGGE- или TTGE-электрофорезом. (Ботина С.Г., Климина К.М., Коробан Н.В., Амерханова A.M., Зинченко В.В., Даниленко В.Н. Реклассификация отечественных пробиотических культур бактерий рода Lactobacillus. Генетика, 2010, том 46, №11, с.1485-1492; Ward L. J. H., Timmins M. J. Differentiation of Lactobacillus casei, Lactobacillus paracasei and Lactobacillus rhamnosus by polymerase chain reaction. Letters in Applied Microbiology, 1999, 29, 90-92; Dobson С.M., Chaban В., Deneer, H., Ziola B. Lactobacillus casei, Lactobacillus rhamnosus, and Lactobacillus zeae isolates identified by sequence signature and immunoblot phenotype. Canadian Journal of Microbiology, 2004, 50, 482-488; Vasquez, A, Ahme S., Pettersson В., Molin, G. Temporal temperature gradient gel electrophoresis (TTGE) as a tool for identification of Lactobacillus casei, Lactobacillus paracasei, Lactobacillus zeae and Lactobacillus rhamnosus. Letters in Applied Microbiology, 2001, 32, 215-219; Walter J., Tannock G. W, Tilsala-Timisjarvi A., Rodtong S., Loach D. M., Munro K., et al. Detection and identification of gastrointestinal Lactobacillus species by DGGE and species-specific PCR primers. Applied and Environmental Microbiology, 2000, 66, 297-303; Kullen, M. J., Sanozky-Dawes, R. В., Crowell, D. C., & Klaenhammer, T. R. Use of the DNA sequence of variable regions of the 16S rRNA gene for rapid and accurate identification of bacteria in the Lactobacillus acidophilus complex. Journal of Applied Microbiology, 2000, 89, 511-516; Berthier F., Ehrlich S. D. Rapid species identification within the groups of closely related Lactobacilli using PCR primers that target the 16S/23S rRNA spacer region. FEMS Microbiology Letters, 1998, 161, 97-106; Nissen H., Dainty R. H. Comparison of the use of rRNA probes and conventional methods in identifying strains of Lactobacillus sake and L. curvatus isolated from meat. International Journal of Food Microbiology, 1995, 25, 311-315).
По нуклеотидным последовательностям генов 16S рРНК разработаны родо-, групп - по - и видоспецифичные праймеры, амплификация с которыми в ряде случаев может проводиться одновременно в общей реакционной смеси (так называемая мультиплексная ПЦР - multiplex PCR). Данный метод обычно используется для определения вида микроорганизмов, однако неприменим для идентификации близкородственных видов и штаммов. (Song, Y., Kato, N., Liu, C., Matsumiya, Y., Kato, H., Watanabe, K.. Rapid identification of 11 human intestinal Lactobacillus species by multiplex PCR assays using group and species-specific primers derived from the 16S-23S rRNA intergenic spacer region and its flanking 23S rRNA. FEMS Microbiology Letters, 2000, 187, 167-173; Lee J., Jang J., Kirn В., Kim J., Jeong G., Han H. Identification of Lactobacillus sakei and Lactobacillus curvatus by multiplex PCR-based restriction enzyme analysis. Journal of Microbiological Methods, 2004, 59, 1-6.).
2. RAPD - амплификация с использованием случайных праймеров. Данный метод позволяет различить штаммы микроорганизмов, однако результаты не всегда воспроизводимы в разных лабораториях, метод плохо поддается стандартизации (Ward L. J. H., Timmins M. J. Differentiation of Lactobacillus casei, Lactobacillus paracasei and Lactobacillus rhamnosus by polymerase chain reaction. Letters in Applied Microbiology, 1999, 29, 90-92; Rodas A. M., Ferrer, S., Pardo, I. Polyphasic study of wine Lactobacillus strains, taxonomic implications. International Journal of Systematic and Evolutionary Microbiology, 2005, 55, 197-207; Torriani S., Felis G. E., Dellaglio F. Differentiation of Lactobacillus plantarum, L. pentosus and L. paraplantarum by rec A gene sequence analysis and multiplex PCR assay with recA gene derived primers. Applied and Environmental Microbiology, 2001, 67, 3450-3454; Berthier F., Ehrlich, S. D. Genetic diversity within Lactobacillus sakei and Lactobacillus curvatus and design of PCR primers for its detection using randomly amplified polymorphic DNA. International Journal of Systematic Bacteriology, 1999, 49, 997-1007-Andrighetto C., Zampese L., Lombardi A. RAPD-PCR characterization of lactobacilli isolated from artisanal meat plants and traditional fermented sausages ofVeneto region (Italy). Letters in Applied Microbiology, 2001, 33, 26-30).
3. Анализ последовательностей консервативных генов «домашнего хозяйства», в том числе recA, tuf. Т.н. метод MLST (multi locus sequencing typing) идентифицирует нуклеотидную последовательность и ее вариации для ряда генов «домашнего хозяйства»; эти данные позволяют выделить внутри вида конкретные штаммы. Метод универсален, однако достаточно сложен и трудоемок. В настоящее время базы данных по MLST существуют только для двух видов лактобацилл - L.casei и L.salivarius (Raftis EJ, Salvetti E, Torriani S, Felis GE, O'Toole PW, Genomic diversity of Lactobacillus salivarius, Appi Environ Microbiol. 2011 77(3):954-65); Cai H., Rodeigez B.T., Zhang W., Broadbent J.R., Steele J.L. Genotypic and phenotypic characterization of Lactobacillus casei strains isolated from different ecological niches suggests frequent recombination and niche specificity. Microbiology, 2007, V.I 53, 2655-2665; Chavagnat F., Haueter M., Jimeno J., Casey M. G. Comparison of partial tuf gene sequences for the identification of lactobacilli. FEMS Microbiology Letters, 2002, 217, 177-183; Felis G. E., Dellaglio F., Mizzi L, Torriani S. Comparative sequence analysis of a rec A gene fragment brings new evidence for a change in the taxonomy of the Lactobacillus casei group. International Journal of Systematic and Evolutionary Microbiology, 2001, 51, 2113-2117; Torriani S., Felis G. E., Dellaglio F. Differentiation of Lactobacillus plantarum, L. pentosus and L. paraplantarum by rec A gene sequence analysis and multiplex PCR assay with recA gene derived primers. Applied and Environmental Microbiology, 2001,67,3450-3454).
4. Типирование с использованием рестриктаз EcoRI, EcoRI/PstI, Bfal и Msel. (Ryu С.S., Czajka J. W., Sakamoto M., Benno Y. Characterization of Lactobacillus casei group and the Lactobacillus acidophilus group by automated ribotyping. Microbiology and Immunology, 2001, 45, 271-275; Giraffa G., DeVecchi P., Rossetti L. Identification of Lactobacillus delbrueckii subspecies bulgaricus and subspecies lactis dairy isolates by amplified rDNA restriction analysis. Journal of Applied Microbiology, 1998, 85, 918-924; Miteva V., Boudakov I., Ivanova-Stoyancheva G., Marinova В., Mitev V., Mengaud J. Differentiation of Lactobacillus delbrueckii subspecies by ribotyping and amplified ribosomal DNA restriction analysis (ARDRA). Journal of Applied Microbiology, 2001, 90, 909-918; Lick S., Brockmann E., Heller K. J. Identification of Lactobacillus delbrueckii and subspecies by hybridization probes and PCR. Systematic and Applied Microbiology, 2000, 23, 251-259; Rodas, A. M„ Ferrer, S., Pardo, I. Polyphasic study of wine Lactobacillus strains, taxonomic implications. International Journal of Systematic and Evolutionary Microbiology, 2005, 55, 197-207).
Однако перечисленные выше методы в основном применимы для типирования групп и отдельных видов лактобацилл, известные методы штаммоспецифичной идентификации не являются универсальными и простыми в использовании и не позволяют проводить штамм-специфическую идентификацию, поскольку нуклеотидные последовательности генов, используемых для групповой и видовой идентификации, полностью идентичны для разных штаммов одного вида.
Для типирования штаммов L.delbrueckii разработан так называемый мультилокусный метод, основанный на RFLP - ПЦР трех белок-кодирующих генов: β-галактозидаза, лактоза пермеаза и пролин дипептидаза (Giraffa G., Lazzi С., Gatti M., Rossetti L., Mora D., Neviani, E. Molecular typing of Lactobacillus delbrueckii of dairy origin by PCR-RFLP of protein-coding genes. International Journal of Food Microbiology, 2003, 82, 163-172).
Для штаммов вида L.ramnosus до настоящего времени не разработаны методы штамм-специфической идентификации.
Раскрытие изобретения
Задачей настоящего изобретения является создание метода универсальной и быстрой молекулярно-генетической идентификации филогенетических групп, видов и штаммов лактобацилл в микробиоте человека (гастроэнтерологический тракт, вагинальная полость и др.), а также в пищевой цепочке кисломолочных продуктов.
Метод идентификации основан на комбинации и полиморфизме генов систем токсин-антитоксин relBE и mazEF (Yamaguchi Y, Park JH, Inouye M. Toxin-antitoxin systems in bacteria and archaea. Annu Rev Genet. 2011, Dec 15;45:61-79; Прозоров А.А., Даниленко В.Н. Системы "токсин-антитоксин" у бактерий: инструмент апоптоза или модуляторы метаболизма? Микробиология. 2010. Т.79. №2. С.147-159).
Компонентами систем токсин-антитоксин (ТА) обычно являются два гена, располагающиеся один за другим (иногда перекрываясь), снабженные, по крайней мере, одним промотором, и образующие оперон. Ген токсина обуславливает образование долгоживущего «отравляющего» бактерию продукта, а ген антитоксина - лабильного продукта, способного нейтрализовать этот токсин, связываясь с ним (или препятствуя его образованию). (Engelberg-Kulka H, Amitai S, Kolodkin-Gal I, Hazan R. Bacterial programmed cell death and multicellular behavior in bacteria. PLoS Genet. 2006, 2 (10): 1518-1526.)
1. Биоинформатический анализ наличия генов систем токсин-антитоксин mazEF и relBE у лактобацилл.
Авторами настоящего изобретения был проведен компьютерный поиск генов систем mazEF и relBE в геномах всех 4 групп лактобацилл, различающихся GC-составом, с использованием программ National Center for Biotechnology Information (NCBI) (http://www.ncbi.nlm.nih.gov/) и UniProt (http://www.uniprot.org/). Сравнительный анализ последовательностей обнаруженных генов проводили с использованием программ Blast (http://blast.ncbi.nlm.nih.gov/) и CLUSTAL W (www.ch.embnet.org/software/ClustalW.html).
В результате биоинформатического анализа для 3 групп лактобацилл обнаружены существенные различия по наличию генов данных систем токсин-антитоксин.
У штаммов филогенетической группы Lactobacillus acidophilus (L. acidophilus; L. helveticus; L. Amylovorus; L. crispatus) обнаружено по 1 модулю системы mazEF, от 1 до 4 генов токсина relE и от 1 до 2 генов антитоксина relB. Причем штаммы всех видов, относящихся к данной группе, содержат 1 ген антитоксина relB, который может быть использован для идентификации данной группы лактобацилл. Штаммы, относящиеся к виду L. Helveticus, содержат еще по 1 высокогомологичному гену антитоксина relB. Данный ген может быть использован для идентификации вида L. helveticus.
У штаммов филогенетической группы Lactobacillus plantarum (L. Plantarum; L.pentosus) обнаружено по 1 гену токсина mazF, который также может быть использован для идентификации этой группы лактобацилл.
У штаммов филогенетической группы Lactobacillus casei (L.casei; L.paracasei; L.rhamnosus) обнаружено от 1 до 2 модулей системы mazEF и от 1 до 2 модулей системы relBE, причем ген токсина mazF присутствует у всех штаммов данной группы и может быть использован для идентификации данной группы лактобацилл. Все остальные гены имеют видовую и штаммовую специфичность.
Для штаммов вида L.rhamnosus обнаружены существенные различия по наличию генов систем токсин-антитоксин mazEF и relBE (см. табл.1), что позволяет разработать метод штамм-специфической идентификации, основанный на комбинации генов.
2. Разработка олигонуклеотидов для идентификации филогенетических групп и отдельных видов лактобацилл
2.1. Идентификация филогенетической группы Lactobacillus acidophilus
К группе Lactobacillus acidophilus относятся виды L.acidophilus; L. helveticus; L. amylovorus; L. crispatus. Поскольку все штаммы этой группы содержат ген антитоксина relB, на основании нуклеотидных последовательностей этого гена были сконструированы олигонуклеотиды для групповой идентификации.
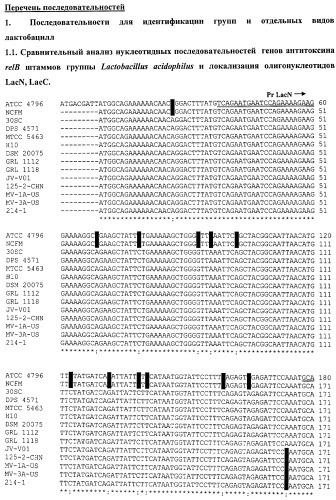
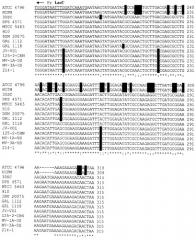
На последовательности 1.1. (см. перечень последовательностей) представлен сравнительный анализ нуклеотидных последовательностей генов антитоксина relB L.acidophilus (штаммы АТСС 4796, NCFM, 30SC); L. helveticus (штаммы DPC 4571, МТСС 5463, НЮ, DSM 20075); L. amylovorus (штаммы GRL 1112, GRL1118); L. crispatus (штаммы JV-V01, 125-2-CHN, MV-1A-US, MV-3A-US, 214-1).
На всех представленных нуклеотидных последовательностях отличия выделены красным цветом. Гомология нуклеотидных последовательностей данного гена для штаммов разных составляет от 95 до 100%.
На основании сравнительного анализа последовательностей разработаны олигонуклеотиды LacN и LacC для идентификации группы L.acidophilus.
Поскольку в геномах штаммов L. helveticus присутствует дополнительно еще 1 ген антитоксина relB, проведен сравнительный анализ данного гена, представленный на последовательности 1.2., и разработаны олигонуклеотиды LhvN1 и LhvC1 для видовой идентификации L. helveticus.
2.2. Идентификация филогенетической группы Lactobacillus plantarum
К группе Lactobacillus plantarum относятся виды L.plantarum и L.pentosus. Поскольку все штаммы этой группы содержат ген токсина mazF, на основании нуклеотидных последовательностей этого гена были сконструированы олигонуклеотиды для групповой идентификации.
На последовательности 1.3. (см. перечень последовательностей) представлен сравнительный анализ нуклеотидных последовательностей генов токсина mazF L.plantarum (штаммы АТСС 14917, WCFS1, JDM1, ST-III); L.pentosus (штаммы IG1, МР-10).
На основании сравнительного анализа последовательностей разработаны олигонуклеотиды LplN и LplC для идентификации группы L.plantarum.
2.3. Идентификация филогенетической группы Lactobacillus сasei
К группе Lactobacillus casei относятся виды L.casei, L.paracasei, L.rhamnosus. Поскольку все штаммы этой группы содержат ген токсина mazF, на основании нуклеотидных последовательностей этого гена были сконструированы олигонуклеотиды для групповой идентификации.
На последовательности 1.4. (см. перечень последовательностей) представлен сравнительный анализ нуклеотидных последовательностей генов токсина mazF L.casei (штаммы АТСС 334, BL23, Zhang, LC2W, BD-II); L.paracasei (штаммы АТСС 25302, 8700:2), L.rhamnosus (штаммы GG, Lc 705, HN001, LMS2-1, ATCC 8530).
На основании сравнительного анализа последовательностей разработаны олигонуклеотиды LcsN и LcsC для идентификации группы L.casei, олигонуклеотиды LcsN1 и LcsC1 для видовой идентификации L.casei.
Для штаммов вида L.rhamnosus проведен сравнительный анализ нуклеотидных последовательностей генов токсина relE и разработаны олигонуклеотиды RelER1N и RelER1C для видовой идентификации L.rhamnosus (см. последовательность 1.5.).
3. Идентификация штаммов вида L.rhamnosus
С увеличением числа доступных секвенированных геномов лактобацилл появилась возможность идентифицировать участки ДНК, специфичные для каждого конкретного штамма.
Как было сказано выше, для штаммов вида L.rhamnosus обнаружены существенные различия в комбинации генов систем токсин-антитоксин mazEF и relBE.
Наибольшее количество генов антитоксина присутствует в геноме штамма LMS2-1: 2 модуля системы mazEF (mazEF2 и mazEF3) и дополнительно 1 ген токсина mazFl, 1 модуль системы relBE (геlВЕ2) и дополнительно 1 ген токсина relE1 и 1 ген антитоксина rеlВ3.
В геноме штамма Lc705 присутствуют: 1 модуль системы mazEF (mazEF2) и дополнительно 1 ген токсина mazF1, 1 модуль системы relBE (rеlВЕ2) и дополнительно 1 ген токсина relE1 и 1 ген антитоксина геlВЗ.
В геноме штамма HN001 присутствуют: 1 модуль системы mazEF (mazEF3) и дополнительно 1 ген токсина mazF1, 2 модуля системы relBE (relBE1 и rеlВЕ2).
В геноме штамма АТСС 8530 присутствуют: 1 модуль системы mazEF (mazEF2) и дополнительно 1 ген токсина mazF1, 1 модуль системы relBE (rеlВЕ2) и дополнительно 1 ген токсина relE1.
В геноме штамма GG присутствуют: 1 ген токсина mazF1, 1 модуль системы relBE (relBE1).
Проведен сравнительный анализ нуклеотидных последовательностей всех выявленных генов систем mazEF и relBE штаммов L.rhamnosus (см. последовательности 2.1.-2.5.), на основании которого разработаны олигонуклеотиды для штаммовой идентификации.
Таким образом:
1) Метод по настоящему изобретению основан на комбинации генов токсин-антитоксин систем mazEF и relBE.
2) Предлагаемый метод включает набор пар олигонуклеотидов для идентификации групп и отдельных видов лактобацилл.
3) Предлагаемый метод включает набор пар олигонуклеотидов для штаммовой идентификации L.rhamnosus
Осуществление изобретения
Культуру лактобацилл выращивают на среде МРС в течение 48 часов при 37°С при анаэробных условиях в анаэростате HiAnaerobicTM (Компания "HiMedia" (Индия)), после чего выделяют геномную ДНК.
Клетки из 20 мл ночной культуры осаждают центрифугированием при 4000g в течение 10 мин и ресуспендируют в 10 мл буфера (10 мМ Трис-HCl, 10 мМ ЭДТА-Nа2, рН 8,0). Полученную суспензию центрифугируют в тех же условиях и осадок ресуспендируют в 500 мкл выше указанного буфера, затем суспензию переносят в 2-мл центрифужную пробирку и добавляют 50 мкл хлороформа. Смесь энергично встряхивают с помощью вортекса (5 раз по 10 с), вносят 100 мкл раствора лизоцима (60 мг/мл) и инкубируют 30 мин при 37°С. Добавляют 6 мкл РНКазы А (10 мг/мл) и инкубируют еще 30 мин при 37°С. Для лизиса клеток к суспензии добавляют 200 мкл 10% SDS и 200 мкл 5М NaCl, осторожно перемешивают и инкубируют 16 ч при 65°С.Полученный лизат клеток остужают до комнатной температуры, добавляют 1 мл смеси фенол/хлороформ (1:1) и перемешивают путем переворачивания пробирки в течение 5 мин до состояния гомогенной эмульсии. Затем смесь центрифугируют при 12000g в течение 15 мин. Водную фазу, содержащую ДНК, отбирают в новую 1,5-мл пробирку и смешивают с 600 мкл изопропанола. Смесь инкубируют 30 мин при комнатной температуре и центрифугируют при 12000g в течение 20 мин. Осадок ДНК трижды промывают порциями по 0,5 мл 75% этанола с центрифугированием по 5 мин при 12000g и растворяют в 100 мкл воды.
Амплификацию ДНК проводят с использованием набора «Амплификация» фирмы «Dialat Ltd» на приборе «Терцик» («ДНК-технология»). Состав смеси для ПЦР (на 100 мкл): 10 мкл 10×ПЦР буфера, 10 мкл смеси 2,5mM ∑dNTPs, 4 мкл 50 тМ MgCl2, 0,3 мкг геномной ДНК и 0,8 мкл фермента Taq-полимеразы. Олигонуклеотидные праймеры добавляют в концентрации 20 пмоль на 100 мкл смеси.
Параметры ПЦР реакции: 95°С в течение 5 мин (лизис клеток и денатурация геномной ДНК); затем 30 циклов амплификации - 94°С - 1 мин (денатурация), 60°С в течение 1 мин (отжиг олигонуклеотидов), 72°С - 1 мин (достройка (элонгация) цепи); финальная элонгация фрагментов при 72°С - 10 мин, хранение при 4°С.
Результаты исследования учитывают путем анализа продуктов амплификации исследуемых образцов методом электрофореза в 1% агарозном геле. В пробирки с исследуемыми образцами после завершения амплификации вносят аккуратно под масло 1/5 объема раствора 6Х DNA Loading Dye («Fermentas»), перемешивают и 8 мкл полученного образца вносят в лунки агарозного геля. Электрофорез проводят в камере для горизонтального электрофореза "SE-2" (Компания «Хеликон») с источником питания " Эльф-4" («ДНК-технология») при напряжении 120 вольт в течение 60 мин. Результаты электрофореза учитывают в ультрафиолетовом свете с длиной волны 254 нм на трансиллюминаторе TCP-20 MC («Vilber Lorn-mat», Франция). В качестве контроля размера полученного фрагмента используют ДНК маркер GeneRuler™ 100+п.н. («Fermentas»).
Примеры идентификации по настоящему изобретению.
Пример 1.
Идентификация групп и отдельных видов лактобацилл
Для идентификации групп L.acidophilus, L. plantarum и L.casei, видов L.acidophilus, L. helveticus и L.rhamnosus проводят амплификацию с геномной ДНК с использованием олигонуклеотидов, представленых в таблице 2. ПЦР продукты анализируют в 1% агарозном геле. Размер полученного фрагмента определяют с помощью ДНК-маркера GeneRulerTM 100+п.н. («Fermentas»).
Исследуемый штамм относится к группе L.acidophilus в случае наработки фрагментов при использовании олигонуклеотидов LacN и LacC; в случае наличия ПЦР продуктов с использованием олигонуклеотидов Lhv1N и Lhv1C - к виду L. helveticus.
Исследуемый штамм относится к группе L. plantarum в случае наработки фрагментов при использовании олигонуклеотидов Lp1N и Lp1C.
Исследуемый штамм относится к группе L.casei в случае наработки фрагментов при использовании олигонуклеотидов LcsN и LcsC; в случае наличия ПЦР продуктов с использованием олигонуклеотидов Lcs1N и Lcs1C к виду L.casei; RelER1N и RelER1C - к виду L.rhamnosus.
Пример 2.
Идентификация штаммов Lactobacillus ramnosus.
Для штамм-специфической идентификации проводят амплификацию с геномной ДНК с использованием олигонуклеотидов, представленых в таблице 3. ПЦР продукты анализируют в 1% агарозном геле. Размер полученного фрагмента определяют с помощью ДНК-маркера GeneRulerTM 100+п.н. («Fermentas»).
Исследуемый штамм идентичен штамму L.ramnosus GG в случае наработки фрагментов при использовании олигонуклеотидов RelBR1N-RelBR1C. При использовании остальных олигонуклеотидов не должно быть ПЦР-продуктов.
Исследуемый штамм идентичен штамму L.ramnosus Lc705 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов RelBR2N-RelBR2C и RelBR3N-RelBR3C.
Исследуемый штамм идентичен штамму L.ramnosus HN001 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов RelBR1N-RelBR1C и RelBR2N-RelBR2C.
Исследуемый штамм идентичен штамму L.ramnosus LMS2-1 в случае наличия ПЦР-продуктов при использовании олигонуклеотидов RelBR2N-RelBR2C и RelBR3N-RelBR3C, а также при использовании олигонуклеотидов MazFR3N-MazFR3C и MazER3N- MazER3C.
Исследуемый штамм идентичен штамму L.ramnosus ATCC 8530 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов RelBR2N-RelBR2C.
Пример 3.
Идентификация штаммов лактобактерий, выделенных из микробиоты жителей России.
С использованием сконструированных праймеров для генов систем токсин-антитоксин mazEF и relBE проведена идентификация групп штаммов лактобактерий из коллекции лаборатории генетики микроорганизмов Института общей генетики им. Н.И.Вавилова РАН, выделенных из микробиоты практически здоровых людей Центрального региона России.
В коллекции обнаружены штаммы, принадлежащие к видам L. plantarum, L. rhamnosus, L. casei и L. helveticus.
На втором этапе проводили штаммовую идентификацию вида Z. rhamnosus. Исходя из композиции генов систем mazEF и relBE среди 15 штаммов L. rhamnosus не обнаружено ни одного идентичного, таким образом, данный подход позволяет идентифицировать штаммовую специфичность в пределах вида L. rhamnosus.
| Таблица 1 | ||||
| Распределение генов систем токсин-антитоксин mazEF и relBE в геномах секвенированных штаммов Lactobacillus ramnosus | ||||
| Система mazEF | Система relBE | |||
| Штаммы L.ramnosus | Наличие генов mazF - токсина | Наличие генов mazE - антитоксина | Наличие генов relE - токсина | Наличие генов relB - антитоксина |
| Штаммы из GenBank | ||||
| GG | mazF1 | relE1 | relB1 | |
| Lc705 | mazF1 | relE1 | relB2 | |
| mazF2 | mazE2 | relE2 | relB3 | |
| HN001 | mazF1 | relE1 | relB1 | |
| mazF3 | mazE3 | relE2 | relB2 | |
| LMS2-1 | mazF1 | relE1 | ||
| mazF2 | mazE2 | relE2 | relB2 | |
| mazF3 | mazE3 | relB3 | ||
| АТСС 8530 | mazF1 | relE1 | ||
| mazF2 | mazE2 | relE2 | relB2 |
| Таблица 2 | ||||
| Олигонуклеотиды для идентификации групп и отдельных видов лактобацилл | ||||
| Название олигонуклеотида | Структура олигонуклеотида 5'-3' | Место посадки | Название гена | Размер фрагмента, нуклеотиды |
| LacN | TCAGAATGAATCCAGAAAAGAAG | 29-51 | relB1 | 164 |
| LacC | CATTTGATCCAAATTATCCCATG | 192-170 | ||
| LhvIN | ATGGATTACAAAGATCAAACTTCG | 1-24 | rеlВ2 | 297 |
| LhvIC | TGAAAGAGTGGCTGAATGAAGAA | 297-275 | ||
| LpIN | AGCTGACATTAAGCGTGGTG | 9-28 | mazF | 320 |
| LpIC | ACGCATTATCAACTCGGCGCA | 328-308 | ||
| LcsN | ACCGGCCAGTACTTATTATTCA | 79-100 | mazF | 275 |
| LcsC | TATTATATGTTTTGGGCTTGGGCA | 376-353 | ||
| LcsIN | GTGTCCGTAAAACGCGGCG | 7-25 | mazF | 323 |
| LcsIC | GCCAACGCGCGGTCGACT | 329-312 | ||
| RelERIN | GCCCACCTCCCTGCCCCT | 3-20 | relE1 | 239 |
| RelERIC | AGCGGATCGCGTCTGGTTC | 241-222 |
| Таблица 3 | ||||
| Олигонуклеотиды для штаммовой идентификации лактобацилл вида Lactobacillus ramnosus | ||||
| Название олигонуклеотида | Структура олигонуклеотида 5'-3' | Место посадки | Название гена | Размер фрагмента, нуклеотиды |
| RelBR1N | GCAAAAGAATCCCGTATCCG | 3-23 | relB1 | 246 |
| RelBR1C | TCCTTCATCAAAGCACCAACA | 248-228 | ||
| RelBR2N | CGAATTATAGTGATTTCCGCCG | 11-32 | relВ2 | 245 |
| RelBR2C | GTCCGTTAACAACTCGTGCT | 255-236 | ||
| RelBR3N | GACCAAAAAATCCCGCATCAG | 3-23 | relВ3 | 245 |
| RelBR3C | CCTTCATCAAAGCATCGACAG | 247-227 | ||
| MazFR3N | ATGACTTAGTCAGCCTATACG | 11-31 | mazF3 | 489 |
| MazFRSC | TAGGCCGATCGTTTTTATTTTC | 499-478 | ||
| MazER3N | AGGAAAAGAAACGGGTCCAAG | 11-31 | mazE3 | 258 |
| MazER3C | GATCGTTGAGCCAATCAGCG | 268-249 |
Способ идентификации лактобацилл групп L. acidophilus, L. plantarum и L. casei, видов L. acidophilus, L. helveticus и L. rhamnosus и штаммов L. rhamnosus, отличающийся тем, что основан на комбинации и полиморфизме генов токсин-антитоксин суперсемейств RelBE и MazEF, и характеризующийся тем, что для идентификации проводят амплификацию геномных ДНК с использованием набора олигонуклеотидов, представленых в таблицах 2 и 3, ПЦР-продукты анализируют в агарозном геле, а размер полученного фрагмента определяют с помощью ДНК-маркера; исследуемый штамм относят к группе L. acidophilus в случае наработки фрагмента при использовании олигонуклеотидов LacN и LacC, при использовании остальных олигонуклеотидов не должно быть ПЦР-продуктов; исследуемый штамм относят к виду L. helveticus в случае наработки фрагмента при использовании олигонуклеотидов Lhv1N и Lhv1C; исследуемый штамм относят к группе L. plantarum в случае наработки фрагмента при использовании олигонуклеотидов LplN и LplC; исследуемый штамм относят к группе L. casei в случае наработки фрагмента при использовании олигонуклеотидов LcsN и LcsC; исследуемый штамм относят к виду L. casei в случае наработки фрагмента при использовании олигонуклеотидов Lcs1N и Lcs1C; исследуемый штамм относят к виду L. rhamnosus в случае наработки фрагмента при использовании олигонуклеотидов RelER1N и RelER1C; исследуемый штамм признают идентичным штамму L. ramnosus GG в случае наработки фрагментов при использовании олигонуклеотидов RelBR1N-RelBR1C, а при использовании остальных олигонуклеотидов не должно быть ПЦР-продуктов; исследуемый штамм признают идентичным штамму L. ramnosus Lc705 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов RelBR2N-RelBR2C и RelBR3N-RelBR3C; исследуемый штамм признают идентичным штамму L. ramnosus HN001 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов RelBR1N-RelBR1C и RelBR2N-RelBR2C; исследуемый штамм признают идентичным штамму L. ramnosus LMS2-1 в случае наличия ПЦР-продуктов при использовании олигонуклеотидов RelBR2N-RelBR2C и RelBR3N-RelBR3C, а также при использовании олигонуклеотидов MazFR3N-MazFR3C и MazER3N-MazER3C; исследуемый штамм признают идентичным штамму L. ramnosus ATCC 8530 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов RelBR2N-RelBR2C.