Способ анализа соматических мутаций в генах egfr, kras и braf с использованием lna-блокирующей мультиплексной пцр и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом)
Иллюстрации
Показать всеИзобретение относится к биотехнологии, а именно к способу анализа соматических мутаций в генах EGFR, KRAS и BRAF. Способ включает амплификацию фрагментов генов EGFR, KRAS и BRAF с помощью LNA-блокирующей мультиплексной «гнездовой» ПЦР, в которой в качестве матрицы для амплификации используется образец опухолевой ДНК. Получают одноцепочечный флуоресцентно меченый ПЦР-продукт. Получают биочип для идентификации соматических мутаций в генах EGFR, KRAS и BRAF. Проводят гибридизацию флуоресцентно меченого ПЦР-продукта на биочипе. Регистрируют и интерпретируют результаты гибридизации на биочипе. Предложенное изобретение позволяет провести анализ в короткий срок и с небольшими материальными затратами. 2 н. и 2 з.п. ф-лы, 2 ил., 4 табл., 4 пр.
Реферат
Область техники, к которой относится изобретение
Изобретение относится к области генетики, молекулярной биологии и медицины и касается способа определения чувствительности ряда онкологических заболеваний к таргетной терапии посредством анализа соматических мутаций в генах EGFR, KRAS и BRAF с помощью технологии LNA-блокирующей мультиплексной ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом).
Уровень техники
Известно большое количество способов определения соматических мутаций, в которых используются различные инструменты для анализа уникальной нуклеотидной последовательности ДНК человека. Условно среди них можно выделить шесть групп:
1) Ферментативные подходы:
полиморфизм длин амплифицированных фрагментов (AFLP);
полиморфизм длин рестрикционных фрагментов (RFLP);
расщепление клевазой (CFLP);
расщепление резольвазой (EMD);
анализ, основанный на лигазной реакции (LDR, LCR);
ПЦР, обогащенная мутантной последовательностью;
PNA-блокирующая ПЦР в реальном времени (PNA-clamp PCR);
LNA-блокирующая ПЦР в реальном времени (LNA-clamp PCR);
ПЦР с прямой терминацией синтеза (DT-PCR);
аллель-специфическая ПЦР (AS-PCR, PCR-SSP);
SMart-амплификация;
PNA-LNA-блокирующая ПЦР в реальном времени (PNA-LNA-clamp PCR);
секвенирование по Сенгеру;
высокопроизводительное секвенирование (NGS).
2) Химические методы:
химическое расщепление гетеродуплексов;
химическое лигирование.
3) Методы, основанные на различной электрофоретической подвижности полиморфных участков ДНК:
анализ конформации одноцепочечных фрагментов (SSCP);
гетеродуплексный анализ (НА);
разделение продуктов амплификации посредством капиллярного электрофореза.
4) Детекция на твердой фазе:
гибридизация на олигонуклеотидных матрицах;
оптико-волоконный ДНК-гибридизационный анализ;
элонгация иммобилизованных праймеров (минисеквенирование);
пиросеквенирование.
5) Хроматографические методы:
высокоэффективная жидкостная хроматография (HPLC).
6) Физические методы:
масс-спектрометрия;
резонансное тушение флуоресценции (FRET);
люминесценция, зависящая от локального окружения.
Наиболее распространенными из представленных методов являются:
1. Секвенирование амплифицированных фрагментов ДНК по Сенгеру.
Frequent epidermal growth factor receptor gene mutations in malignant pleural effusion of lung adenocarcinoma / S-G. Wu, C-H. Gow, C-J. Yu et al. // Eur Respir J. - 2008. - Vol. 32. - P. 924-930.
Detection of Epidermal Growth Factor Receptor Mutation in Transbronchial Needle Aspirates of Non-Small Cell Lung Cancer / A. Horiike, H. Kimura, K. Nishio et al. // Chest. - 2007. - Vol. 131. - P. 1628-1634.
Activating mutations in the epidermal growth factor receptor underlying responsiveness of non-small-cell lung cancer to gefitinib / T.J. Lynch, D.W. Bell, R. Sordella et al. // N Engl J Med. - 2004. - Vol. 350. - P. 2129-2139.
A comparison of Direct sequencing, Pyrosequencing, High resolution melting analysis, TheraScreen D×S, and the K-ras StripAssay for detecting KRAS mutations in non small cell lung carcinomas / S. Jancik, J. Drabek, J. Berkovcova et al. // Journal of Experimental & Clinical Cancer Research - 2012. - Vol. 31. - 79.
Sequencing analysis of BRAF mutations in human cancers / R. Wooster, A.P. Futreal, M.R. Stratton // Methods Enzymol. - 2006. - Vol. 407. - P. 218-224.
2. Анализ конформации одноцепочечных фрагментов ДНК (SSCP).
EGFR gene amplification is related to adverse clinical outcomes in cervical squamous cell carcinoma, making the EGFR pathway a novel therapeutic target / K. Iida, K. Nakayama, M.T. Rahman et al. // Br J Cancer. - 2011. - Vol. 105. - P. 420-427.
EGFR Mutations in Non-Small-Cell Lung Cancer: Analysis of a large series of cases and development of a rapid and sensitive method for diagnostic screening with potential implications on pharmacologic treatment / A. Marchetti, C. Martella, L. Felicioni et al. // J Clin Oncol. - 2005. - Vol. 23. - 857-865.
High Prevalence of BRAF Mutations in Thyroid Cancer: Genetic Evidence for Constitutive Activation of the RET/PTC-RAS-BRAF Signaling Pathway in Papillary Thyroid Carcinoma / E.T. Kimura, M.N. Nikiforova, Z. Zhu et al. // Cancer Res. - 2003. - Vol. 63. - P. 1454-1457.
3. Аллель-специфичная ПЦР (AS-PCR).
Two allele-specific PCR assays for screening epidermal growth factor receptor gene hotspot mutations in lung adenocarcinoma / R. Dahse, A. Berndt, A.K. Dahse et al. // Mol Med Report. - 2008. - Vol. 1. - P. 45-50.
PCR-based testing for therapy-related EGFR mutations in patients with non-small cell lung cancer / R. Dahse, A. Berndt, H. Kosmehl // Anticancer Res. - 2008. - Vol. 28. - 2265-2270.
KRAS mutations are associated with inferior clinical outcome in patients with metastatic colorectal cancer, but are not predictive for benefit with cediranib / J.C. Smith, L. Brooks, P.M. Hoff et al. // Eur J Cancer. - 2013. - Vol. 49. - P. 2424-2432.
4. Гибридизация с биочипами.
Combined detection of p53, p16, Rb, and EGFR mutations in lung cancer by suspension microarray / Y. Ye, D. Wang, C. Su et al. // Genet Mol Res. - 2009. - Vol. 8. - P. 1509-1518.
The KRAS StripAssay for detection of KRAS mutation in Egyptian patients with colorectal cancer (CRC): a pilot study / Y. Abd El Kader, G. Emera, E. Safwat et al. // J Egypt Natl Canc Inst. - 2013. - Vol. 25. - P. 37-41.
5. SMart-амплификация.
Correlation between computed tomography findings and epidermal growth factor receptor and KRAS gene mutations in patients with pulmonary adenocarcinoma / M. Sugano, K. Shimizu, T. Nakano et al. // Oncol Rep. - 2011. - Vol. 26. - P. 1205-1211.
Rapid Detection of Epidermal Growth Factor Receptor Mutations in Lung Cancer by the SMart-Amplification Process / K. Hoshi, H. Takakura, Y. Mitani et al. // Clin Cancer Res. - 2007. - Vol. 13. - P. 4974-4983.
6. PNA-блокирующая ПЦР в реальном времени.
PNA-mediated Real-Time PCR Clamping for Detection of EGFR Mutations / J. Choi, M. Cho, M. Oh et al. // Bull. Korean Chem. Soc. - 2010. - Vol. 31. - P. 3525-3529.
7. LNA-блокирующая ПЦР в реальном времени.
Rebiopsy of Lung Cancer Patients with Acquired Resistance to EGFR Inhibitors and Enhanced Detection of the T790M Mutation Using a Locked Nucleic Acid-Based Assay / G.J. Riely, S.B. Solomon, M.F. Zakowski et al. // Clin Cancer Res. - 2011. - Vol. 17. - P. 1169-1180.
8. PNA-LNA-блокирующая ПЦР в реальном времени.
Reliable detection of rare mutations in EGFR gene codon L858 by PNA-LNA PCR clamp in non-small cell lung cancer / M. Skronski, J. Chorostowska-Wynimko, E. Szczepulska et al. // Adv Exp Med Biol. - 2013. - Vol. 756. - P. 321-331.
First-Line Gefitinib for Patients With Advanced Non-Small-Cell Lung Cancer Harboring Epidermal Growth Factor Receptor Mutations Without Indication for Chemotherapy / A. Inoue, K. Kobayashi, K. Usui et al. // J Clin Oncol. - 2009. - Vol. 27. - P. 1394-1400.
Peptide nucleic acid-locked nucleic acid polymerase chain reaction clamp-based detection test for gefitinib-refractory T790M epidermal growth factor receptor mutation / H. Miyazawa, T. Tanaka, Y. Nagai et al. // Cancer Sci. - 2008. - Vol. 99. - P. 595-600.
Genetic Heterogeneity of the Epidermal Growth Factor Receptor in Non-Small Cell Lung Cancer Cell Lines Revealed by a Rapid and Sensitive Detection System, the Peptide Nucleic Acid-Locked Nucleic Acid PCR Clamp // Y. Nagai, H. Miyazawa, Huqun et al. // Cancer Res. - 2005. - Vol. 65. - P. 7276-7282.
9. Капиллярный электрофорез.
EGFR mutations in lung adenocarcinomas: clinical testing experience and relationship to EGFR gene copy number and immunohistochemical expression / A.R. Li, D. Chitale, G.J. Riely et al. // J Mol Diagn. - 2008. - Vol. 10. - P. 242-248.
10. ПЦР, обогащенная мутантной последовательностью.
Detection of EGFR gene mutation in lung cancer by mutant-enriched polymerase chain reaction assay / H. Asano, S. Toyooka, M. Tokumo et al. // Clin Cancer Res. - 2006. - Vol. 12. - P. 43-48.
11. Анализ кривых плавления высокого разрешения.
High resolution melting analysis for rapid and sensitive EGFR and KRAS mutation detection in formalin fixed paraffin embedded biopsies / H. Do, M. Krypuy, P.L. Mitchell et al. // BMC Cancer. - 2008. - Vol. 8. 142.
High-Resolution Melting Analysis for Rapid Detection of KRAS, BRAF, and PIK3CA Gene Mutations in Colorectal Cancer / L. Simi, N. Pratesi, M. Vignoli et al. // Am J Clin Pathol. - 2008. - Vol. 130. - P. 247-253.
Метод 1 широко распространен и является достаточно информативным. Он предоставляет полную информацию о последовательности ДНК в исследуемом локусе и позволяет определять мутации de novo. Однако он не обладает достаточной чувствительностью для использования в клинической практике при анализе соматических мутаций (для данного метода образец должен содержать не менее 25% мутантной ДНК), отличается низкой производительностью и необходимостью в дорогостоящем оборудовании и реактивах. Кроме того, для дальнейшей расшифровки хроматограмм необходима высокая квалификация персонала, что препятствует использованию данного метода в широкомасштабных исследованиях.
Метод 2 сложен в исполнении и трактовке результатов, дает лишь косвенную информацию о нуклеотидной последовательности и не подлежит автоматизации.
Метод 3 распространен достаточно широко, однако требует постановки независимых параллельных ПЦР-реакций, соответствующих количеству анализируемых однонуклеотидных замен. Для оценки результатов также требуется постановка гель-электрофореза, что резко снижает его ценность для широкомасштабного генотипирования.
Метод 4 является наиболее перспективным для проведения одновременного генотипирования большого количество генетических локусов, однако, на данный момент отсутствуют дешевые тест-системы на основе данного метода, которые позволяли бы за один анализ определять большинство клинически важных мутаций в исследуемом гене.
Метод 5 быстр в исполнении, но требует постановки параллельных реакций по количеству анализируемых мутаций. К недостаткам этого метода можно отнести и то, что с его помощью можно выявлять только известные мутации.
Методы 6-8 также требуют постановки нескольких независимых реакций для каждого пациента. Эти методы позволяют выявить только факт наличия/отсутствия мутации в образце, но не дает информации о нуклеотидной последовательности анализируемых локусов. Если в одной пробирке анализируется присутствие только одной мутации, данный подход оправдан, однако, при одновременном анализе в одной пробирке сразу нескольких типов мутаций (например, несколько различных вариантов делеций в 19 экзоне EGFR), этот метод уже недостаточно информативен.
Метод 9 позволяет с высокой точностью и достоверностью выявлять делеций, однако дает лишь косвенное представление об их нуклеотидной структуре. При определении однонуклеотидных замен требуются дополнительные манипуляции (в частности использование рестриктаз), что существенно увеличивает время анализа и снижает его удобство для рутинного использования в клинической практике.
Метод 10 обладает достаточно высокой чувствительностью, но требует большого количества манипуляций. Кроме того, он также требует постановки нескольких параллельных реакций для каждого пациента.
Метод 11 позволяет с высокой точностью и достоверностью диагностировать мутации в гене EGFR, однако оборудование для него достаточно дорого.
Таким образом, в настоящий момент существует острая потребность в разработке способа диагностики значительного числа мутаций в гене EGFR, который бы выгодно отличался от известных решений простотой проведения анализа и низкой стоимостью.
В настоящее время существует множество патентов на методы анализа мутаций в генах EGFR, KRAS, BRAF, ассоциированных с чувствительностью опухоли к таргетной терапии. Как правило, подобные методы ориентированы на определение наличия/отсутствия мутаций в исследуемом образце и не определяют индивидуальную структуру генетического изменения (например, китайский патент CN 101565742). В случае генотипирования точечных мутаций (например, L858R и Т790М EGFR) такой подход может быть вполне оправдан, однако при анализе делеций в 19 экзоне EGFR, которые не обладают консервативной структурой, исследователь не получает информации о том, какая именно делеция присутствует в клиническом образце. В недавних исследованиях было показано, что различные делеции в 19 экзоне гена EGFR по-разному влияют на эффективность противоопухолевой терапии.
Многие патенты, описывающие методы анализа мутаций в гене EGFR, включают в себя только маркеры, ассоциированные с повышенной чувствительностью опухоли к терапии, как, например, в патентных заявках WO 2011035538 и WO 2009129693, но не включают маркеры резистентности.
В китайском патенте CN 101041850 и американской патентной заявке WO 2006086777, напротив, предлагается метод анализа только мутации Т790М EGFR, которая является маркером резистентности опухоли к анти-EGFR терапии. В патентной заявке WO 2006086777 описывается метод, который заключается в амплификации нуклеотидной последовательности гена EGFR, включающей 790 кодон, обработке продуктов амплификации рестриктазой MaHI, которая избирательно расщепляет только последовательность, несущую однонуклеотидную замену в данном кодоне, и последующую детекцию продуктов рестрикции с помощью капиллярного электрофореза (прибор ABI 3100 Avant, Applied Biosystems, Foster City, California, United States). Анализ только этой мутации недостаточен для выбора таргетной противоопухолевой терапии при немелкоклеточном раке легкого. Но данный анализ может быть полезен при наблюдении за ходом лечения пациента анти-EGFR препаратами, так как эта соматическая мутация часто возникает в опухоли уже в процессе лечения.
В патенте CN 1687457 описан метод определения мутаций в 18-21 экзонах гена EGFR, включающий в себя амплификацию с помощью HotStarTaq Master Mix и последующее секвенирование в двух направлениях. Он представляет собой модификацию метода 1. Для использования этого метода требуется дорогостоящее оборудование и высококвалифицированный персонал, что делает его малоподходящим для использования в рутинной клинической диагностике.
В японском патенте JP 2007202546 описаны праймеры, избирательно амплифицирующие только мутантную последовательность гена EGFR. Для визуализации продуктов амплификации предлагается использовать электрофорез в агарозном геле (метод 3).
В патенте GB 2424886 представлен набор праймеров для амплификации мутаций или полиморфизмов в гене EGFR. Эти праймеры могут применяться для анализа гена EGFR посредством методики ARMS (amplification-refractory mutation system, амплификация рефракторной мутационной системы). Этот метод нашел довольно широкое применение для детекции мутаций при ряде наследственных заболеваний, однако сложности в подборе праймеров и в выборе оптимального режима ПЦР ограничивают его широкое применение.
Множество патентов предлагают различные варианты постановки ПЦР в реальном времени. В американском патенте US 2013095491 и в европейском ЕР 2540838 представлена методика определения мутаций с помощью аллель-специфичной ПЦР в гене BRAF. Описания методов на основе аллель-специфичной ПЦР для определения мутаций в гене EGFR содержатся в китайских патентах CN 102021228 и CN 101565742 (генотипирование проводится в 19 и 21 экзонах). В китайских патентах CN 101434985, CN 101899496, CN 102041313 представлены наборы реагентов на основе метода PNA-блокирующей ПЦР в реальном времени для обнаружения мутаций в различных кодонах KRAS. Такое большое количество тест-систем, основанных на методе ПЦР в реальном времени, объясняется тем, что данный метод достаточно прост в освоении, часто не требует дополнительных навыков и дорогостоящих реактивов, но для его осуществления требуется приобретение дорогостоящего оборудования. Также, применение его в клинической практике при одновременном анализе множества мутаций является достаточно трудоемким процессом.
В китайских патентах CN 102443140, CN 101070538 и CN 101092644 предлагается метод амплификации фрагментов гена EGFR.
В российском патенте RU 2454464 представлен способ генотипирования мутаций в гене EGFR с помощью аллель-специфичной ПЦР с визуализацией продуктов амплификации с помощью электрофореза в полиакриламидном геле (метод 3). Данный метод обладает высокой чувствительностью, специфичностью, не требует специфического оборудования, дорогостоящих реактивов и высокой квалификации персонала. Но постановка полиакриламидного гель-электрофореза значительно снижает потенциал клинического использования данного подхода.
С помощью различных способов амплификации анализируются мутации и в других генах. В патентной заявке WO 2005071109 с помощью сочетания SSCP и электрофореза анализируются мутации в генах MSH, MLH и мутация Т1796А (V600E) в гене BRAF. В патентной заявке WO 2012068562 описан способ генотипирования мутаций в гене BRAF у больных меланомой с помощью секвенирования. В патентной заявке WO 2005066346 с помощью метода SSCP детектируются мутации в 599 кодоне гена BRAF и в 12, 13 кодонах гена KRAS. Данный способ генотипирования обладает всеми вышеперечисленными достоинствами и недостатками.
Опубликованы несколько патентов и патентных заявок, в которых приведены различные технологии определения мутаций посредством гибридизации с биочипами (метод 4). В корейском патенте KR 101133 242 описан способ гибридизации фрагментов гена EGFR с зондами, расположенными на наночастицах золота, серебра или платины. Данный метод обладает высокой точностью, но и высокой стоимостью. В американском патенте US 7833757 приведен способ анализа мутаций в генах АРС, KRAS, beta-catenin, BRAF при раке толстого кишечника посредством гибридизации с олигонуклеотидами, иммобилизованными на микрочипе и последующей детекции с использованием системы биотин-стрептавидин. В патентной заявке WO 2009129693 описан метод анализа 19 и 21 экзонов гена EGFR с помощью технологии жидкостных биочипов. Анализ обладает высокой точностью и высокой скоростью проведения. В китайском патенте CN 102443626 отражена методика наиболее полного генотипирования при раке легкого. С помощью сочетания технологии флуоресцентной ПЦР и гибридизации с зондами анализируются мутации в генах EGFR, KRAS, BRAF, а также транслокация EML4-ALK. Анализ обладает хорошей чувствительностью, специфичностью и высокой скоростью.
Поскольку все имеющиеся в настоящее время методы определения соматических мутаций в генах EGFR, KRAS, BRAF обладают теми или иными недостатками, существует реальная потребность в создании простого, недорогого и специфичного метода для выявления соматических мутаций, ассоциированных с чувствительностью опухоли к таргетной терапии, с целью дальнейшего внедрения в любую стандартную клиническую лабораторию.
Такой способ обеспечивается настоящим изобретением.
Раскрытие изобретения
Сущность изобретения заключается в обеспечении способа определения соматических мутаций в генах EGFR, KRAS и BRAF, влияющих на чувствительность опухоли к таргетной терапии. Данный способ позволяет проводить детекцию точечных соматических мутаций G12S, G12A, G12C, G12D, G12R, G12V, G13D, G13V, G13A, G13C, G13R, Q61L, Q61H гена KRAS, V600E гена BRAF, G719A, G719C, G719S, T790M, L858R гена EGFR и делеций в 19 экзоне EGFR: E746-A750del (2 варианта: c. 2235-2249del, c. 2236-2250del), E746-T751del ins A, E746-T751del ins V, E746-S752del ins V, L747-A750del ins P, L747-T751del ins P, L747-T751del, L747-P753del ins S, E746-S752del ins V, E746-A750del insQP, E746-T751del ins VA, E746-P753del ins VS.
Потенциальные эффекты влияния соматических мутаций в генах EGFR, KRAS и BRAF на эффективность лечения таргетными противоопухолевыми препаратами представлены в табл. 1.
Список литературы к Таблице 1
1. Predictive and prognostic impact of epidermal growth factor receptor mutation in non-small-cell lung cancer patients treated with gefitinib / S.W. Han, T.Y. Kim, P.G. Hwang, et al. // J Clin Oncol. - 2005. - Vol. 23. - P. 2493-2501.
2. Activating mutations in the epidermal growth factor receptor underlying responsiveness of non-small-cell lung cancer to gefitinib / T.J. Lynch, D.W. Bell, R. Sordella et al. // N Engl J Med. - 2004. - Vol. 350. - P. 2129-2139.
3. Mutations in the tyrosine kinase domain of the EGFR gene associated with gefitinib response in non-small-cell lung cancer / R. Rosell, Y. Ichinose, M. Taron et al. // Lung Cancer. - 2005. - Vol. 50. - P. 25-33.
4. Activating mutations in the tyrosine kinase domain of the epidermal growth factor receptor are associated with improved survival in gefitinib-treated chemorefractory lung adenocarcinomas / M. Taron, Y. Ichinose, R. Rosell et al. // Clin Cancer Res. - 2005. - Vol. 11. - P. 5878-5885.
5. EGFR mutation and resistance of non-small-cell lung cancer to gefitinib / S. Kobayashi, T.J. Boggon, T. Dayaram et al. - New Eng. J Med. - 2005. - Vol. - 352. - P. 786-792.
6. Inherited susceptibility to lung cancer may be associated with the T790M drug resistance mutation in EGFR / D.W. Bell, I. Gore, R.A. Okimoto et al. // Nat Genet. - 2005. - Vol. 37. - P. 1315-1316.
7. EGFR mutation and response of lung cancer to gefitinib / S. Toyooka, K. Kiura, T. Mitsudomi // N Engl J Med. - 2005. - Vol. 352. - author reply 2136.
8. Response to erlotinib and prognosis for patients with de novo epidermal growth factor receptor (EGFR) T790M mutations / G.J. Riely, H.A. Yu, M.E. Arcila et al. // J Clin Oncol. - 2013. - Vol. 31. - Abstr 8018.
9. Biomarker analyses and final overall survival results from a phase III, randomized, open-label, first-line study of gefitinib versus carboplatin/paclitaxel in clinically selected patients with advanced non-small-cell lung cancer in Asia (IPASS) / M. Fukuoka, Y.L. Wu, S. Thongprasert et al. // J Clin Oncol. - 2011. - Vol. 29. - P. 2866-2874.
10. Clinicopathologic Significance of the Mutations of the Epidermal Growth Factor Receptor Gene in Patients with Non-Small Cell Lung Cancer / Y. Tomizawa, H. Iijima, N. Sunaga et al. // Clin Cancer Res. - 2005. - vol. 11. - P. 6816-6822.
11. EGFR mutations in lung cancer: correlation with clinical response to gefitinib therapy / J.G. Paez, P.A. Janne, J.C. Lee et al. // Science. - 2004. - Vol. 304. - P. 1497-1500.
12. EGF receptor gene mutations are common in lung cancers from ′never smokers′ and correlate with sensitivity of tumors to gefitinib (Iressa) and erlotinib (Tarceva) / W. Pao, V. Miller, M.F. Zakowski // Proc Natl Acad Sci USA. - 2004. - Vol. 101. - P. 13306-13311.
13. Biological and clinical significance of KRAS mutations in lung cancer: an oncogenic driver that contrasts with EGFR mutation / K. Suda, K. Tomizawa, T. Mitsudomi // Cancer Metastasis Rev. - 2010. - Vol. 29. - P. 49-60.
14. KRAS mutation status is predictive of response to cetuximab therapy in colorectal cancer / A. Lièvre, J.B. Bachet, D. Le Corre et al. // Cancer Res. - 2006. - Vol. 66. - P. 3992-3995.
15. A patient with BRAF V600E lung adenocarcinoma responding to vemurafenib / O. Gautschi, С Pauli, K Strobel et al. // J Thorac Oncol. - 2012. - Vol. 7(10): e23-24.
16. Molecular characterization of acquired resistance to the BRAF inhibitor dabrafenib in a patient with BRAF-mutant non-small-cell lung cancer / C.M. Rudin, K. Hong, M. Streit et al. // J Thorac Oncol. - 2013. - Vol. 8. - e41-42. doi: 10.1097/JTO.0b013e31828bb1b3.
17. Analysis of PTEN, BRAF, and EGFR status in determining benefit from cetuximab therapy in wild-type KRAS metastatic colon cancer / P. Laurent-Puig, A. Cayre, G. Manceau et al. // J Clin Oncol. - 2009. - Vol. 27. - P. 5924-5930.
18. Clinical development of dabrafenib in BRAF mutant melanoma and other malignancies / G.T. Gibney, J.S. Zager // Expert Opinion on Drug Metabolism & Toxicology. - 2013. - Vol. 9(7). - P. 893-899. doi: 10.1517/17425255.2013.794220.
19. B-Raf and the inhibitors: From bench to bedside / T. Huang, M. Karsy, J. Zhuge et al. // Journal of Hematology & Oncology. - 2013. - Vol. 6. - P. 30. doi: 10.1186/1756-8722-6-30.
20. Improved survival with vemurafenib in melanoma with BRAF V600E mutation / P.B. Chapman, A. Hauschild, C. Robert et al. // N Engl J Med. - 2011. - Vol. 364. - P. 2507-2516. doi: 10.1056/NEJMoa1103782.
21. http://www.fda.gov/Drugs/InformationOnDrugs/ApprovedDrugs/ucm354478.htm
Основными аспектами данного изобретения являются LNA-блокирующая мультиплексная «гнездовая» ПЦР и биочип, содержащий набор иммобилизованных дифференцирующих олигонуклеотидов.
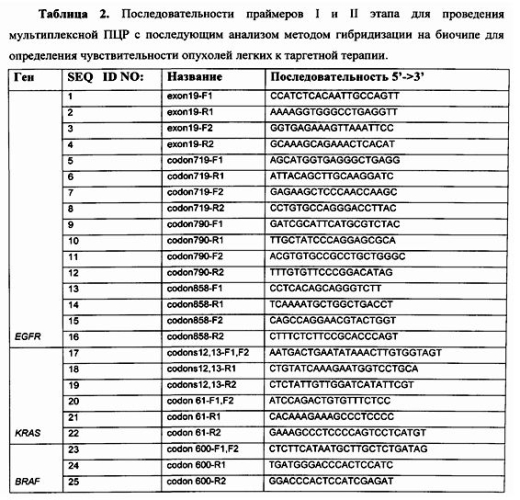
Первым важным аспектом изобретения являются праймеры для амплификации ДНК-локусов исследуемых генов EGFR, KRAS, BRAF, набор которых используется для получения изучаемых флуоресцентно меченых фрагментов ДНК в требуемом количестве. Последовательности праймеров приведены в табл. 2 и Перечне последовательностей (SEQ ID NO: 1-25).
Вторым важным аспектом изобретения являются LNA-олигонуклеотиды, обеспечивающие преимущественную амплификацию ДНК-локусов, содержащих соматические мутации. Преимущественная амплификация мутантных ДНК-локусов происходит в результате того, что LNA-олигонуклеотиды узнают и специфически связываются с фрагментами ДНК «дикого типа», препятствуя их амплификации. Последовательности LNA-олигонуклеотидов приведены в табл. 3 и Перечне последовательностей (SEQ ID NO: 26-32).
Третьим важным аспектом изобретения является биочип, содержащий набор иммобилизованных дифференцирующих олигонуклеотидов, последовательности которых приведены в табл. 4 и Перечне последовательностей (SEQ ID NO: 33-72). Дифференцирующие олигонуклеотиды иммобилизуются в ячейках гидогелевого микрочипа, как описано в патенте [Композиция для иммобилизации биологических макромолекул в гидрогелях, способ приготовления композиции, биочип, способ проведения ПЦР на биочипе / А.Д. Мирзабеков, А.Ю. Рубина, С.В. Паньков, и др. // Патент RU 2206575 C2. - 2003] в концентрации 200-400 мкМ. Схема расположения ячеек биочипа для анализа соматических мутаций в гене EGFR, KRAS и BRAF приведена на Фиг. 1.
Набор праймеров и LNA-олигонуклеотидов используется на стадии предварительной амплификации ДНК-локусов посредством проведения мультиплексной LNA-блокирующей «гнездовой» ПЦР для подготовки ДНК-мишени к гибридизации на биочипе. На первом этапе, благодаря использованию LNA-блокирующей мультиплексной ПЦР, проходит преимущественная амплификация ДНК-локусов, содержащих соматические мутации в целевых последовательностях генов EGFR, KRAS и BRAF (если образец содержит мутации). На втором этапе с амплифицированного фрагмента нарабатывается одноцепочечная ДНК с одновременным введением флуоресцентной метки. Далее проводится гибридизация флуоресцентно меченых фрагментов ДНК, полученных после проведения второго этапа ПЦР, с иммобилизованными в ячейках геля олигонуклеотидами, расположенными на пластиковой подложке. После проведения гибридизации и отмывки биочипа проводится анализ полученной флуоресцентной картины, на основании которого делается вывод о генотипе в исследуемом образце. Пример гибридизационной картины приведен на Фиг. 2.
Осуществление изобретения
Задача настоящего изобретения состоит в создании способа экспресс-анализа, позволяющего выявлять соматические мутации в генах EGFR, KRAS и BRAF.
Для обеспечения оптимального состава и структуры ПЦР-праймеров, используемых в протоколе мультиплексной ПЦР, необходимы такие праймеры, которые не образуют между собой высокоэнергетических внутренних структур (шпильки и дуплексы), обеспечивают специфичную амплификацию необходимого количества продукта и при этом подобраны таким образом, чтобы их отжиг на мишени происходил при одинаковой для всех температуре. Для обеспечения сбалансированной эффективной амплификации исследуемых образцов также должны быть оптимизированы такие параметры как концентрация MgCl2 и ПЦР-праймеров в ПЦР-смеси, соотношение прямых и обратных праймеров, количество циклов амплификации на обоих этапах, время элонгации, денатурации и отжига праймеров на каждом этапе. В результате проведенной работы подобраны праймеры 1 и 2 этапа, которые позволяют осуществлять эффективную наработку выбранных фрагментов генов EGFR, KRAS и BRAF.
Для обеспечения оптимального состава и структуры LNA-олигонуклеотидов, используемых в протоколе LNA-блокирующей мультиплексной ПЦР, необходимы такие LNA-олигонуклеотиды, которые не образуют между собой и с праймерами высокоэнергетических внутренних структур (шпильки и дуплексы), обеспечивают специфичное связывание с целевой последовательностью и при этом подобраны таким образом, чтобы при условиях ПЦР они образовывали с ДНК-мишенью стабильные совершенные дуплексы и не образовывали несовершенные дуплексы. В результате проведенной работы подобраны LNA-олигонуклеотиды, позволяющие осуществлять преимущественную наработку выбранных фрагментов генов EGFR, KRAS и BRAF, несущих соматические мутации.
Олигонуклеотиды для иммобилизации на биочипе для предсказания эффективности лечения пациентов таргетными препаратами подбираются таким образом, чтобы идентифицировать все выбранные для анализа соматические мутации в генах EGFR, KRAS и BRAF.
Олигонуклеотиды должны соответствовать следующим критериям:
1) Дискриминирующий зонд должен обладать высокой специфичностью к выбранному для анализа мутантному локусу, который представляет собой участок гена, амплифицированный с помощью ПЦР, с включенной флуоресцентной меткой.
2) Вариабельный нуклеотид должен находиться в серединной области зонда, поскольку такое положение позволяет добиться большей дискриминации между совершенными и несовершенными дуплексами.
3) Выбранные олигонуклеотиды не должны содержать высокостабильных вторичных структур, наличие которых может приводить к снижению эффективности гибридизации.
На биочипе для определения соматических мутаций в генах EGFR, KRAS и BRAF иммобилизовано 40 высокоспецифичных дифференцирующих олигонуклеотидных зондов (табл. 4 и Перечень последовательностей (SEQ ID NO: 33-72)), структура которых обеспечивает связывание только с полностью комплементарными ДНК-мишенями, что обуславливает яркий флуоресцентный сигнал в соответствующих им ячейках биочипа и дает наиболее четкие картины распределения гибридизационных сигналов. Неспецифическое связывание сведено к минимуму, что практически исключает ложноположительное «срабатывание» ячеек.
Так как при амплификации интересующих нас ДНК-локусов используется LNA-блокирующая ПЦР, ячейки, содержащие зонды с последовательностью «дикого типа», может иметь крайне низкий уровень флуоресцентного сигнала.
Приведем последовательность анализа с использованием данного метода. Амплификация ПЦР-продуктов для последующей гибридизации на биочипе проводится посредством «гнездовой» мультиплексной LNA-блокирующей ПЦР в два этапа, при этом в качестве матрицы для проведения реакции используют образец ДНК. Амплификацию каждого образца ДНК проводят в двух пробирках: в первой пробирке проводится амплификация фрагментов гена EGFR, во второй - фрагментов генов KRAS и BRAF.
ПЦР может быть проведена с использованием любого вида термостабильной полимеразы (Taq-полимераза, Tth-полимераза, Tfl-полимераза, Pfu-полимераза, Vent-полимераза, DeepVent-полимераза и т.п. коммерчески доступные ферменты, выделенные из термофилов), работающей в соответствующем буфере. Для построения новой цепи в буфер добавляется смесь дНТФ (дАТФ, дГТФ, дЦТФ, дТТФ) в принятых концентрациях, при этом вместо дТТФ может быть использован дУТФ. Для проведения ПЦР могут быть использованы готовые коммерчески доступные наборы, содержащие все необходимые компоненты за исключением праймеров.
На первом этапе проходит LNA-блокирующая амплификация ДНК-локусов генов EGFR, KRAS и BRAF с преимущественной наработкой мутантных фрагментов (если образец содержит мутацию). Продукт первого этапа ПЦР используют в качестве матрицы на втором этапе, который проводят в реакционной смеси того же состава, но не добавляют LNA-олигонуклеотиды и добавляют избыток одного из праймеров для получения избытка флуоресцентно меченого одноцепочечного ПЦР-продукта, способного к гибридизации с аллель-специфичными ДНК-зондами на биочипе. В качестве флуоресцентной метки используется дезоксинуклеотидтрифосфат, а именно Су5-дУТФ, который встраивается в de novo синтезируемую ДНК-цепь. В качестве флуоресцентной метки также может быть использован любой флуорохром без ограничения (например, FITC, Texas red, Су-3 и т.д.), а также биотин.
Праймеры, LNA-олигонуклеотиды и олигонуклеотидные зонды синтезируют с использованием различных химических подходов, таких как фосфодиэфирный метод, гидрофосфорильный метод и т.д., при этом наиболее распространенным в настоящее время является фосфоамидитный метод синтеза. Синтез праймеров осуществляют, используя автоматические ДНК/РНК синтезаторы, например производства фирмы «Applied Biosystems» (США).
При изготовлении биочипа могут быть использованы олигонуклеотиды, несущие по 5′- или 3′-концу активную группу, обеспечивающую иммобилизацию. Модификация олигонуклеотида для введения активной группы может быть осуществлена как в автоматическом режиме при синтезе с использованием широкого спектра коммерчески доступных модификаторов, так и постсинтетически в ручном режиме. Например, при синтезе олигонуклеотидных зондов с помощью 3′-Ammo-Modifier C7 CPG 500 («Glen Research», США) на 3′-конец олигонуклеотидов вводится спейсер со свободной аминогруппой, используемый для последующей иммобилизации олигонуклеотида на биочипе.
Для проведения мультиплексной LNA-блокирующей «гнездовой» ПЦР используют праймеры SEQ ID NO: 1-25 и LNA-олигонуклеотиды SEQ ID NO: 26-32, приведенные в Перечне последовательностей в табл. 2 и 3.
В мультиплексных реакциях I этапа используют следующие праймеры и LNA-олигонуклеотиды: первая мультиплексная реакция (SEQ ID NO: 1, 2, 5, 6, 9, 10, 13, 14, 26-29), вторая мультиплексная реакция (SEQ ID NO: 17, 18, 20, 21, 23, 24, 30-32).
В мультиплексной реакции II этапа используют следующие праймеры: первая мультиплексная реакция (SEQ ID NO: 3, 4, 7, 8, 11, 12, 15, 16), вторая мультиплексная реакция (SEQ ID NO: 17, 19, 20, 22, 23, 25).
Далее проводится гибридизация флуоресцентно меченых фрагментов ДНК, полученных после проведения второго этапа ПЦР, с иммобилизованными в ячейках геля олигонуклеотидами, которые представляют собой участки целевых последовательностей генов EGFR, KRAS и BRAF, и являются комплементарными последовательности «дикого типа» или последовательности, содержащей мутации.
Перед постановкой гибридизации ампликон денатурируют путем прогрева готовой гибридизационной смеси при 95°C в течение 5 мин с последующим быстрым охлаждением на льду в течение 2 мин. Гибридизация может быть проведена в любом известном специалисту в данной области гибридизационном буфере, например в гуанидиновом или SSPE-буфере. Типичное время гибридизации составляет 12-14 ч при 37°C. Анализ генотипов и аллелей в исследуемом образце проводится с учетом расположения олигонуклеотидных зондов на биочипе, схема которого позволяет определить, какие соматические мутации присутствуют в том или ином образце.
Анализируемый фрагмент ДНК образует совершенные гибридизационные дуплексы только с полностью комплементарными ему олигонуклеотидами. Если последовательность анализируемой ДНК полностью комплементарна последовательности зонда, то образуется стабильный совершенный дуплекс (детектируется сигнал флуоресценции). В случае если искомого фрагмента нет или в нем находится некомплементарное основание, стабильного дуплекса не образуется (сигнал флуоресценции отсутствует). Дискриминацию совершенных и несовершенных дуплексов проводят после отмывки биочипа, сравнивая интенсивности сигналов флуоресценции соответствующих ячеек биочипа. Отмывка может быть проведена в любом известном в данной области техники буфере с добавлением соли (SSC, SSPE и т.п.) или в деионизованной воде, но за более короткое время [Molecular cloning: a laboratory manual / J.F. Sambrook, D.W. Russell // Cold Spring Harbor Laboratory Press. - 2001].
После проведения гибридизации и отмывки биочипа проводится визуализация результатов гибридизации с помощью возбуждения флуоресценции прогибридизовавшегося меченого ПЦР-продукта второго раунда ПЦР, на основании которой делается вывод о генотипе в исследуемом образце. Регистрация картины гибридизации может быть произведена с помощью любой детектирующей системы, распознающей флуоресцентный сигнал (флуоресцентный микроскоп с ПЗС-камерой, лазерный сканер, портативный анализатор биочипов и т.п. коммерчески доступные флуоресцентные анализаторы, например портативный анализатор биочипов, снабженный ПЗС-камерой и специальным программным обеспечением, производства ООО «БИОЧИП-ИМБ» (Москва, Россия).
Биочипы могут быть изготовлены посредством последовательного нанесения на поверхность стеклянной подложки матрицы из ячеек акриламидного геля, активации ячеек и ковалентной иммобилизации в ячейках модифицированных олигонуклеотидов, несущих активные группы [Analysis of SNPs and other genomic variations using gel-based chips / A. Kolchinsky, A. Mirzabekov // Hum Mutat. - 2002. - Vol. 19. - P. 343-360. Review]. В качестве подложки помимо стекла может быть использован другой материал, в том числе металл, гибкие мембраны и пластик [Применение немодифицированных полимерных материалов для изготовления подложки биочипов, биочип на их основе и способ его изготовления, способ иммобилизации гидрогелей на немодифицированных полимерных материалах / С.В. Паньков, Э.Я. Крейндлин, О.Г. Сомова, и др. // Патент RU 2309959. - 2007]. Бочипы также могут быть изготовлены любыми другими известными специалисту в данной области способами [Arrays of immobilized oligonucleotides-contributions to nucleic acids technology / H. Seliger, M. Hinz, E. Happ // Curr Pharm Biotechnol. - 2003. - Vol. 4. - P. 379-395].
Для изготовления биочипа в настоящем изобретении используется набор олигонуклеотидов SEQ ID NO: 33-72, приведенных в Перечне последовательностей, а также в табл. 4. В качестве контроля прохождения гибридизации в настоящем изобретении используют образец контрольной ДНК. Расположение конкретных олигонуклеотидных зондов на биочипе может варьироваться и определяется только удобством интерпретации результатов гибридизации.
Далее приводятся примеры, которые показывают применение способа анализа соматических мутаций в гене EGFR, KRAS и BRAF, ассоциированных с чувствительностью рака легких к противоопухолевой таргетной терапии. Следует понимать, что приводимые примеры служат исключительно для иллюстрации и не предназначены для ограничения объема притязаний, выраженных в формуле изобретения. На основании настоящего описания специалист в данной области сможет легко предложить свои варианты и модификации осуществления изобретения, не отходя от общей концепции настоящего изобретения и без привлечения собственной изобретательской деятельности, так что должно быть понятно, что такие варианты и модификации также будут входить в объем притязаний настоящего изобретения.
Пример 1. Амплификация фрагментов генов EGFR, KRAS и BRAF методом «гнездовой» LNA-блокирующей ПЦР с целью получения флуоресцентно меченого ПЦР-продукта в необходимом количестве.
Из операционного материала, фиксированного в парафиновых блоках, образцы опухолевых тканей получали с помощью ручной микродиссекции под гистологическим контролем. Геномную ДНК выделяли с помощью набора QIAamp DNA FFPE Tissue Kit (Qiagen, Hilden, Германия).
Наработку локусов генов EGFR, KRAS и BRAF проводили в двух параллельных мультиплексных реакциях. Первая мультиплексная реакция содержала следующие праймеры и LNA-олигонуклеотиды - SEQ ID NO: 1, 2, 5, 6, 9, 10, 13, 14, 26-29; вторая мультиплексная реакция - SEQ ID NO: 17, 18, 20, 21, 23, 24, 30-32. ПЦР проводили на приборе Dyad («Bio-Rad», США). ПЦР-смесь первого этапа общим объемом 25 мкл включала в себя: 1 × ПЦР-буфер (67 мМ Трис-HCl, pH 8.6, 166 мМ (NH4)2SO4, 0,01% Тритон Х-100), 1.5 мМ MgCl2, 0.2 мМ каждого из дНТФ («Силекс», Россия), 2.5 U Taq-полимеразы («Силекс», Россия), по 0.2 мкМ праймеров, 0.05 мкМ LNA-олигонуклеотидов и 25 нг ДНК. Амплификацию проводили по следующей схеме: денатурация при 94°C (3 мин 30 с), далее 35 циклов: 94°C (30 с), 62°C (20 с), 72°C (10 с), затем элонгация при 72°C в течение 3 мин. Смесь второго этапа ПЦР отличалась составом и концентрацией праймеров. Она содержала по 0.2 мкМ прямых праймеров (первый мультиплекс: SEQ ID NO: 3, 7, 11, 12, 15; второй мультиплекс: SEQ ID NO: 17, 20, 23), и по 2 мкМ обратных праймеров (первый мультиплекс: SEQ ID NO: 4, 8, 12, 16; второй мультиплекс: SEQ ID NO: 19, 22, 25) и не содержала LNA-олигонуклеотида. Для флуоресцентного мечения ПЦР-продукта второго раунда смесь содержала 0.2 нМ флуоресцентно меченого дУТФ-Су5, который встраивался в цепь в процессе амплификации. В качестве матрицы в смесь добавляли 2 мкл продукта I этапа ПЦР и проводили амплификацию по следующей схеме: денатурация при 94°C (3 мин 30 с), далее 35 циклов: 94°C (30 с), 62°C (20 с), 72°C (10 с), затем элонгаци