Последовательность днк-аптамеров, связывающаяся с бактериями escherichia coli o157:h7
Иллюстрации
Показать всеИзобретение относится к биотехнологии, а именно к последовательности ДНК-аптамера. Указанный ДНК-аптамер связывается с бактериями Escherichia coli O157:Н7 и представлен одноцепочечной ДНК O157Flank длиной 81 нуклеотид и молекулярной массой 27 кДа. ДНК-аптамер обладает высокой специфичностью связывания в отношении бактерий Escherichia coli O157:Н7 и имеет первичную последовательность CATACGTTCGACTGCTACTCTGAGTCTGACCTTACAGTCAGTCAATCATGGGACAT ACGAAATGTGAGATGTACAGACTAG. Последовательность ДНК-аптамера отобрана в результате раундов позитивной селекции против бактерий Escherichia coli O157:Н7 и негативной селекции против бактерий других серогрупп Escherichia coli и близкородственных микроорганизмов с использованием тРНК в качестве молекулы-компетитора за связывание. ДНК-аптамер способен специфически детектировать бактерии Escherichia coli O157:H7 в реакции иммуноаптамерной ПЦР в концентрации до 103 микробных клеток на 1 мл раствора. Предложенное изобретение позволяет с высокой эффективностью детектировать бактерии Е. coli O157:Н7. 3 ил., 2 табл., 6 пр.
Реферат
Изобретение относится к области биоинженерии, биотехнологии, медицинской микробиологии, биохимии, диагностики и терапии особо опасных инфекций.
Основными областями применения ДНК-аптамеров к бактериям штамма Escherichia coli O157:H7 являются микробиологические и клинические исследования, медицинская диагностика кишечных инфекций, в том числе создание высокочувствительных и высокоэффективных систем для детекции данного особо опасного инфекционного возбудителя.
Е. coli O157:H7 является одним из наиболее опасных возбудителей острых кишечных инфекций и вызывает тяжелое острое кишечное заболевание, часто сопряженное с развитием угрожающего жизни гемолитико-уремического синдрома (ГУС). ГУС характеризуется острой почечной недостаточностью, тромбоцитопенией и микроангеопатической гемолитической анемией [Boyer О., Niaudet Р. // Int. J. Nephrol. -2011. - doi: 10.4061/2011/908407]. Вероятность развития ГУС составляет от 3-7% случаев при спорадических инфекциях, вызванных Е. coli O157:Н7, до 20% случаев при эпидемическом распространении заболевания [Mead P.S., Griffin P.M. // Lancet - 1998. - V. 352. - №9135. - P. 1207-1212]. При развитии ГУС острая почечная недостаточность наблюдается в 55-70% случаев, при этом смертность может составлять 3-7% [Parmar M.S. // Medscape and Medicine. http://emedicine.medscape.com/article/201181-overview#a0104]. У 30% переболевших могут развиться остаточные последствия в форме хронической почечной недостаточности, гипертонии и неврологических расстройств [Noris M., Caprioli J. с соавт.// Clin. J. Am. Soc. Nephrol. - 2010. - V.5. - №10. - P. 1844-1859]. Наиболее тяжело болезнь протекает у детей младше 5 лет и пожилых людей [Ruggenenti Ν., Remuzzi P.M. с соавт.// Kidney Int. - V.60. - №3. - P. 831-846]. Качество жизни пациентов после перенесенного заболевания существенно снижается, и в ряде случаев требуется многолетняя поддерживающая терапия для реабилитации функции почек.
В настоящее время острые кишечные инфекции, вызванные штаммами Е. coli O157:Н7, встречаются более чем в 30 странах мира, включая США, Канаду, Японию, страны Европы, страны Африки, Россию. Наиболее высокая частота ассоциированного с E. coli O157:H7 ГУС наблюдается в Аргентине [Rivero М.А., Passucci J.A. с соавт.// Foodborne pathogens and disease. - 2011. - V.8. - №8. - P.901-906]. Гемолитический уремический синдром (ГУС) более чем в 70% случаев является следствием инфекции, вызванной Е. coli серотипа O157:H7. Диагностика инфекции E coli O157:H7 на ранних этапах заболевания, а также своевременная идентификация бактерий в подозрительных продуктах питания и раннее применение адекватной терапии обеспечивают благоприятный прогноз исхода заболевания.
Основным фактором патогенности штаммов E. coli O157:H7 является продукция шига-подобных токсинов STX-1 и STX-2, которые, проникая в клетку гломерулярного эндотелия, связывают глоботриазилцерамид Gb3, что в силу различных, частично не до конца расшифрованных механизмов, приводит к инициации целого каскада событий сигнальной трансдукции, выражающихся в разнообразных физиологических последствиях - апоптозе, связывании лейкоцитов с клетками эндотелия, выбросе цитокинов, тромбообразовании, микроангиопатическом гемолизе [Psotka М.А., Obata F. с соавт.// Infect. Immun. - 2009. - V.77. - №3. - P.959-969]. Таким образом, шига-подобные токсины (а чаще, кодирующая их в составе бактерии ДНК) как правило, являются основной мишенью при разработке современных экспресс-методов диагностики патогенных штаммов E. coli в клинической практике (включая клиническую детекцию E. coli O157:H7), большинство из которых представляет собой ПЦР с последовательностей, кодирующих токсины [Bélanger S.D., Boissinot M. с соавт.// J. Clin. Microbiol. - 2002. - V.40. - №4. - P. 1436-1440]. Также шига-подобные токсины представляют собой основную мишень для разработки средств экстренной терапии заболевания [Tzipori S., Sheoran А. с соавт. // Clin. Microbiol. Rev. - 2004. -V.17. - №4. - Р.926-941]. Будучи в основном достаточно успешной, подобная стратегия часто ведет к признанию диагностической эквивалентности обнаружения шига-токсина как фактора вирулентности E. coli O157:H7 и наличия штамма-возбудителя заболевания, однако, она не проходила адекватной клинической валидации и в перспективе может оказаться терапевтически весьма рискованной, поскольку в настоящий момент широко известны случаи не только внутривидового (как в случае возникновения патогенного штамма E. coli О104:Н4 [Muniesa M., Hammerl J.А. с соавт. // Appl. Environ. Microbiol. - V.78. - №12. - Р.4065-4073], но и межвидового горизонтального переноса как генов, кодирующих шига-токсины, так и генов антибиотикоустойчивости [Me Manon M.A.S., Blair I.S. с соавт.// J. Appl. Microbiol. - 2007. - V.103. - №5. - P. 1883-1888, Diaz-Sánchez S., Sánchez S. с соавт.// Vet. Microbiol. - 2013. - V.163. - №3-4. - Р.:274-281]. Постоянное нарастание антибиотикоустойчивости патогенных штаммов может потребовать в дальнейшем разработки видо- и даже серогруппоспецифической противоинфекционной терапии. Однако, как ПЦР-анализ, так и рутинная иммунологическая идентификация шига-токсинов крайне затруднены в случае необходимости выявления сверхнизких количеств возбудителя в сложных комплексных образцах - продуктах питания, образцах окружающей среды и др. Таким образом, остро стоит вопрос о разработке высокочувствительных подходов к видо- и серогруппоспецифической идентификации E. coli O157:H7 и поиске молекулярных инструментов для осуществления данных процедур.
В мировой практике было неоднократно показано, что иммунологическая идентификация бактерий E. coli O157:H7 с помощью поли- и моноклональных антител осуществляется весьма успешно в силу существования у них уникальной для 0157 серогруппы структуры поверхностного О-антигена, состоящего из 40-80 повторяющихся субъединиц из 4 сахаров, включая N-ацетилпирозамин, L-фукозу, D-глюкозу и N-ацетил-D-галактозу [Wang L., Reeves P.R. // Infect. Immun. - 1998. - V.66. - №8. - P. 3545-3551], что облегчает получение высокоспецифических вариантов антител к данному патогену [Westerman R. В., Не Y. // J. Clin. Microbiol. - 1997. - V.35. - №3. -P.679-684]. На сегодняшний день диагностические тест-системы на основе ИФА активно используются для прямой идентификации E. coli O157:H7 наряду с ПЦР и стандартными микробиологическими методами [3M™ Tecra™ Е. coli O157 Visual Immunoassays (3M, США), Е. coli 0157 Microwell ELISA (SafePath, США) и др].
Однако антитела являются не единственным типом аффинных молекул, на основе которых возможна разработка средств эффективной детекции данного патогена. В качестве диагностических молекул также могут быть успешно использованы аптамеры - одноцепочечные олигонуклеотиды, которые благодаря образованию сложных трехмерных структур и пространственному расположению зарядов в молекуле имеют высокую аффинность в отношении заданных молекул. Направленный отбор аптамеров против бактериальных мишеней осуществляется по технологии SELEX ("Systematic Evolution of Ligands by Exponential Enrichment") [US Patent 5567588] с применением различных модификаций технологии, таких как cell-SELEX [Ohuchi S. // Biores. Open Acess. - 2012. - V.l. - №6. - P.265-272], проточная цитометрия [Raddatz M.S., Dolf А. с соавт. // Angew. Chem. Int. Ed. Engl. - 2008. - V.47. - P. 5190-5193], FRET [BrunoJG1, Carrillo MP. J Fluoresc. 2010 Nov; 20(6):1211-23.] и др.
Известны как РНК, так и ДНК аптамеры, полученные к различным белкам Е. coli - РНК-полимеразе [Kulbachinskiy Α., Feklistov А. с соавт.// Eur. J. Biochem. - 2004. -V.271. - №23-24. - P.4921-4931], фактору высвобождения I (RFI) Ogawa Α., Nishi Т. с соавт. // Nucleic Acids Symp Ser (Oxf).- 2005. - V.49. - P.269-270], внешним мембранным белкам (OMP) [Bruno J.G., Carrillo M.P., Phillips T. // J. Fluoresc. - 2010. - V.20. - №6. -P.1211-1223; Lee H.J., Kim B.C. // Biosens. Bioelectron. - 2009. - V.24. - №12. - P.3550-3555.].
Известен РНК-аптамер, специфичный к бактериям Е. coli 0157 серогруппы, полученный корейскими исследователями [Патент WO 2012081907 A3; Lee Y.J., Han S.R. с соавт. // Biochem Biophvs Res Commun. 2012 Jan 6;417(1):414-20]. Данный аптамер 1-11 был идентифицирован в виде последовательности, содержащей 99 нуклеотидов из которой позже была выделена функциональная часть длиной 64 нуклеотида, способная связываться с бактериальными клетками (KD=110 нМ). Однако для данного аптамера не приведены результаты исследования специфичности его взаимодействия с клетками E. coli O157:H7, а эффективность связывания с клетками невысока. Кроме того, РНК малоустойчива во внешней среде и быстро деградирует, таким образом, без оптимизации и стабилизации данного аптамера он не может быть использован для целей диагностики и терапии.
Известен наиболее близкий аналог данного изобретения, ДНК-аптамер, описанный китайскими исследователями и примененный для детекции бактерий Е. coli O157:H7 в разработанных аптасенсорных системах с использованием колориметрической детекции [Wu W., Zhang J. с соавт. // PLOS One. - 2012. - V.7. - №11. - P. 1-9] и детекции на основе золотых наночастиц [Wu W., Li M. с соавт. // Nanoscale Research Letters. - 2012. - V.7. - №:658. - P.1-7]. В работах приводится первичная последовательность использованного аптамера, информация о специфичности детекции бактерий, проанализированная на панели близкородственных микроорганизмов, но не дается информации о способе его получения и параметрах связывания с бактерией. Тем не менее, можно на основе опубликованных данных можно сделать вывод, что чувствительность детекции с помощью данного аптамера составляет до 104 бактериальных клеток Е. coli O157:H7 в зависимости от примененного метода, что значительно ниже чувствительности тест-систем для детекции данных бактерий, основанных, в частности, на ПЦР-детекции, сопряженной с применением высокоспецифических моноклональных антител [Патент WO 1998020148 А1].
Задачей настоящего изобретения является получение ДНК-аптамеров к бактериям Е. coli O157:H7, обладающих аффинностью и специфичностью в отношении данных бактерий, стабильностью при длительном хранении и пригодных для разработки диагностических тест-систем.
Поставленная задача решается с использованием разработанной схемы селекции аптамеров к бактериальным клеткам, основанной на применении тРНК в качестве конкурента за связывание, что позволяет отобрать аптамеры с высокой специфичностью к мишени в результате малого числа раундов селекции.
Также поставленная задача решается применением раундов негативной и позитивной селекции библиотеки ДНК-олигонуклеотидов против бактерий Е. coli O157:H7, непатогенного штамма Е. coli DH12S и близкородственных микроорганизмов, что позволяет удалить из селектируемого пула последовательности ДНК, неспецифичные в отношении бактерий Е. coli серогруппы О157.
Также поставленная задача решается путем клонирования селектированных последовательностей ДНК в плазмидный вектор, определения их первичной структуры и проверки индивидуальных последовательностей ДНК на связывание с бактериями Е. coli O157:H7 при помощи колориметрической детекции в твердофазном анализе и иммуноаптамерной ПЦР.
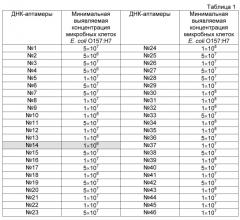
Техническим результатом изобретения является одноцепочечный ДНК-аптамер O157Flank длиной 81 нуклеотид с молекулярной массой 27кДа и первичной последовательностью
CATACGTTCGACTGCTACTCTGAGTCTGACCTTACAGTCAGTCAATCATGGGACAT ACGAAATGTGAGATGTACAGACTAG,
специфически связывающийся с бактериями Е. coli O157:H7 и способный детектировать их в реакции иммуноаптамерной ПЦР в концентрации до 103 микробных клеток на 1 мл раствора.
Предлагаемое техническое решение предусматривает сочетание раундов негативной и позитивной селекции, что существенно повышает специфичность отобранных последовательностей.
Также техническое решение предполагает применение тРНК в качестве конкурента за связывание бактериальной клетки, что позволяет выделить при отборе исключительно те ДНК-аптамеры, которые специфически связываются с поверхностью бактерий.
Также техническое решение предполагает тестирование отобранных аптамеров против панели близкородственных к Е. coli O157:H7 микроорганизмов, что позволяет отобрать последовательности ДНК, специфичные исключительно к данным бактериям.
Важным преимуществом предлагаемой системы получения ДНК-аптамеров, специфичных к Е. coli O157:H7, является возможность отбора пула высокоаффинных ДНК-аптамеров с минимальным содержанием низкоаффинных молекул при применении малого количества раундов селекции.
Изобретение осуществляют следующим образом.
Синтезируют комбинаторную библиотеку ДНК-олигонуклеотидов вида 5′-constl-N45-const2, где N45 - вариабельная область, состоящая из 45 нуклеотидов, а const1 и const2 - константные фланкирующие области длиной по 18 нуклеотидов, которые служат для связывания со специфическими праймерами, позволяющими амплифицировать пул олигонуклеотидов после этапов селекции (Фиг. 1А). Негативную селекцию из комбинаторной библиотеки осуществляют в отношении фиксированных 1% формалина бактерий непатогенного штамма Е. coli DH12S и инактивированных культур штаммов панели близкородственных бактерий. Разделение бактерий со связанными аптамерами от несвязавшихся последовательностей ДНК осуществляют центрифугированием бактериальной суспензии при 6000 g в течение 10 минут. На каждом раунде селекции пул несвязавшихся олигонуклеотидов амплифицируют в ПЦР с применением праймеров: прямого ForFAM, меченого F AM по 5′-концу цепи, и обратного RevP, несущего на 5′-конце остаток фосфорной кислоты (Фиг. 1Б). Двуцепочечный ПЦР-продукт обрабатывают 5′-экзонуклеазой фага лямбда для получения одноцепочечных ДНК-олигонуклеотидов. Деградацию цепи, фосфорилированной по 5′-концу, визуально контролируют электрофорезом в ПААГ по наличию в электрофореграмме единичной меченой FAM полосы одноцепочечной ДНК. Таким образом проводят три раунда негативной селекции.
Проводят пять последовательных раундов позитивной селекции с использованием бактерий штамма Е. coli O157:H7, инактивированных 1% формалина, как описано выше для процесса негативной селекции.
Из отобранного пула получают индивидуальные последовательности ДНК-аптамеров. Для этого пул олигонуклеотидов, прошедший все раунды селекции, амплифицируют с применением прямого ForP и обратного RevP праймеров (Фиг. 1Б), фосфорилированных по 5′-концу. Двуцепочечный ПЦР-продукт лигируют в вектор pBluescriptll SK(-) (Agilent Technologies, США), расщепленный эндонуклеазой рестрикции Smal (Thermo, США) и обработанный щелочной фосфатазой (Invitrogen, США). Полученной плазмидой трансформируют электрокомпетентные клетки Е. coli DH12S. Клетки высевают на агаризованную питательную среду, содержащую ампициллин, X-Gal и IPTG. По принципу бело-голубой селекции выбирают и отсевают колонии, содержащие вставку в составе плазмиды. Проверяют наличие искомой вставки в ПЦР со стандартными праймерами M13/pUC. Колонии, содержащие вставку, соответствующую по размеру единичному аптамеру, подвергают амплификации с собственных праймеров ForFAM и RevP и получают одноцепочечные фрагменты методом, описанным выше.
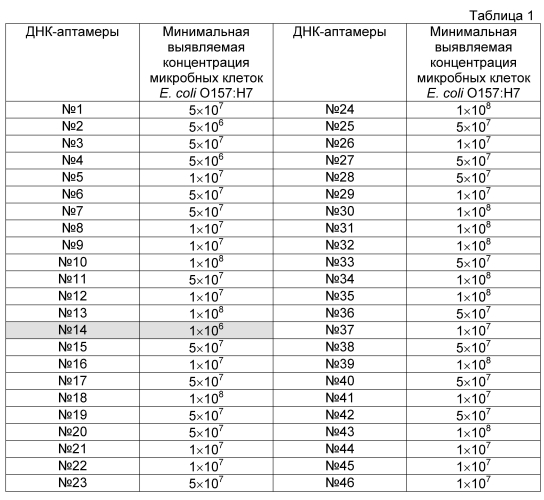
Проводят скрининг полученных индивидуальных одноцепочечных последовательностей ДНК в отношении чувствительности и специфичности связывания с инактивированными бактериями Е. coli O157:H7 и инактивированными бактериями близкородственных микроорганизмов. Для этого одноцепочечную ДНК индивидуальных аптамеров амплифицируют в ПЦР с использованием праймера, содержащего на 5′-конце биотин (ForBio, Фиг. 1В), получают одноцепочечную биотинилированную ДНК обработкой 5′-экзонуклеазой фага лямбда и очищают одноцепочечный продукт реакции с помощью набора QIAquick PCR Purification kit (Qiagen, США). Полученную ДНК инкубируют в лунках ПЦР-планшета, на поверхности которых иммобилизованы инактивированные бактерии Е. coli O157:H7 либо бактерии близкородственных микроорганизмов. Лунки планшета промывают от свободных молекул ДНК, а затем инкубируют с конъюгатом нейтравидин-пероксидаза (Pierce, США) в течение часа. По окончании инкубации промывают лунки от несвязавшегося конъюгата и проявляют добавлением раствора ТМВ Substrate Kit (Pierce, США). Отбирают ДНК-аптамеры, обнаружившие высокий уровень связывания с бактериями Е. coli O157:H7, но при этом не взаимодействующие с близкородственными бактериями (Таблица 1). Дополнительные проверки отобранных клонов проводят в реакции иммуноптамерной ПЦР.
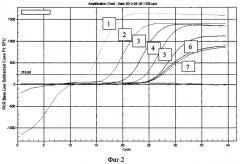
Для постановки иммуноаптамерной ПЦР парамагнитные частицы, несущие антитела к Е. coli O157:H7 (Dynabeads® anti-is. coli O157, Applied Biosystems, США), разносят в лунки планшета, добавляют к частицам различные концентрации инактивированных бактерий Е. coli O157:H7 (от 106 до 101 м.к. на 1 мл) и инкубируют в течение 40 минут при комнатной температуре. По окончании инкубации планшет промывают с использованием магнитной платы на вошере Bio-Plex Pro (Bio-Rad, США), блокируют свободные валентности связывания раствором бычьего сывороточного альбумина, добавляют одноцепочечную ДНК индивидуальных аптамеров (0,1 мг/мл) и инкубируют в течение 30 минут со встряхиванием. После завершения инкубации парамагнитные частицы в лунках планшета шестикратно отмывают от несвязавшейся ДНК трис-солевым буфером с использованием магнитной платы вошера Bio-Plex Pro и обрабатывают частицы 10 мМ NaOH в течение 5 минут. Раствор щелочи, содержащий денатурированную ДНК, переносят в лунки ПЦР-планшета для постановки реакции в режиме реального времени, нейтрализуют, добавляют смесь для ПЦР-амплификации с красителем SYBR Green 1 и проводят ПЦР в режиме реального времени с праймеров, соответствующих фланкирующим константным зонам ДНК-аптамеров. На основании результатов ПЦР-амплификации отбирают ДНК-аптамеры, эффективно взаимодействующие с бактериями Ε coli O157:H7, но не взаимодействующие с близкородственными микроорганизмами, в частности, аптамер O157Flank (Фиг. 2, Таблица 2), детектирующий бактерии Е. coli O157:H7 в концентрации 103 м.к./мл раствора и выше.
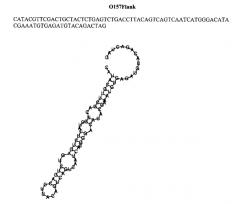
Для ДНК-аптамеров, обнаруживших наилучшие характеристики чувствительности и специфичности связывания с бактериями, в частности, аптамера 0157Flank, определяют первичную структуру методом капиллярного секвенирования и проводят моделирование вторичной структуры с использованием программы RNAFold (http://rna.tbi.univie.ac.at/cgi-bin/RNAfold.cgi) (Фиг. 3).
Изобретение иллюстрируют следующие графические материалы.
Фиг. 1. Последовательности ДНК, используемые для отбора молекул ДНК, для обогащения селектированного пула, проведения амплификации и анализа полученных аптамерных последовательностей.
A. Последовательность олигонуклеотидной библиотеки, используемой в процессе селекции, где N45 - вариабельная область, const1 и const2 - константные области для отжига специфических праймеров;
Б. Последовательности праймеров, используемых в реакциях обогащения библиотеки, для клонирования и наращивания количества последовательностей ДНК-аптамеров;
B. Последовательность биотинилированного праймера, используемого в реакции амплификации для получения биотинилированных вариантов олигонуклеотидных последовательностей аптамеров.
Фиг. 2. Детекция бактерий Е. coli O157:H7 аптамером O157Flank по методу иммуноаптамерной ПЦР.
1-6 - результаты детекции микробных клеток Е. coli O157:H7 в концентрациях 1×106 (1), 1×105 (2), 1×104 (3), 1×103 (4), 1×102 (5), 1×101 (6) м.к./мл, 7 - отрицательный контроль (реакция РВ-ПЦР без добавления аптамера)
Фиг. 3. Первичная последовательность и результат моделирования вторичной структуры аптамера O157Flank, специфичного к бактериям Е. coli O157:H7.
Таблица 1. Результаты анализа по определению эффективности связывания аптамеров с микробными клетками Е. coli O157:H7.
Таблица 2. Результаты (номера циклов) ПЦР-амплификации при анализе специфичности взаимодействия ДНК-аптамера O157Flank с бактериями O157:H7 и близкородственными бактериями по методу иммуноаптамерной ПЦР
Для лучшего понимания сущности изобретения ниже следуют примеры его конкретного выполнения.
Пример 1. Проведение раунда негативной селекции последовательностей ДНК-олигонуклеотидов из пула комбинаторной библиотеки олигонуклеотидов против бактерий Е. coli DH12S и микроорганизмов, близкородственных Е. coli O157:H7.
Для проведения раундов негативного отбора используют инактивированные 1% формалина культуры генно-инженерного штаммов Е. coli DH12S и штаммов близкородственных Е. coli O157:H7 микроорганизмов (Е. coli О6 25922 АТСС, Е. coli O55 3912/41, Shigella dysenteriae 1362, Salmonella typhi 13311 АТСС, Yersinia enterocoliticavar. 1 Красноярск-6, Yersinia pseudotuberculosis III 1) в концентрации 1×108 м.к./мл.
Инактивированные 1% формалина культуры E. coli DH12S и микроорганизмов, близкородственных E. coli O157:H7, в концентрации 1×108 м.к./мл отмывают 5 раз путем центрифугирования (12000×g 3 мин) трисовым буфером (ТБ), содержащим 20 мМ трис-HCl, 50 мМ NaCl, pH 7,0. Осадок бактерий ресуспендируют в 100 мкл того же буфера, содержащего 50 нМ тРНК и 0,05% БСА, и добавляют библиотеку олигонуклеотидов в соотношении 1×106 молекул ДНК на 1 микробную клетку. После инкубации в течение 30 минут при температуре 37°C удаляют супернатант с не связавшимися молекулами ДНК путем центрифугирования (12000 × g 3 мин). Несвязавшиеся с микробными клетками последовательности ДНК, содержащиеся в супернатанте, амплифицируют с использованием прямого - For FAM и обратного - RevP праймеров при следующих условиях: предварительная денатурация ДНК - 3 минуты при температуре 95°С; 25 циклов, состоящих из денатурации ДНК в течение 30 с при температуре 95°С, отжига праймеров в течение 20 с при температуре 54°С, элонгации комплементарной цепи ДНК в течение 10 с при температуре 72°C с использованием Taq полимеразы. Фосфорилированную цепь ДНК расщепляют 5′-экзонуклеазой фага лямбда в течение 1 часа при 37°С. Полученный пул одноцепочечных последовательностей экстрагируют смесью фенол-хлороформ (1:1), осаждают этанолом, растворяют в 100 мкл деионизованной автоклавированной воды.
Получение одноцепочечных молекул ДНК контролируют в 10% ПААГ электрофорезе с мочевиной. В качестве электродного буфера используют буфер ТБЕ (трис - 108 г/л, борная кислота - 55 г/л, ЭДТА - 7,4 г/л).
Пример 2. Проведение раунда позитивной селекции последовательностей ДНК-олигонуклеотидов против бактерий Е. coli O157:H7.
Инактивированную 1% формалина культуру Е. coli O157:H7 в концентрации 1×108 м.к./мл отмывают 5 раз путем центрифугирования (12000×g 3 мин) трисовым буфером (ТБ), имеющим следующий состав: 20 мМ трис - HCl, 50 мМ NaCl, pH 7,0. Осадок ресуспендируют в 100 мкл того же буфера, содержащего 50 нМ тРНК и 0,05% БСА, и добавляют пул олигонуклеотидов, отобранных в результате трех раундов негативной селекции против бактерий Е. coli DH12S и микроорганизмов, близкородственных Е. coli O157:H7, в соотношении 1×106 молекул ДНК на 1 микробную клетку. После инкубации в течение 30 минут при температуре 37°С удаляют не связавшиеся молекулы ДНК путем центрифугирования (12000×g 3 мин), многократно отмывая осадок клеток буфером ТБ с 0,05% БСА. Связавшиеся с микробными клетками последовательности ДНК амплифицируют с использованием прямого - ForFAM и обратного - RevP праймеров при следующих условиях: предварительная денатурация ДНК - 3 минуты при температуре 95°С; 25 циклов, состоящих из денатурации ДНК в течение 30 с при температуре 95°С, отжига праймеров в течение 20 с при температуре 54°С, элонгации комплементарной цепи ДНК в течение 10 с при температуре 72°C с использованием Taq полимеразы. Фосфорилированную цепь ДНК расщепляют 5′-экзонуклеазой фага лямбда в течение 1 часа при 37°С. Полученный пул одноцепочечных последовательностей экстрагируют смесью фенол-хлороформ (1:1), осаждают этанолом, растворяют в 100 мкл деионизованной автоклавированной воды.
Получение одноцепочечных молекул ДНК контролируют в 10% ПААГ электрофорезе с мочевиной. В качестве электродного буфера используют буфер ТБЕ (трис - 108 г/л, борная кислота - 55 г/л, ЭДТА - 7,4 г/л).
Пример 3. Получение индивидуальных аптамерных кандидатов.
Фрагменты для клонирования в коммерческий вектор pBluescriptll SK(-) (Agilent Technologies, США) нарабатывают на основе пула селектированных олигонуклеотидов при помощи ПЦР с использованием прямого и обратного фосфорилированных праймеров ForP и RevP (отжиг праймеров при 54°С, элонгация 15 секунд) (Фиг. 1). Плазмиду pBluescriptll SK(-) обрабатывают эндонуклеазой рестрикции Smal в буфере Tango (Thermo, США), после чего обрабатывают щелочной фосфатазой (Invitrogen, США). Фосфорилированные двуцепочечные фрагменты ДНК лигируют в подготовленную плазмиду.
Электрокомпетентные клетки Е. coli DH12S трансформируют подготовленной ранее плазмидой pBluescriptll SK(-), содержащей вставки селектированных последовательностей, применив электротрансформацию (прибор Eppendorf Electroporator 2510 (Eppendorf, Германия), режим 1,7 KB). Трансформированные клетки высевают на плотную среду с 2хУТ-агаром, содержащим 50 мкг/мл ампициллина, 80 мкг/мл X-Gal и 20 мМ IPTG, и выращивают в течение ночи при 37°С.
Единичные колонии, потенциально содержащие вектор со вставкой (по принципу бело-голубой селекции [Ullmann A, Jacob F, Monod J. // J Mol Biol. - 1967. -V. 24. - N. 2. - P. 339-343.]) верифицируют на наличие единичного встроенного фрагмента ДНК искомой длины в плазмиде с помощью ПЦР со стандартными прямым и обратным праймерами M13/pUC (Fermentas, США). Наращивают количество копий индивидуальных последовательностей клонированных фрагментов ДНК амплификацией с использованием праймеров ForFAM и RevP (Фиг. 1). Получают одноцепочечный продукт для каждого из клонов обработкой 5′-экзонуклеазой фага лямбда с визуальным контролем в электрофорезе. Таким образом, нарабатывают клонированные одноцепочечные индивидуальные последовательности из 500 бактериальных клонов.
Пример 4. Скрининг индивидуальных олигонуклеотидных последовательностей на связывание с бактериями Е. coli O157:H7 в твердофазном анализе с использованием нейтравидин-пероксидазы.
Для проведения скрининга 96-луночные планшеты сенсибилизируют инактивированными микробными клетками штаммов Е. coli O157:H7, штаммов Е. coli других серогрупп и близкородственных микроорганизмов, использованных для негативного отбора, в концентрации 1×106 м.к./лунку. В лунки, служащие отрицательным контролем, вносят буфер ТБ, не содержащий микробной суспензии. Аптамерные последовательности амплифицируют с использованием прямого биотинилированного праймера - ForBio (Фиг. 1) и обрабатывают 5′-экзонуклеазой фага лямбда, получая биотинилированные одноцепочечные молекулы ДНК. Аптамеры вносят в лунки планшетов в 50 мкл буфера ТБ в концентрации 10 нМ/лунку. После инкубации в течение 1 часа при 37°С планшеты 10 раз отмывают тем же буфером.
По окончании инкубации в лунки планшетов вносят по 100 мкл буфера ТБ, содержащего нейтравидин, конъюгированный с пероксидазой хрена (Pierce, США) в рабочем разведении 1:1000. После 30-ти минутной инкубации шесть раз отмывают планшеты от несвязавшегося конъюгата и добавляют в лунки планшетов по 100 мкл субстрата ТМВ Substrate Kit (Pierce, США). Реакцию останавливают 2 M серной кислотой (50 мкл/лунку) после развития окраски. Результаты считают положительными, если значения оптической плотности в лунках с образцами не менее чем в два раза превышают значение оптической плотности в лунке с отрицательным контролем (K-). Учет результатов производят спектрофотометрически при длине волны 450 нм. Результаты оформляют графически в виде таблицы (Таблица 1), на основе которой отбирают варианты аптамеров, наиболее эффективно взаимодействующие с клетками Е. coli O157:H7, в частности ДНК-аптамер O157:Flank (№14).
Пример 5. Анализ взаимодействия аптамера O157Flank с бактериями Е. coli O157:H7 с использованием иммуноаптамерной ПЦР.
Для проведения анализа в микропробирки типа «эппендорф» вносят по 50 мкл суспензии парамагнитных частиц с иммобилизованными моноклональными антителами к Е. coli O157:H7 (Dynabeads® anti-E. coli O157, Applied Biosystems, США) и добавляют по 200 мкл инактивированной культуры штамма Е. coli O157:H7 1282ГИСК в концентрациях от 1×106 м.к./мл до 1×101 м.к./мл с десятикратным шагом. В качестве отрицательного контроля используют пробирку с буфером, не содержащим микробных клеток. После 40 минут инкубации удаляют несвязавшиеся бактерии троекратной промывкой буфером ТБ с использованием магнитной платы на вошере Bio-Plex Pro (Bio-Rad, США). Блокируют свободные сайты связывания 3% раствором бычьего сывороточного альбумина в буфере ТБ в течение 60 минут при температуре 37°С при встряхивании, снова троекратно промывают частицы буфером ТБ. Добавляют 200 мкл раствора анализируемого ДНК-аптамера (0157Flank) в концентрации 0,1 мг/мл и инкубируют планшет в течение 30 минут на шейкере, после чего шестикратно отмывают частицы от несвязавшейся ДНК с использованием магнитной платы на вошере Bio-Plex Pro (Bio-Rad, США). Частицы обрабатывают 10 мМ NaOH в течение 5 минут. Раствор щелочи, содержащий денатурированную ДНК, переносят в лунки ПЦР-планшета для постановки реакции в режиме реального времени, нейтрализуют добавлением 100 мМ соляной кислоты, добавляют смесь для ПЦР-амплификации с красителем SYBR Green 1 (Синтол, Россия) и проводят ПЦР в режиме реального времени с праймеров, соответствующих фланкирующим константным зонам ДНК-аптамеров (ForP, RevP, Фиг. 1) по протоколу, рекомендованному производителем прибора для ПЦР в режиме реального времени (iCycler IQ PCR Detection System, Bio-Rad), а именно: предварительная денатурация ДНК в течение 3 минут при температуре 95°С; 45 циклов, состоящих из денатурации ДНК в течение 30 с при температуре 95°С, отжиг праймеров в течение 20 с при температуре 54°С, элонгация комплементарной цепи ДНК в течение 10 с при температуре 72°C с использованием Taq полимеразы.
Аналогичным образом проводят тестирование взаимодействия аптамера и инактивированных культур бактерий, близкородственных Е. coli O157:H7 (Таблица 2).
Результаты проведенного анализа демонстрируют, что ДНК-аптамер O157Flank специфически выявляет микробные клетки Е. coli O157:H7 в тест-системе на основе иммуноаптамерной ПЦР в концентрациях 1×103 м.к./мл и выше и не взаимодействует с близкородственными бактериями (Фиг. 2, Таблица 2).
Пример 6. Определение первичной и моделирование вторичной структуры полученных ДНК-аптамеров к бактериям Е. coli O157:H7.
Определение первичной структуры аптамеров проводят методом капиллярного секвенирования. Моделирование вторичной структуры осуществляют с использованием программы RNAFold (http://rna.tbi.univie.ac.at/cgi-bin/RNAfold.cgi). Аптамер О157Flank имеет нуклеотидную последовательность и предположительную первичную структуру, представленные на рисунке (Фиг. 3).
| Таблица 2 | ||||||
| Штаммы микроорганизмов | Концентрация исследуемых микроорганизмов, м.к./мл | |||||
| 1×106 | 1×105 | 1×104 | 1×103 | 1×102 | 1×101 | |
| Циклы ПЦР | ||||||
| E. coli O157:H7 1282 ГИСК (положительный контроль) | 13 | 17 | 22 | 24 | 27 | 27 |
| E. coli O6 25922 АТСС | 27 | 27 | 27,9 | 28,1 | 28,8 | 28,9 |
| E. coli O55 3912/41 | 27,2 | 27,7 | 28,2 | 28,1 | 28,2 | 28,3 |
| Salmonella enteritidis var. Issat 2 | 27,2 | 27,9 | 28,6 | 28,4 | 28,0 | 28,1 |
| Shigella dysenteriae 1362 | 27,7 | 27,5 | 28,3 | 28,3 | 28,5 | 28,7 |
| Shigella flexneri 4888 | 28,2 | 28,1 | 28,0 | 28,3 | 28,6 | 29,1 |
| Shigella sonnei 4385 | 27,3 | 28,0 | 27,3 | 27,5 | 28,2 | 28,3 |
| Vibrio cholerae non O1 9741-P | 28,2 | 28,2 | 28,1 | 28,2 | 28,1 | 29,3 |
Перечень последовательностей к заявке на получение патента «Последовательность ДНК-аптамеров, связывающаяся с бактериями Escherichia coli O157:H7»
Последовательность ДНК-аптамеров, связывающаяся с бактериями Escherichia coli O157:Н7 представлена одноцепочечной ДНК O157Flank длиной 81 нуклеотид и молекулярной массой 27 кДа, обладает высокой специфичностью связывания в отношении бактерий Escherichia coli O157:Н7 и имеет первичную последовательность CATACGTTCGACTGCTACTCTGAGTCTGACCTTACAGTCAGTCAATCATGGGACAT ACGAAATGTGAGATGTACAGACTAG, отобрана в результате раундов позитивной селекции против бактерий Escherichia coli O157:Н7 и негативной селекции против бактерий других серогрупп Escherichia coli и близкородственных микроорганизмов с использованием тРНК в качестве молекулы-компетитора за связывание, способна специфически детектировать бактерии Escherichia coli O157:H7 в реакции иммуноаптамерной ПЦР в концентрации до 103 микробных клеток на 1 мл раствора.