Способ мультиплексной пцр-детекции atopobium vaginae, leptotrichia amnionii, sneathia sanguinegens и eggerthella spp. в клиническом материале
Иллюстрации
Показать всеИзобретение относится к области медицины и предназначено для ПЦР-детекции Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. в клиническом материале. Осуществляют выделение ДНК, амплификацию и электрофорез. Проводят мультиплексную ПЦР с использованием специфичных праймеров в циклическом режиме. При электрофоретическом обнаружении ампликонов размерами 286 п.н., и/или 768 п.н., и/или 135 п.н., и/или 428 п.н. определяют наличие Atopobium vaginae, и/или Leptotrichia amnionii, и/или Sneathia sanguinegens, и/или Eggerthella spp. соответственно. Изобретение обеспечивает одновременное выявление в клиническом материале до 4 видов ассоциируемых с бактериальным вагинозом бактерий. 1 ил., 2 табл., 10 пр.
Реферат
Предлагаемое изобретение относится к медицине, а именно к клинической лабораторной диагностике и медицинской микробиологии, и может быть использовано в медицинской практике для ускоренного обнаружения и идентификации Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. в клиническом материале для последующей диагностики бактериального вагиноза.
Установлено, что при бактериальном вагинозе (нарушении состояния микрофлоры влагалища) часто обнаруживаются микроорганизмы Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. Показано, что наличие данных микроорганизмов в клиническом материале коррелирует со степенью тяжести бактериального вагиноза. Соответственно, их раннее обнаружение имеет диагностическое значение [Fredricks D.N., Fiedler T.L., Marrazzo J.M. Molecular identification of bacteria associated with bacterial vaginosis. N. Engl. J. Med. 2005. 353:1899-1911].
В микробиологической практике для детекции и идентификации микроорганизмов в клиническом материале используют главным образом методы микроскопического исследования микропрепаратов (окраска по Граму, метиленовым синим и др.) и культурный метод (выделение чистых культур микроорганизмов) [Методики клинических лабораторных исследований: Справочное пособие. Том 3. / Под ред. В.В. Меньшикова. - М.: Лабора, 2009. - 880 с]. Указанные методы имеют ряд существенных недостатков: микроскопическое исследование с окраской микропрепарата по методу Грама позволяет дифференцировать по тинкториальным признакам лишь чистые культуры бактерий, выделенные преимущественно в виде колонии с поверхности плотных агаризованных сред. При окраске клинического материала (соскобы, отделяемое слизистых и др.) стандартизация условий жизнедеятельности бактерий и, соответственно, поддержание постоянства их тинкториальных признаков невозможно, поэтому в подавляющем большинстве случаев однозначная дифференцировка грамположительных и грамотрицательных бактерий по цвету проблематична. Микроскопия как метод в принципе не позволяет устанавливать видовую принадлежность микроорганизмов. Кроме того, при микроскопии микропрепаратов можно выявить микроорганизмы, присутствующие в биоматериале в количестве, обычно превышающем 105 КОЕ/мл, тогда как многие факультативно-анаэробные и аэробные бактерии могут проявлять патогенный эффект при сравнительно небольшом их количестве (до 104 КОЕ/мл), которое не выявляется при микроскопии. Недостатками световой микроскопии является то, что выявляют не более 10 морфотипов, при этом многие виды и роды бактерий, обнаруживаемые в микропрепаратах, морфологически однотипны. Например, Atopobium vaginae не имеет специфических микроскопических признаков и выглядит под микроскопом как коринебактерии, довольно часто встречающиеся у здоровых женщин. Световая микроскопия не позволяет дифференцировать ультраструктурные особенности исследуемых бактерий, в связи с этим идентификация бактерий до вида, опираясь при этом лишь на данные о форме, размерах и взаимном расположении, невозможна. Наконец, к недостаткам метода можно отнести субъективизм и зависимость результата исследования от квалификации врача-лаборанта. Поэтому в целом диагностическая ценность микроскопического исследования клинического материала относительно невысока, и микроскопия как метод применяется преимущественно для скрининга.
Более специфичным и чувствительным является культуральный метод. Однако многие представители микробиоты и бактерии, с которыми связывают дисбиотические состояния, включая Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp., являются облигатными анаэробами и не культивируются в обычных условиях. Более того, культуральное исследование отличает продолжительность (до 7 дней) и высокая себестоимость (специальные питательные среды и др.), а также целый ряд серьезных требований к преаналитическому этапу (сроки, транспортные среды и др.). Себестоимость культурального исследования возрастает на порядки в ходе биохимической идентификации выделенных культур, основанной на дополнительной характеристике колоний по более чем 50-70 биохимическим признакам, при этом не исключаются ошибки, обусловленные фенотипической изменчивостью бактерий.
В связи с этим для достоверного обнаружения Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. перспективными представляются молекулярно-биологические методы исследования, в частности полимеразная цепная реакция (ПЦР) с детекцией продуктов амплификации в агарозном геле.
Полимеразная цепная реакция (ПЦР) - метод амплификации ДНК in vitro, с помощью которого в течение нескольких часов можно выявить специфическую последовательность ДНК [Mullis К.В. The unusual origin of the polymerase chain reaction. Sci. Am., 1990, 262, 56]. Суть метода ПЦР состоит в том, что из исследуемого образца предварительно выделяется тотальная ДНК, далее вносятся олигонуклеотидные затравки (праймеры), ограничивающие специфические фрагменты искомой ДНК. В процессе амплификации происходит нарастание концентрации ДНК в геометрической прогрессии и поэтому, даже если в исходном растворе первоначально находилась только одна двуцепочечная молекула ДНК, то за 30-40 циклов в растворе накапливается около 108 молекул ампликона. Этого количества достаточно для достоверной визуальной детекции этого фрагмента методом электрофореза в агарозном геле. Метод позволяет в короткие сроки (в течение нескольких часов) выявить и идентифицировать любые микроорганизмы в любом биоматериале, полученном из области патологического процесса, без выделения чистой культуры при использовании специально подобранных специфичных праймеров. Достоинствами метода ПЦР являются высокая специфичность и чувствительность, универсальность процедуры, простота и удобство проведения анализа.
Перспективны возможности ПЦР реакции в контексте возможности одновременной детекции и идентификации в одной пробирке геномной ДНК микроорганизмов нескольких разных видов, так называемая мультиплексная ПЦР (multiplex PCR). Это обеспечивается одновременным внесением в реакционную смесь нескольких пар видоспецифичных праймеров, амплификацией и обнаружением видоспецифичных ампликонов, различающихся между собой размерами.
Наиболее близким аналогом (прототипом) является способ идентификации микроорганизмов микрофлоры влагалища, в том числе Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp., включающий ПЦР-амплификацию 16S pPHK с детекцией результатов в 2-3% агарозном геле и проведение сравнительного анализа конкретных представителей нормо- и условно-патогенной биоты у женщин с бактериальным вагинозом и без него [Fredricks D.N., Fiedler T.L., Marrazzo J.M. Molecular identification of bacteria associated with bacterial vaginosis. N. Engl. J. Med. 2005. 353:1899-1911]. Однако данный способ не позволяет проводить ПЦР детекцию в режиме мутиплексной реакции.
Задачей изобретения является разработка способа мультиплексной идентификации Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. в клиническом материале.
Технический результат, достигаемый при использовании изобретения, - сокращение продолжительности исследования, существенное увеличение чувствительности и специфичности одновременной детекции в клиническом материале бактерий четырех видов: Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp.
Указанный технический результат достигается тем, что в способе ПЦР-детекции Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. в клиническом материале, включающем выделение ДНК из исследуемого материала, амплификацию и электрофорез, согласно изобретению проводят мультиплексную ПЦР с использованием специфичных праймеров к Atopobium vaginae: 5′ gcgtaggcggtctgttaggtca 3′ и 5′ gttagctgcggcacggaaagtat 3′, к Leptotrichia amnionii: 5′ ctacggcactaaaggagag 3′ и 5′ cccgaggatgtcaagat 3′, к Sneathia sanguinegens: 5′ gcgatacatggcaaaactaagaaa 3′ и 5′ tgtgtacaagaccccgagaac 3′ и к Eggerthella spp.: 5′ ttgctcaagcggaacctctaatct 3′ и 5′ acgtttccgccgcttcacctaca 3′ в циклическом режиме, предусматривающем начальный цикл в течение 1 мин при температуре 94°C; затем 30 циклов, каждый из которых включает стадию денатурации в течение 30 сек при 94°C, стадию отжига праймеров в течение 30 сек при температуре 59°C и стадию элонгации в течение 30 сек при температуре 72°C; заключительный цикл в течение 2 мин при температуре 72°C, при электрофоретическом обнаружении ампликонов размерами 286 п.н. и/или 768 п.н. и/или 135 п.н. и/или 428 п.н. определяют наличие Atopobium vaginae и/или Leptotrichia amnionii и/или Sneathia sanguinegens и/или Eggerthella spp. соответственно.
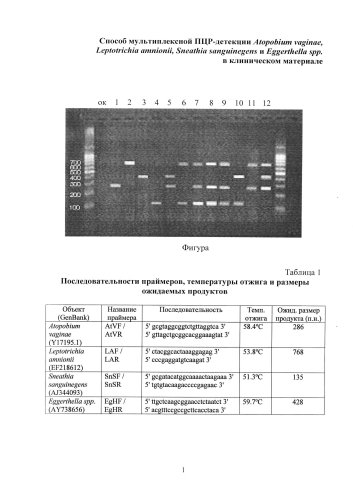
Изобретение иллюстрируется фигурой, на которой представлена электрофореграмма результатов амплификации с использованием мультиплексной тест-системы.
Подбор праймеров. В качестве ДНК-мишеней выбраны последовательности генов 16S рРНК. Выбор именно этих генов обусловлен доступностью огромных массивов данных о структуре генов рРНК у самых разнообразных микроорганизмов. Гены рРНК обнаружены у всех клеточных форм жизни, происходят от общего предка и функционально постоянны, генетически стабильны. Ген 16S рРНК имеет высококонсервативные и вариабельные участки. Использование в качестве мишеней для отжига праймеров высококонсервативных участков позволяет надежно идентифицировать искомые бактерии до вида, избегая ошибок, связанных с генетической изменчивостью.
Для конструирования тест-системы был осуществлен поиск последовательностей ДНК БВ-ассоциированных бактерий, представленных в международном банке нуклеотидных последовательностей GenBank (www.ncbi.nlm.nih.gov): Atopobium vaginae (Y17195.1), Leptotrichia amnionii (EF218612), Sneathia sanguinegens (AJ344093), Eggerthella spp. (AY738656). При использовании программы «MegAlign» (США) был проведен сравнительный анализ нуклеотидных последовательностей указанных микроорганизмов и обнаружены наиболее консервативные и видоспецифичные участки, которые позволяют с наибольшей достоверностью определять видовую принадлежность бактерий. С использованием программы «PrimerSelect» пакета компьютерных программ «Lasergene» («DNASTAR, Inc.», США) были подобраны пары олигонуклеотидных праймеров, отвечающие следующим требованиям: неспособность образовывать между собой стабильные димеры и формировать шпилечные структуры; температура отжига 55-70°C; 3′-конец праймера размером 10 нуклеотидов абсолютно комплементарен цепи ДНК; минимальная разность температур отжига праймеров одной пары; длина праймера 18-30 нуклеотидов. Далее осуществлялся поиск сходных последовательностей по всей базе данных генов с помощью программы «Megablast», доступной на вебсайте NCBI (www.ncbi.nlm.nih.gov), что позволило установить отсутствие гомологии фрагмента анализируемого гена исследуемого микроорганизма аналогичным генам других микроорганизмов. Это предполагает возможность их использования в качестве мишени и видоспецифичность подобранных последовательностей праймеров. Для идентификации генетического материала исследуемых микроорганизмов методом мультиплексной ПЦР были подобраны пары праймеры (таблица 1).
Предлагаемый способ детекции включает три этапа: выделение генетического материала, мультиплексную амплификацию (ПЦР) и электрофорез.
На первом этапе из клинического материала (отделяемое из заднего свода влагалища) выделяют тотальную ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и получения достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляют с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис». Набор реагентов «ДНК-сорб-АМ» - одна из модификаций метода Boom, максимально адаптированная к выделению ДНК из различных типов клинического материала для диагностики урогенитальных инфекций [Boom R., et al. 1990. Rapid and Simple Method for Purification of Nucleic Acids. J. Clin. Microbiol. 28: 495-503].
1. В пробирку с 100 мкл исследуемого материала вносят 300 мкл лизирующего раствора. Отдельным наконечником в пробирку вносят 25 мкл ресуспендированного сорбента.
2. Пробы тщательно перемешивают на вортексе и прогревают 5 мин при температуре 65°C. После окончания инкубации содержимое повторно перемешивают на вортексе и оставляют при комнатной температуре на 2 мин.
3. Сорбент осаждают центрифугированием при 10 тыс об/мин в течение 30 сек. Надосадочную жидкость удаляют с использованием вакуумного отсасывателя.
4. Далее в пробирку вносят 1 мл отмывочного раствора, перемешивают на вортексе до полного ресуспензирования сорбента.
5. Сорбент осаждают центрифугированием при 10 тыс об/мин в течение 30 сек. Надосадочную жидкость удаляют с использованием вакуумного отсасывателя.
6. Пробирки помещают в термостат с температурой 65°C на 5-10 мин для подсушивания сорбента, при этом крышки пробирок открыты.
7. В пробирки добавляют по 100 мкл ТЕ-буфера для элюции ДНК. Перемешивают на вортексе до полного ресуспензирования сорбента. Помещают в термостат с температурой 65°C на 5 мин.
8. Центрифугируют пробирки при 12 тыс об/мин в течение 1 мин на микроцентрифуге.
Надосадочная жидкость содержит препарат очищенной ДНК, готовый к постановке ПЦР. Сроки хранения препаратов: 1 неделя - при +2-8°C или 1 год - при минус 16°C.
II этап. Амплификация ДНК.
1. Полученный препарат ДНК вносят в реакционную смесь (25 мкл) следующего состава: 3 мкл исследуемой ДНК, 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е. каждый, таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносят по 1 капле стерильного минерального масла.
3. Амплификацию искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляют на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2). На первом этапе проводят денатурацию ДНК при 94°C в течение 1 мин, затем 30 циклов, каждый из которых включает стадию денатурации ДНК в течение 30 сек при 94°C, стадию отжига праймеров в течение 30 сек при температуре 59°C и стадию элонгации в течение 30 сек при температуре 72°C. На заключительном этапе реакционную смесь выдерживают при температуре 72°C в течение 2 мин.
III этап. Электрофоретический анализ продуктов ПЦР.
Анализ осуществляют с использованием стандартных наборов на сертифицированном оборудовании (Биоком, г. Москва) в 1,7% горизонтальном агарозном геле. В качестве электролита для электрофореза применяют 1х-ный ТБЕ буфер. Параметры электрофореза: сила тока 400 мА, мощность 80 Вт, напряжение 120 В. Электрофорез проводят в течение 25-30 минут. Детекцию результатов проводят путем окрашивания агарозного геля бромистым этидием с последующей визуализацией при освещении УФ на трансиллюминаторе «УВТ-1» (Биоком, Россия). Документирование результатов проводят с использованием системы для фотодокументации: цифровой видеокамеры «Mintron» и программы «Biotest-D» (Биоком, Россия). Продукты амплификации проявляются в виде светящейся оранжево-красной полосы. Для контроля длин продуктов амплификации используют соответствующий маркер длин ДНК 100+bp DNA Ladder (ЕвроГен), состоящий из 9 фрагментов ДНК в диапозоне 100-1000 п.н. и дополнительного фрагмента 1500 п.н.
Для интерпретации результатов анализа используют таблицу 1. При электрофоретическом обнаружении продукта амплификации размером 286 п.н. констатируют факт присутствия Atopobium vaginae в клиническом материале, при наличии ПЦР-продукта размером 768 п.н. - Leptotrichia amnionii, размер ПЦР-продукта 135 п.н. - указывает на присутствие Sneathia sanguinegens, размер ПЦР-продукта 428 п.н. - Eggerthella spp.
Сущность изобретения поясняется следующими примерами.
Пример 1.
I этап. Выделение тотальной ДНК.
Из клинического образца №1 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленными авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 1.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продукта амплификации размером 286 п.н. указывает на присутствие Atopobium vaginae в клиническом материале. Отсутствие других четких полос указывает на отсутствие Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. в клиническом материале.
Пример 2.
I этап. Выделение тотальной ДНК.
Из клинического образца №2 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 2.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продукта амплификации размером 768 п.н. указывает на присутствие Leptotrichia amnionii в клиническом материале. Отсутствие других четких полос указывает на отсутствие Atopobium vaginae, Sneathia sanguinegens и Eggerthella spp. в клиническом материале.
Пример 3.
I этап. Выделение тотальной ДНК.
Из клинического образца №3 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл), следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 3.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продукта амплификации размером 428 п.н. указывает на присутствие Eggerthella spp. B клиническом материале. Отсутствие других четких полос указывает на отсутствие Atopobium vaginae, Leptotrichia amnionii и Sneathia sanguinegens в клиническом материале.
Пример 4.
I этап. Выделение тотальной ДНК.
Из клинического образца №4 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 4.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продукта амплификации размером 135 п.н. указывает на присутствие Sneathia sanguinegens в клиническом материале. Отсутствие других четких полос указывает на отсутствие Atopobium vaginae, Leptotrichia amnionii и Eggerthella spp. в клиническом материале.
Пример 5.
I этап. Выделение тотальной ДНК.
Из клинического образца №5 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 5.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продуктов амплификации размером 286 п.н. и 428 п.н. указывает на присутствие Atopobium vaginae и Eggerthella spp. в клиническом материале. Отсутствие других четких полос указывает на отсутствие Leptotrichia amnionii и Sneathia sanguinegens в клиническом материале.
Пример 6.
I этап. Выделение тотальной ДНК.
Из клинического образца №6 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл), следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 6.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продуктов амплификации размером 135 п.н. и 768 п.н. указывает на присутствие Sneathia sanguinegens и Leptotrichia amnionii в клиническом материале. Отсутствие других четких полос указывает на отсутствие Atopobium vaginae и Eggerthella spp. в клиническом материале.
Пример 7.
I этап. Выделение тотальной ДНК.
Из клинических образцов №7, 8, 9 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 7, 8, 9.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продуктов амплификации размером 135 п.н., 286 п.н. и 768 п.н. указывает на присутствие Sneathia sanguinegens, Atopobium vaginae и Leptotrichia amnionii в клиническом материале. Отсутствие других четких полос указывает на отсутствие Eggerthella spp. в клиническом материале.
Пример 8.
I этап. Выделение тотальной ДНК.
Из клинического образца №10 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 10.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продуктов амплификации размером 135 п.н. и 428 п.н. указывает на присутствие Sneathia sanguinegens и Eggerthella spp. в клиническом материале. Отсутствие других четких полос указывает на отсутствие Atopobium vaginae и Leptotrichia amnionii в клиническом материале.
Пример 9.
I этап. Выделение тотальной ДНК.
Из клинического образца №11 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПНР.
Результаты представлены на фигуре, дорожка 11.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продуктов амплификации размером 286 п.н. и 768 п.н. указывает на присутствие Atopobium vaginae и Leptotrichia amnionii в клиническом материале. Отсутствие других четких полос указывает на отсутствие Sneathia sanguinegens и Eggerthella spp. в клиническом материале.
Пример 10.
I этап. Выделение тотальной ДНК.
Из клинического образца №12 (отделяемое из заднего свода влагалища) выделяется препарат тотальной ДНК. Выделение ДНК проводят в условиях, регламентированных Методическими указаниями МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеспечения необходимой степени чистоты ДНК и достаточного ее количества используют методику нуклеосорбции. Выделение ДНК осуществляли с использованием стандартного набора «ДНК-сорб-АМ» серии «АмплиПрайм» фирмы «Интерлабсервис».
II этап. Амплификация ДНК.
1. 3 мкл полученной ДНК вносят в реакционную смесь (25 мкл) следующего состава: 2.5 мкл 10×Taq - буфера, 2.5 мкл раствора dNTP, по 0.5 мкл каждого праймера (8 праймеров по 2 о.е., таблица 1), 0.5 мкл Taq-полимеразы и 12.5 мкл mQ.
2. Для предотвращения испарения в каждую пробирку дополнительно вносится 1 капля стерильного минерального масла.
3. Амплификация искомых фрагментов генов 16S рибосомальной РНК (рРНК) Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. осуществляется на амплификаторе «Терцик МС-2» («ДНК-технология», Россия) в соответствии с установленным авторами оптимальным режимом (таблица 2).
III этап. Электрофоретический анализ продуктов ПЦР.
Результаты представлены на фигуре, дорожка 12.
Для интерпретации результатов анализа используют таблицу 1. Обнаружение продуктов амплификации размером 286 п.н., 428 п.н. и 768 п.н. указывает на присутствие Atopobium vaginae, Eggerthella spp. и Leptotrichia amnionii в клиническом материале. Отсутствие других четких полос указывает на отсутствие Sneathia sanguinegens в клиническом материале.
Таким образом, предлагаемый способ мультиплексной ПЦР-детекции в клиническом материале позволяет с высокой чувствительностью и специфичностью выявлять и идентифицировать Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp.
Способ ПЦР-детекции Atopobium vaginae, Leptotrichia amnionii, Sneathia sanguinegens и Eggerthella spp. в клиническом материале, включающий выделение ДНК из исследуемого материала, амплификацию и электрофорез, отличающийся тем, что проводят мультиплексную ПЦР с использованием специфичных праймеров к Atopobium vaginae: 5′ gcgtaggcggtctgttaggtca 3′ и 5′ gttagctgcggcacggaaagtat 3′, к Leptotrichia amnionii: 5′ ctacggcactaaaggagag 3′ и 5′ cccgaggatgtcaagat 3′, к Sneathia sanguinegens: 5′ gcgatacatggcaaaactaagaaa 3′ и 5′ tgtgtacaagaccccgagaac 3′ и к Eggerthella spp.: 5′ ttgctcaagcggaacctctaatct 3′ и 5′ acgtttccgccgcttcacctaca 3′ в циклическом режиме, предусматривающем начальный цикл в течение 1 мин при температуре 94°C; затем 30 циклов, каждый из которых включает стадию денатурации в течение 30 с при 94°C, стадию отжига праймеров в течение 30 с при температуре 59°C и стадию элонгации в течение 30 с при температуре 72°C; заключительный цикл в течение 2 мин при температуре 72°C, при электрофоретическом обнаружении ампликонов размерами 286 п.н., и/или 768 п.н., и/или 135 п.н., и/или 428 п.н. определяют наличие Atopobium vaginae, и/или Leptotrichia amnionii, и/или Sneathia sanguinegens, и/или Eggerthella spp. соответственно.