Способ и устройство для анализа взаимодействий биологических молекул на биологическом микрочипе на основе флуоресценции аминокислотных остатков триптофана
Иллюстрации
Показать всеГруппа изобретений относится к молекулярной биологии, биофизике, биохимии и биотехнологии и касается способа анализа взаимодействий между молекулами, флуоресцирующими в ультрафиолетовой (УФ) области спектра, включая белки, пептиды, гликопротеины, протеогликаны и другие соединения, последовательность которых содержит один или более аминокислотных остатков триптофана (Trp), и биологическими молекулами, иммобилизованными в ячейках биологического микрочипа (биочипа), при котором анализ взаимодействий производится на основе измерения интенсивности УФ-флуоресценции аминокислотных остатков триптофана. Группа изобретений также касается устройства для осуществления указанного способа анализа в УФ области спектра. Группа изобретений обеспечивает возможность исключения применения флуоресцентных меток для регистрации связывания и обеспечивает анализ взаимодействия биологических молекул с молекулами на биочипе по УФ-флуоресценции природного флуорофора - аминокислотного остатка триптофана. 2 н. и 6 з.п. ф-лы, 2 пр., 8 ил.
Реферат
Область техники, к которой относится изобретение
Изобретение относится к молекулярной биологии, биофизике, биохимии, биоорганической химии, биотехнологии и фармакологии и предлагает способ анализа взаимодействий между биологическими молекулами (нуклеиновыми кислотами, углеводородами, липидами, пептидами и белками), иммобилизованными в ячейках биологического микрочипа (биочипа), и молекулами, флуоресцирующими в ультрафиолетовой (УФ) области спектра, включая белки, пептиды, гликопротеины, протеогликаны и другие соединения, последовательность которых содержит один или более аминокислотных остатков триптофана (Trp). Биологические молекулы, например, молекулы нуклеиновых кислот, иммобилизованные в разных ячейках биочипа, взаимодействуют с раствором анализируемого вещества, например белка, аминокислотная последовательность которого содержит один или более аминокислотных остатков триптофана. Интенсивность флуоресценции белка на биочипе измеряют в УФ области спектра (300-400 нм) с помощью специализированного анализатора изображений биочипов, представляющего собой широкопольный высокоапертурный люминесцентный микроскоп с кварцевой оптикой, оборудованный ПЗС (CCD) камерой, чувствительной в УФ области, и управляемый с помощью компьютера со специализированной программой анализа изображений. Применение изобретения позволяет анализировать взаимодействие белков с различными нуклеиновыми кислотами или их фрагментами, не прибегая к предварительной процедуре введения в молекулы белка флуоресцентной метки, либо к дополнительной процедуре связывания с флуоресцентно меченными антителами.
Уровень техники
Биочипом называется матрица микроячеек, регулярно расположенных на плоской подложке. Размер ячеек и расстояние между ними могут варьироваться от нескольких микрон до сотен микрон. В каждой ячейке биочипа иммобилизованы молекулы одного типа (молекулярные зонды), способные с высокой степенью специфичности связываться с определенным типом биомолекул исследуемого раствора, наносимого на поверхность биочипа, и образовывать комплексы. В случае, когда анализируемое вещество предварительно было помечено флуоресцирующим красителем, взаимодействие его с молекулами зондов определяется по флуоресценции соответствующих ячеек биочипа (Колчинский A.M., Грядунов Д.А., Лысов Ю.П., Михайлович В.М., Наседкина Т.В., Турыгин А.Ю., Рубина А.Ю., Барский В.Е. и Заседателев А.С. 2004. Микрочипы на основе трехмерных ячеек геля: история и перспективы. Молекулярная биология, 38: 5-16; Venkatasubbarao S. 2004. Microarrays-status and prospects. Trends Biotechnol. 22: 630-7).
ДНК- и РНК-связывающие белки играют важную роль в различных клеточных процессах, таких как регуляция транскрипции, трансляции, рекомбинация, репликация, модификация ДНК и РНК. В настоящее время специфичность узнавания для большинства ДНК- и РНК-связывающих белков остается невыясненной. Данные о специфичных сайтах связывания могут быть использованы для того, чтобы предсказать функцию данного белка, возможные гены-мишени, выяснить роль, которую занимает белок в регуляторных механизмах клетки.
Биологические микрочипы широко используют для изучения связывания белков с нуклеиновыми кислотами in vitro. Zasedateleva О.А., Mikheikin A.L., Turygin A.Y., Prokopenko D.V., Chudinov A.V., Belobritskaya E.E., Chechetkin V.R. and Zasedatelev A.S. 2008. Gel-based oligonucleotide microarray approach to analyze protein-ssDNA binding specificity. Nucleic Acids Res. 36: e61. Такой подход, по сравнению с традиционными методами исследования, такими как EMSA (electrophoretic mobility shift assay), а также рядом других, позволяет в одном эксперименте и в одинаковых условиях, оценить сродство связывания исследуемого объекта: белка или лиганда, к определенному набору олигонуклеотидных последовательностей.
В настоящее время разработаны следующие платформы для исследования ДНК-белковых взаимодействий, которые позволяют на одном микрочипе исследовать сродство анализируемого белка к определенным нуклеотидным последовательностям.
1. В отечественном исследовании ДНК-белковых взаимодействий in vitro использовался биочип с трехмерными гидрогелевыми ячейками (3D биочип), содержащий полный перебор всех возможных однонитевых последовательностей ДНК длиной 6 оснований, иммобилизованных в объеме гидрогелевых ячеек. Zasedateleva О.А., Mikheikin A.L., Turygin A.Y., Prokopenko D.V., Chudinov A.V., Belobritskaya E.E., Chechetkin V.R. and Zasedatelev A.S. 2008. Gel-based oligonucleotide microarray approach to analyze protein-ssDNA binding specificity. Nucleic Acids Res. 36: e61. В качестве объекта исследования была использована рибонуклеаза биназа. Биочип содержал 46=4096 ячеек, в каждой из которых были иммобилизованы олигодезоксирибонуклеотиды с фиксированной 6-нуклеотидной последовательностью. Для анализа ДНК-белкового взаимодействия белок ковалентно метили красителем Texas Red. Флуоресцентные сигналы красителя детектировали с помощью широкопольного флуоресцентного микроскопа с цифровой регистрацией на ПЗС камеру. Сродство связывания определяли по термодинамическому параметру - значению равновесной температуры диссоциации комплекса белок-олигонуклеотид микрочипа. Измеряли интенсивности флуоресценции различных ячеек биочипа, содержащих связанные с зондами молекулы биназы, в диапазоне температур от 0 до 60 градусов Цельсия и анализировали полученные кривые диссоциации: чем выше сродство белка к последовательности, тем больше значение температуры диссоциации соответствующего комплекса. Была продемонстрирована возможность определения специфичности связывания на 3D биочипе по значениям флуоресцентных сигналов при 40°С.
Для изучения ДНК-белковых и РНК-белковых взаимодействий часто используют двумерные (2D) биологические микрочипы, олигонуклеотиды которых иммобилизованы на модифицированной поверхности подложки.
2. Микрочип, содержащий полный перебор всех возможных ДНК дуплексов длиной 8 пар оснований, был применен для исследования сиквенс-специфичности связывания фактора транскрипции Exd (Drosophila) в комплексе с полиамид-пептидными коньюгатами. Warren C.L., Kratochvil N.C., Hauschild K.Е., Foister S., Brezinski M.L., Dervan P.B., Phillips G.N. Jr. and Ansari A.Z. 2006. Defining the sequence-recognition profile of DNA-binding molecules. Proc. Natl. Acad. Sci. 103: 867-72. На микрочипе синтезировали олигодезоксирибонуклеотиды длиной 32 основания вида GCGN1N2N3N4N5N6N7N8GCGTCCTCGCN8N7N6N5N4N3N2N1CGC, где N1N2N3N4N5N6N7N8 одна из всех возможных, 48=65536 последовательностей длиной 8 нуклеотидов. Такая последовательность содержала два самокомплементарных участка, которые образовывали дуплексы-шпильки. Всего микрочип содержал 48/2=32768 различных олигонуклеотидов в четырех повторах. Для детекции ДНК-белкового взаимодействия к анализируемому белку ковалентно присоединяли одну молекулу красителя Су3. Флуоресцентные сигналы красителя детектировали с помощью анализатора микрочипов на основе лазерного сканера.
3. Биочип, содержащий полный перебор всех возможных однонитевых последовательностей ДНК длиной 6 нуклотидов, использовали для исследования сиксенс-специфичности связывания белка холодового шока CspB бактерии Bacillus subtilis. Morgan Н.Р., Estibeiro P., Wear M.A., Max K.E., Heinemann U., Cubeddu L., Gallagher M.P., Sadler P.J. and Walkinshaw M.D. 2007. Sequence specificity of single-stranded DNA-binding proteins: a novel DNA microarray approach. Nucleic Acids Res. 35: e75. Биочип содержал 46=4096 ячеек, в каждой из которых были иммобилизованы олигодезоксирибонуклеотиды с фиксированной 6-нуклотидной последовательностью по свободному 3′-концу. Белок CspB содержал полигистидиновый эпитопный таг (аминокислотная последовательность, которая добавлена к белку с С либо N конца, и которая специфично связывается с антителом). ДНК-белковое взаимодействие детектировали с помощью меченного флуорофором Alexa 532 поликлонального антитела, специфичного к полигистидиновому тагу. Флуоресцентные сигналы детектировали с помощью лазерного сканера.
4. Berger M.F. с соавторами использовали биочип, содержащий все возможные сайты связывания ДНК длиной 10 пар основании (при этом одна ячейка содержала 26 перекрывающихся сайтов длиной 10 пар оснований на отрезке в 35 пар оснований), для исследования сиквенс-специфичности связывания пяти факторов транскрипции из различных структурных классов, из разных организмов: Cbf1 дрожжей S. cerevisiae (содержит основной ДНК-связывающий домен спираль-петля-спираль), Rap1 дрожжей S. cerevisiae (содержит Myb домен), Ceh-22 из червей Caenorhabditis elegans (содержит NK гомеодомен), Zif268 мыши (содержат Cys2His2 домен цинкового пальца) и Oct-1 человека (содержит POU ДНК-связывающий гомеодомен). Berger M.F., Philippakis А.А., Qureshi A.M., He F.S., Estep P.W. 3rd. and Bulyk M.L. 2006. Compact, universal DNA microarrays to comprehensively determine transcription-factor binding site specificities. Nat Biotechnol. 24: 1429-35. Биочип содержал иммобилизованные 60-нуклеотидные молекулы однонитевой ДНК с общей последовательностью длиной 25 нуклеотида и вариабельной частью длиной 35 нуклеотидов. Олигонуклеотиды переводили в двунитевую форму с помощью реакции праймерного роста. Вариабельная часть длиной 35 пар оснований была построена по принципу последовательности де Брейна. Каждая ячейка биочипа содержала 35-10+1=26 перекрывающихся последовательностей длиной 10 пар оснований на вариабельном отрезке длиной 35 пар оснований. Всего биочип содержал 410/26=40330 ячеек. Любая последовательность длиной 10 пар оснований в ряду этих ячеек повторялась один раз. Все пять факторов транскрипции содержали GST (глутатион-S-трансфераза, 220 аминокислот, 26 кDа) таг по N-концу. ДНК-белковое взаимодействие детектировали по флуоресценции красителя Alexa 488, связанного с поликлональным антителом специфичным к GST тагу. Флуоресцентные сигналы флуоресцентно меченного антитела детектировали с помощью сканера. Для определения сродства белка к последовательностям применяли метод статистической обработки полученных сигналов, в котором для каждой последовательности длиной 8 оснований рассматривали массив ячеек, содержащий данную последовательность, и остальной массив ячеек. Обработку данных проводили с применением статистики Wilcoxon-Mann-Whitney.
5. Основываясь на платформе, представленной в п. 4 (4×40330 биочип, белок с GST тагом), была создана база данных по сиквенс-специфичности связывания 175 факторов транскрипции из различных организмов. Newburger D.E. and Bulyk M.L. 2009. UniPROBE: an online database of protein binding microarray data on protein-DNA interactions. Nucleic Acids Res. 37 (Database issue): D77-82. К настоящему времени база данных содержит данные по сиквенс-специфичности связывания для 406 белков. Например, для фактора транскрипции LuxR, активирующего и репрессирующего экспрессию большого количества генов морской бактерии Vibrio harveyi, была определена консенсусная последовательность специфичного связывания длиной 21 пары оснований, в которой два критических для связывания сайта длиной 4 пары оснований разделены неспецифичным участком длиной 8 пар оснований. Pompeani A.J., Irgon J.J., Berger M.F., Bulyk M.L., Wingreen N.S. and Bassler B.L. 2008. The Vibrio harveyi master quorum-sensing regulator, LuxR, a TetR-type protein is both an activator and a repressor: DNA recognition and binding specificity at target promoters. Mol Microbiol. 70:76-88. Сохранение активности GST-LuxR белка подтверждали in vitro и in vivo. Анализ генома бактерии Vibrio harveyi выявил 36 возможных сайтов связывания. Для пяти из них, являющихся наиболее предпочтительными последовательностями для связывания и входящих в состав промотерных участков генов бактерии Vibrio harveyi, были определены уровни экспрессии соответствующих генов в конструкции Е. coli по флуоресценции зеленого флуоресцентного белка (GSP).
Zhu С. с соавторами, используя ту же платформу, определили последовательности специфичного связывания для 89 факторов транскрипции (из 246 кандидатов выделенных для анализа) дрожжей S. cerevisiae. Zhu С., Byers K.J., McCord R.P., Shi Z., Berger M.F., Newburger D.E., Saulrieta K., Smith Z., Shah M.V., Radhakrishnan M., Philippakis A.A., Hu Y., De Masi F., Pacek M., Rolfs A., Murthy Т., Labaer J. and Bulyk M.L. 2009. High-resolution DNA-binding specificity analysis of yeast transcription factors. Genome Res. 19: 556-66.
6. В другой работе с использованием биочипов, аналогичных описанным в п. 4, были проанализированы представители семейства фактров транскрипции NF-κB, контролирующих экспрессию генов иммунного ответа, апоптоза и клеточного цикла. Wong D., Teixeira A., Oikonomopoulos S., Humburg P., Lone I.N., Saliba D., Siggers Т., Bulyk M., Angelov D., Dimitrov S., Udalova I.A. and Ragoussis J. 2011. Extensive characterization of NF-κВ binding uncovers non-canonical motifs and advances the interpretation of genetic functional traits. Genome Biol. 12(7): R70. NF-κВ белки были модифицированы гистидиновым тагом, а полученные комплексы белок-ДНК проявлены флуоресцентно меченными специфичный к гис-тагу антителами. Было получено, что NF-κВ белки не только специфично связываются с каноническими последовательностями типа GGGGGATTTCC, но и некоторые из них, например RELARELA димер, специфично узнает последовательность AGGAANTTTCC. Были проанализированы все возможные сайты связывания этих белков в геноме, а также влияние точечных полиморфизмов на связывание белков NF-κВ, некоторые из которых ведут к аутоиммунным заболеваниям.
Еще одним in vitro методом для исследования взаимодействий нуклеиновых кислот, белков и лигандов с белками на биочипе является метод, основанный на использовании эффекта поверхностного плазмонного резонанса (Surface Plasmon Resonance, SPR). Этот подход позволяет характеризовать количественно, в терминах кинетических и термодинамических параметров, связывание биомолекул на биочипе. Например, на сенсорных биочипах исследуют ДНК- и РНК-белковое взаимодействие. Rajendran, K.S. and Nagy, P.D. 2003. Characterization of the RNA-Binding Domains in the Replicase Proteins of Tomato Bushy Stunt Virus. J. Virol. 77: 9244-9258. Peters H., Kusov Y.Y., Meyer S., Benie A.J., ., Wolff M., Rademacher C., Peters T. and V. 2005. Hepatitis A virus proteinase 3C binding to viral RNA: correlation with substrate binding and enzyme dimerization. Biochem J. 385(Pt 2):363-70. Majka J and Speck C. 2007. Analysis of protein-DNA interactions using surface plasmon resonance. Adv Biochem Eng Biotechnol. 104: pp 13-36. Недостатком сенсорного биочипа является то, что он содержит очень малое количество иммобилизованных зондов для связывания биомолекул, и, таким образом, позволяет в одном эксперименте исследовать сродство связывания белка лишь к небольшому числу лигандов. Поэтому метод измерения взаимодействий биомолекул на биочипе с использованием эффекта поверхностного плазмонного резонанса по количеству лигандов, сродство белка к которым измеряется в одном эксперименте, сопоставим с традиционными методами исследования взаимодействий нуклеиновых кислот с белками (EMSA, измерение анизотропии флуоресценции и др.).
Известно, что при возбуждении в ультафиолетовой (УФ) области спектра ароматические аминокислоты - триптофан, тирозин и фенилаланин - флуоресцируют в УФ диапазоне. При этом в нейтральных водных растворах их спектры флуоресценции характеризуются следующими значениями длин волн в максимумах: триптофан - 348 нм, тирозин - 303 нм, фенилаланин - 282 нм. Teale F.W. and Weber G. 1957. Ultraviolet fluorescence of the aromatic amino acids. Biochem J. 65:476-482. Один или более аминокислотных остатков триптофана содержится в большинстве белков. Существует несколько возможных процессов и факторов - тушение, резонансный перенос энергии, более низкое поглощение, более низкий квантовый выход, - из-за которых в белках, аминокислотная последовательность которых содержит все три ароматических аминокислотных остатка, флуоресцентные составляющие фенилаланина и тирозина отсутствуют, а присутствует лишь флуоресцентная составляющая триптофана. Teale F.W. J. 1960. The ultraviolet fluorescence of proteins in neutral solution. Biochem J. 76:381-388. Chen Y. and Barkley M.D. 1998. Toward understanding tryptophan fluorescence in proteins. Biochemistry. 37:9976-9982. Mocz G. and Ross J.A. 2013. Fluorescence techniques in analysis of protein-ligand interactions, p. 169-210. In M.A. Williams, and T. Daviter (Eds.), Protein-Ligand Interactions: Methods and Applications, Methods in Molecular Biology, Springer Science + Business Media, New York.
Недостатки существующих методик исследования взаимодействий биологических молекул на биочипе.
В существующих методиках исследования взаимодействия между биологическими молекулами, иммобилизованными в ячейках биочипа (нуклеиновыми кислотами, углеводородами, липидами, пептидами, белками), и другими биологическими молекулами, включая белки, пептиды, гликопротеины и протеогликаны, для обеспечения возможности количественной регистрации связывания необходимо либо предварительно модифицировать анализируемые неиммобилизованных биомолекулы с помощью ковалентного присоединения к ним флуоресцентного красителя, либо использовать флуоресцентно меченные антитела, специфичные к анализируемым неиммобилизованным биомолекулам.
Ковалентное присоединение флуоресцентного красителя к анализируемой биологической молекуле (белку, пептиду, гликопротеину, протеогликану) может повлиять на специфичность анализируемой биомолекулы, что является существенным недостатком данного существующего подхода. Поэтому обычно проводят контроль сохранения свойств модифицированной молекулы, по сравнению с немодифицированной (примеры 1 и 2 раздела "Уровень техники").
Существенным недостатком использования флуоресцентно меченных антител для количественной регистрации связывания биомолекул на биочипе является то, что такой подход делает невозможным изучение образования комплексов в динамическом равновесии. Также в данном подходе обычно анализируемые белки и пептиды содержат по N-, либо С-концу дополнительную аминокислотную последовательность - эпитопный таг, - специфичную для связывания флуоресцентно меченного антитела (примеры 3-6 раздела "Уровень техники"). Поэтому при таком подходе также обычно необходимо проводить контроль свойств модифицированного тагом белка и пептида по сравнению с немодифицированным.
В основу предлагаемого изобретения положена задача разработать основанный на регистрации флуоресценции метод и устройство, позволяющие исследовать на биочипе связывание биологических молекул (белков, пептидов, гликопротеинов, протеогликанов и других соединений), флуоресцирующих в УФ диапазоне, с иммобилизованными биологическими молекулами (нуклеиновыми кислотами, углеводородами, липидами, пептидами, белками), без введения дополнительных флуоресцентных меток или использования флуоресцентно меченных антител. Поставленная задача решается предлагаемым изобретением.
Раскрытие изобретения
Настоящее изобретение предлагает способ и устройство для анализа взаимодействий между биологическими молекулами (нуклеиновыми кислотами, углеводородами, липидами, пептидами и белками), иммобилизованными в ячейках биологического микрочипа, и молекулами, флуоресцирующими в УФ области спектра, включая белки, пептиды, гликопротеины, протеогликаны и другие соединения, последовательность которых содержит один или более аминокислотных остатков триптофана (Trp). Биологические молекулы, например молекулы нуклеиновых кислот, иммобилизованные в разных ячейках биочипа, взаимодействуют с раствором анализируемого вещества, например белка, аминокислотная последовательность которого содержит один или более аминокислотных остатков триптофана (Trp). Интенсивность флуоресценции флуорофор-содержащих аминокислотных остатков триптофана на биочипе измеряют в УФ области спектра (300-400 нм) с помощью специализированного анализатора биочипов, приспособленного для работы в УФ диапазоне (возбуждение 250-300 нм, флуоресценция 300-400 нм). Применение изобретения позволяет исключить использование флуоресцентных меток для регистрации связывания и анализировать взаимодействие биологических молекул, с биологическими молекулами на биочипе по УФ флуоресценции природного флуорофора - аминокислотного остатка триптофана.
В качестве подложки биочипа для анализа взаимодействия биологических молекул на биочипе на основе флуоресценции в УФ диапазоне можно использовать стекло, кварцевое стекло, сапфир, керамику, металл, пластик, а также подложку, покрытую слоем металла.
Крышка камеры, покрывающей биочип для анализа взаимодействия биологических молекул на биочипе на основе флуоресценции в УФ диапазоне, может быть изготовлена из кварцевого стекла.
Анализ взаимодействий между молекулярными зондами биочипа и биологическими молекулами анализируемого вещества в УФ диапазоне можно проводить в случае любых молекул анализируемого вещества (белков, пептидов, гликопртеинов, протеогликанов и других соединений), последовательность которых содержит по крайней мере один аминокислотный остаток триптофана (Trp).
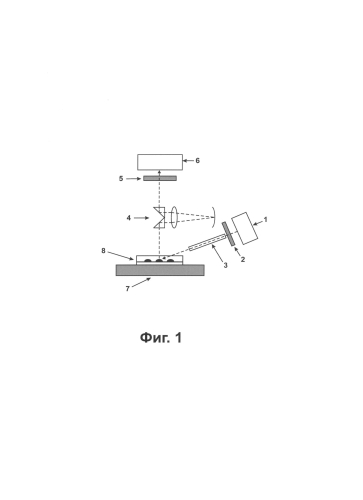
В одном из воплощений способа и устройства для анализа взаимодействия биологических молекул на биочипе на основе флуоресценции в УФ диапазоне в качестве анализатора изображения биочипа можно использовать флуоресцентный микроскоп с кварцевой оптикой, который оснащен источником ультрафиолетового излучения (например, газоразрядной лампой, излучающей в области 250-300 нм), набором фильтров и светоприемником, позволяющим регистрировать испускаемое объектом излучение в диапазоне длин волн 300-400 нм.
В одном из воплощений способа и устройства для анализа взаимодействия биологических молекул на биочипе на основе флуоресценции в УФ диапазоне в качестве анализатора изображения биочипа можно использовать сканер, позволяющий регистрировать флуоресценцию в области 300-400 нм при возбуждении в области 250-300 нм.
В одном из воплощений способа анализа взаимодействия биологических молекул на биочипе на основе флуоресценции в УФ диапазоне можно использовать биочип, который представляет собой подложку с расположенными на ней гелевыми ячейками, в объеме которых иммобилизованы молекулярные зонды.
Таким образом, технический результат настоящего изобретения выражается в визуализации и количественном анализе немодифицированных биомолекул на биочипе. Технический результат достигается за счет применения специализированного способа иммобилизации нуклеиновых кислот и белков в сочетании с новым способом их анализа, основанным на регистрации УФ излучения анализируемых объектов с помощью разработанного широкопольного высокоапертурного цифрового люминесцентного УФ микроскопа.
Далее настоящее изобретение будет подробно раскрыто со ссылкой на чертежи.
Краткое описание чертежей
Фиг. 1. Схема флуоресцентного микроскопа, предназначенного для анализа взаимодействий между молекулами, флуоресцирующими в ультрафиолетовой (УФ) области спектра, и биологическими молекулами, иммобилизованными в ячейках биологического микрочипа.
Фиг. 2. Спектральные характеристики биомолекул (белка Бычий Сывороточный Альбумин (BSA), фермента биназы, аминокислоты триптофан) и фильтров, используемых при регистрации флуоресценции белков в УФ диапазоне на биочипе.
1 - Нормированный спектр возбуждения флуоресценции BSA, записанный при фиксированной длине волны излучения 345 нм.
2 - Нормированный спектр флуоресценции BSA, записанный при облучении белка на фиксированнной длине волны возбуждения 280 нм.
3 - Нормированный спектр флуоресценции фермента биназы (сплошная линия), записанный при облучении белка на фиксированнной длине волны возбуждения 280 нм.
4 - Нормированный спектр флуоресценции аминокислоты триптофан (прерывистая линия), записанный при облучении аминокислоты на фиксированнной длине волны возбуждения 280 нм.
5 - Спектр пропускания фильтра 280ВР30 (Omega Optical, США) в области длин волн возбуждения (λмакс.=280 нм).
6 - Спектр пропускания фильтра ФС1 (Россия) в области длин волн излучения (λмакс.=395 НМ).
7 - Спектр флуоресценции BSA (2), нормированный на спектр пропускания фильтра ФС1 (6).
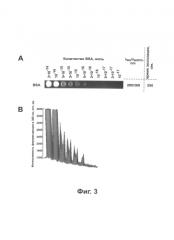
Фиг. 3. (А) Изображение ячеек биочипа, расположенных на зеркальной поверхности слоя алюминия и содержащих разное количество молекул белка BSA. Изображение получено при облучении длинами волн с максимумом на 280 нм и регистрацией испускаемого излучения с максимумом на длине волны 360 нм. Условия регистрации дополнительно указаны справа от изображения. Содержание BSA в ячейках в молях указано над изображением биочипа. (Б) Распределение величины интенсивности флуоресценции по площади изображения Фиг. 3А для первых семи ячеек биочипа Фиг. 3А.
Фиг. 4. Изображение биочипа, в гелевых ячейках которого иммобилизован белок BSA. (А) Изображение биочипа, полученное в области ультрафиолетовых длин волн (ближнем УФ диапазоне), при облучении биочипа длинами волн с максимумом в районе 280 нм и регистрацией испускаемого излучения с максимумом на длине волны 360 нм в течение 50 секунд. Условия регистрации дополнительно указаны справа от изображения биочипа, количество BSA в гелевых ячейках указано над изображением, тип иммобилизованных молекул BSA указан слева от изображения. Ковалентно меченный красителем Техасский красный (Texas Red) белок, BSA-TR, содержит в среднем 0.4 остатка молекулы красителя на одну молекулу альбумина. Над изображением биочипа показано распределение величины интенсивности флуоресценции по площади изображения биочипа для первых семи столбцов ячеек. (Б) Изображение для первых семи столбцов ячеек того же биочипа, что и на Фиг. 4А, полученное с выдержкой 0.2 секунды при облучении биочипа потоком света с максимумом на длине волны 580 нм и регистрацией испускаемого излучения с максимумом на длине волны 630 нм. (В) Изображение для последних четырех столбцов ячеек того же биочипа, что и на Фиг. 4А, полученное с выдержкой 30 секунд при облучении биочипа потоком света с максимумом на длине волны 580 нм и регистрацией испускаемого излучения с максимумом на длине волны 630 нм. Условия регистрации изображений (Б) и (В) дополнительно указаны справа от рисунка. Количество и тип иммобилизованных молекул BSA такие же, как и на Фиг. 4А. Под изображениями биочипа (Б) и (В) показаны распределения величины интенсивности флуоресценции по площади изображения биочипа.
Фиг. 5. Изображение биочипа, содержащего иммобилизованные в гелевых ячейках олигодезоксирибонуклеотиды, после инкубации с немодифицированным белком рибонуклеазой биназой при температуре 20°С. Возбуждение флуоресценции - 280 нм (фильтр - 280ВР30), регистрация - 360 нм (фильтр - ФС1), выдержка 10 секунд. Изображение зарегистрировано через 30 мин после начала инкубации, которые необходимы для достижения равновесия во взаимодействии биназы с олигодезоксирибонуклеотидами биочипа. Последовательности иммобилизованных октадезоксирибонуклеотидов указаны над изображением биочипа. Под изображением биочипа показано распределение величины интенсивности флуоресценции по площади изображения биочипа.
Фиг. 6. Изображение биочипа, представленного на Фиг. 5, после повышения температуры биочипа с 20°С до 40°С и последующей инкубации с немодифицированным белком рибонуклеазой биназой при 40°С в течение 30 мин. Выдержка - 10 с. Условия возбуждения и регистрации флуоресценции те же, что и на Фиг. 5. Под изображением биочипа показано распределение величины интенсивности флуоресценции по площади изображения биочипа.
Фиг. 7. Зарегистрированные в УФ диапазоне нормированные равновесные кривые температурной диссоциации комплексов немодифицированного белка рибонуклеазы биназы с олигодезоксирибонуклеотидами, иммобилизованными в гелевых ячейках биочипа. Последовательности иммобилизованных октадезоксирибонуклеотидов указаны справа от кривых диссоциации. Штрих-линией показана оценка величины температуры диссоциации комплекса биназа - олигонуклеотид 5′-NTTTTTCN-3′, при которой половина иммобилизованных в ячейке олигонуклеотидов находится в комплексе с биназой, а нормированный флуоресцентный сигнал в ячейке составляет, соответственно, 0.5 отн. ед.
Фиг. 8. Сравнение специфичности связывания немодифицированной рибонуклеазы биназы с олигодезоксирибонуклеотидами биочипа, измеренной по флуоресценции флуорофор-содержащих аминокислотных остатков биназы в ультрафиолетовой области, с ранее опубликованными данными по взаимодействию с олигонуклеотидами биочипа рибонуклеазы биназы, ковалентно меченной одним остатком флуоресцентного красителя Техасский красный (Zasedateleva О.А., Mikheikin A.L., Turygin A.Y., Prokopenko D.V., Chudinov A.V., Belobritskaya E.E., Chechetkin V.R. and Zasedatelev A.S. 2008. Gel-based oligonucleotide microarray approach to analyze protein-ssDNA binding specificity. Nucleic Acids Res. 36: e61. Supplementary table 1). (А) Гистограмма распределения значений флуоресцентных сигналов при 40°С для комплексов между немодифицированной биназой и олигонуклеотидами иммобилизованными в гелевых ячейках биочипа (серые столбцы) в сравнениии с гистограммой распределения этих значений для биназы, меченной флуорофором (белые столбцы). Последовательности иммобилизованных октадезоксирибонуклеотидов те же, что на Фиг. 5 и Фиг. 6 и расположены в том же порядке. Значения флуоресцентных сигналов нормированы на значение флуоресцентного сигнала для олигонуклеотида 5′-NAGTGTGN-3′. (Б) Гистограмма распределения значений температур диссоциации комплексов биназы с различными олигонуклеотидами, иммобилизованными в ячейках биочипа, в случае анализа немодифицированной биназы (серые столбцы), и биназы, меченной Техасский красным (белые столбцы). Последовательности олигонуклеотидов расположены в том же порядке, что и на гистограмме (А).
Осуществление изобретения
Настоящее изобретение предлагает способ анализа взаимодействия биологических молекул с биологическими молекулами - молекулярными зондами, - иммобилизованными в ячейках биочипа, на основе флуоресценции остатка аминокислоты триптофана (Trp) в УФ диапазоне, и устройство для осуществления такого анализа.
В качестве биочипов могут быть использованы двумерные (2D) биочипы, молекулярные зонды которых иммобилизованы на поверхности подложки, или трехмерные (3D) биочипы, молекулярные зонды которых иммобилизованы в объеме гелевых ячеек, нанесенных на подложку.
Молекулярными зондами, иммобилизованными в ячейках биочипа могут быть нуклеиновые кислоты, углеводороды, липиды, белки, пептиды, гликопротеины и протеогликаны, а также их комплексы. Перечисленные биомолекулы могут быть иммобилизованы на биочипе ковалентным способом, например, за счет образования ковалентной связи между биомолекулой и поверхностью подложки биочипа, и нековалентным способом, например, за счет образования связи стрептавидин-биотин, или, например, посредством физической адсорбции. . 2009. Immobilization strategies for small molecule, peptide and protein microarrays. J Pept Sci. 15:393-7. Vegas A.J., Fuller J.H. and Koehler A.N. 2008. Small-molecule microarrays as tools in ligand discovery. Chem Soc Rev. 37:1385-94. Rusmini F., Zhong Z. and Feijen J. 2007. Protein immobilization strategies for protein biochips. Biomacromolecules. 8: 1775-89. Taylor S., Smith S., Windle В., and Guiseppi-Elie A. 2003. Impact of surface chemistry and blocking strategies on DNA microarrays. Nucleic Acids Res. 31: e87. Kumar A., Larsson O., Parodi D. and Liang Z. 2000. Silanized nucleic acids: a general platform for DNA immobilization. Nucleic Acids Res. 28:E71. Afanassiev V., Hanemann V. and Wolfl S. 2000. Preparation of DNA and protein micro arrays on glass slides coated with an agarose film. Nucleic Acids Res. 28:E66. End-linked amino-modified 50-mer oligonucleotides as RNA profiling probes on nylon arrays: comparison to UV cross-linked DNA probes. BioTechniques 38: 690-694. Xavier K.A., Eder P.S. and Giordano T. 2000. RNA as a drug target: methods for biophysical characterization and screening. Trends Biotechnol. 18: 349-356; Verhelst S.H., Michiels P.J., van der Marel G.A., van Boeckel C.A. and van Boom J.H. 2004. Surface plasmon resonance evaluation of various aminoglycoside-RNA hairpin interactions reveals low degree of selectivity. Chembiochem. 5: 937-942. Jones, K.D. September 2001. Membrane immobilization of nucleic acids, Part 2: Probe Attachment Techniques. IVD Technology.
В качестве нуклеиновых кислот для иммобилизации на биочипе может быть использована ДНК и РНК любой последовательности и длины, однонитевая без вторичных структур, однонитевая, содержащая вторичные структуры, двунитевая, ДНК, представляющая собой ПЦР-амплифицированные участки генома функциональные и нефункциональные, кДНК (комплементарная, cDNA (complementary)), кРНК (комплементарная, cRNA (complementary)), мРНК (информационная, mRNA (messenger)), тРНК (транспортная, tRNA (transfer)), тмРНК (транспортно-матричная, tmRNA (transfer-messenger)), рРНК (рибосомная, rRNA (ribosomal)), мяРНК (малая ядерная, snRNA (small nuclear)), snoRNA (малая ядрышковая, snoRNA (small nucleolar)), мцРНК (малая цитоплазматическая, scRNA (small citoplasmic)), гяРНК (гетерогенная ядерная, hnRNA (heterogeneous nuclear)), химически синтезированные олигодезокси- и олигорибонуклеотиды. Иммобилизованные на биочипе нуклеиновые килоты могут содержать модификации (Mikheikin A.L., Surzhikov S.A., Zasedateleva О.А., Vasiliskov V.A., Pan′kov S.M., Grechishnikova I.V., Kisselev L.L., Zasedatelev A.S. 2008. An RNA microchip containing immobilized oligoribonucleotides with protective groups at 2′-O-positions. Biotechniques. 44:77-83. Заседателев A.C., Михейкин А.Л., Суржиков С.А., Заседателева О.А. Биологический микрочип с иммобилизованными олигорибонуклеотидами, способ его изготовления и способ анализа взаимодействия РНК с РНК-связывающимися молекулами с его использованием. 27.04.2009 Бюл. №12. Патент RU 2353653).
Белки, гликопротеины, протеогликаны, иммобилизованные в ячейках биочипа, могут принадлежать к любой функциональной группе, любому структурному классу, любому организму.
Анализируемыми на биочипе биологическими молекулами, флуоресцирующими в УФ-диапазоне, могут быть, в том числе, любые из перечисленных ниже соединений: белки, пептиды, гликопротеины, протеогликаны.
В качестве белков, пептидов, гликопротеинов, протеогликанов для анализа их взаимодействия с молекулярными зондами биочипа, можно использовать любой белок (либо функциональный домен белка), пептид, гликопротеин, протеогликан, содержащий в своей последовательности один или более аминокислотных остатков триптофана. Если последовательность белка, гликопротеина, протеогликана заранее не известна, то можно использовать белки, гликопротеины, протеогликаны, которые испускают флуоресцентный сигнал в области 300-400 нм. Если белок не содержит флуорофор-содержащих аминокислотных остатков, то в таком случае белок может быть модифицирован аминокислотным тагом по N-, либо С-концу, содержащим один или более аминокислотных остатка триптофана. Функциональный домен белка, не содержащий флуорисцирующих в УФ диапазоне аминокислотных остатков, можно также модифицировать по N-, либо С-концу аминокислотным тагом, содержащим один или более аминокислотных остатка триптофана.
Анализируемый белок, пептид, гликопротеин, протеогликан может проявлять различное сродство к связыванию с иммобилизованными на биочипе зондами, например, нуклеиновым кислотам. Так, к одним нуклеотидым последовательностям эти соединения могут проявлять низкое сродство (либо вовсе не связываться), а к другим - большое сродство, связываясь с последними специфичным образом. Белок, пептид, гликопротеин, протеогликан может неспецифично связываться с нуклеиновыми кислотами, проявляя примерно одинаковое сродство к различным последовательностям нуклеиновых кислот. Исследуемый белок, гликопротеин, протеогликан может принадлежать к любой функциональной группе, любому структурному классу, любому организму.
В качестве подложки биочипа может быть использовано стекло, включая кварцевое, сапфир, металл, керамика, пластик, мембрана. При этом необходимо, чтобы поверхность подложки не флуоресцировала в диапазоне длин волн 300-400 нм. Полезным примером являются биочипы, содержащие слой металла, обычно золота или серебра, и широко используемые для исследования кинетики и термодинамики образования комплексов между биомолекулами методом поверхностного плазмонного резонанса. Katsamba P.S., Park S. and Laird-Offringa I.A. 2002. Kinetic studies of RNA-protein interactions using surface plasmon resonance. Methods. 26:95-104. В другом примере биочипа, биомолекулы - белки иммобилизовали на модифицированной поверхности слоя из пленки алюминия, покрывающей пластиковую подложку. Avseenko N.V., Morozova T.Ya., Ataullakhanov F.I. and Morozov V.N. 2001. Immobilization of proteins in immunochemical microarrays fabricated by electrospray deposition. Anal Chem. 73:6047-52. Еще одним полезным примером являются биочипы, биомолекулы которых иммобилизованы в объеме гелевых ячеек, расположенных на металлизированной поверхности стеклянной подложки. Zubtsova Zh.I., Zubtsov D.A., Savvateeva E.N., Stomakhin A.A., Chechetkin V.R., Zasedatelev A.S. and Rubina A.Y. 2009. Hydrogel-based protein and oligonucleotide microchips on metal-coated surfaces: enhancement of fluorescence and optimization of immunoassay. J Biotechnol. 144:151-9.
Известно большое число способов изготовления 2D-биочипов, среди которых полезными для осуществления настоящего изобретения могут быть методы ступенчатого синтеза in situ олигонуклеотидов заданной последовательности на поверхности платформы биочипа (микроматрицы), такие как метод фотолитографии, Fodor S.P., Read J.L., Pirrung М.С., Stryer L., Lu A.T. and Solas D. 1991. Light-directed, spatially addressable parallel chemical synthesis. Science. 251:767-73. Pease A.C., Solas D., Sullivan E.J., Cronin M.T., Holmes C.P. and Fodor S.P.A. 1994. Light-generated oligonucleotide arrays for rapid DNA sequence analysis. Proc. Natl. Acad. Sci., 91: 5022-5026; Fodor S.P.A., Stryer L., Read L.J. and Pirrung M.C. Methods of making nucleic acid or oligonucleotide arrays, US Patent 6600031, July 29, 2003. Может также быть применен метод с использованием инжекторной (ink-jet) технологии, Hughes T.R, Мао М., Jones A.R, Burchard J., Marton M.J., Shannon K.W., Lefkowitz S.M., Ziman M., Schelter J.M., Meyer M.R, Kobayashi S., Davis C., Dai H., He Y.D., Stephaniants S.B., Cavet G., Walker W.L., West A., Coffey E., Shoemaker D.D., Stoughton R., Blanchard A.P., Friend S.H. and Linsley P.S. 2001. Expression profiling using microarrays fabricated by an ink-jet oligonucleotide synthesizer. Nature Biotechnology, 19: 342-347.
В других способах изготовления 2D- и 3D-биочипов, полезных для осуществления настоящего изобретения, синтезированные олигонуклеотиды или их смеси с гелевыми мономерами наносят на модифицированную платформу биочипа с помощью роботов с использованием пинов или пьезоэлектрических распылительных насадок. Rubina, A.Y., Pan′kov, S.V., Dementieva, E.I., Pen′kov, D.N., Butygin, A.V., Vasiliskov, V.A., Chudinov, A.V., Mikheikin, A.L., Mikhailovich, V.M. an