Способ выявления кандидатных генов для проведения популяционных исследований генетического полиморфизма у детей, проживающих в условиях стронциевой геохимической провинции
Иллюстрации
Показать всеИзобретение относится к области биохимии и медицины, в частности к способу выявления кандидатных генов для проведения популяционных исследований генетического полиморфизма у детей, проживающих в условиях стронциевой геохимической провинции. Для осуществления указанного способа производят отбор пробы крови у детей, проживающих в стронциевой геохимической провинции не менее 3 лет. Из этой пробы выделяют ДНК и создают библиотеку коротких отрезков ДНК. Проводят их гибридизацию с набором заданных праймеров, представляющих в совокупности жидкий ДНК-биочип. Затем гибридизованные участки подвергают секвенированию, устанавливая фактическую последовательность нуклеотидов, составляющих гены, и сравнивают эту последовательность с референтной последовательностью нуклеотидов в генах. Устанавливают отклонения в последовательности, принимая такие отклонения, как ассоциированные с возможными нарушениями здоровья ребенка под действием стронция. В качестве указанных генов используют: CYP1A2, TLR4, TERT, FAS, FOXP3, ТР53, MTHFR, SULT1A1, VEGF, ZMPSTE, SOD, SIRT3, NOS3, PPARD и СРОХ. В последовательности нуклеотидов каждого из указанных генов идентифицируют количество однонуклеотидных полиморфизмов, и в случае наличия таких полиморфизмов в гене в количестве 6 и более прогнозируют связь такого измененного гена с воздействующей на ребенка стронциевой экспозицией в условиях стронциевой геохимической провинции. Этот ген принимают в качестве кандидатного гена для проведения последующих популяционных исследований генетического полиморфизма у детей, проживающих в условиях стронциевой геохимической провинции. Настоящее изобретение позволяет обеспечить возможность выявления через генетический полиморфизм кандидатных генов у детей, ассоциированных с воздействием стронция, и использования в дальнейшем полученной информации для популяционных исследований с целью установлении на ранней стадии определенных заболеваний, обусловленных нарушенным геном. 3 табл., 1 пр.
Реферат
Настоящее изобретение относится к области медицины и представляет собой способ молекулярно-генетической диагностики индивидуального генетического риска развития осложнений в здоровье у детей, проживающих в условиях стронциевой геохимической провинции и подвергающихся токсическому воздействию стронция. Установленные кандидатные гены и их полиморфные варианты у таких детей могут быть использованы при выявлении нарушений экспрессии белка, кодируемого данным геном, а также для прогностических целей при установлении на ранней стадии определенных заболеваний, обусловленных именно таким нарушенным геном.
В последнее десятилетие возникла проблема воздействия стабильного стронция на здоровье в связи с вовлечением в питьевое водоснабжение больших объемов артезианской воды водоносных горизонтов, где содержание стабильного стронция в 5-20 раз превышает предельно-допустимое - 7 мг/л. Стронций, будучи изоформен кальцию и обладая высокой подвижностью, способен блокировать ионные каналы для последнего, воздействовать на кальций-зависимые рецепторы и конкурировать за активные участки белков, не выполняя физиологической функции, что может определить ингибирующее влияние стронция, например, на иммунную реактивность, на функциональную активность остеокластов, на секрецию нейромедиаторов, на состояние эндотелия сосудистой стенки, на процессы детоксикации и т.д. Вот почему раннее выявление в крови специфических последовательностей нуклеиновых кислот, ассоциированных с различными заболеваниями, является важной проблемой в диагностической медицине.
Появление современных лабораторных методов, в частности технологии секвенирования генов, с целью выявления особенностей их индивидуальной молекулярной структуры, диктует необходимость поиска принципиально новых путей предотвращения развития заболевания и его осложнений. Так, прогнозирование течения заболевания на основании генетически детерминированных индивидуальных особенностей становится перспективным путем решения проблемы ранней диагностики и, как следствие, своевременного и адекватного лечения. Определение предикторов развития осложнений заболевания является критерием для дополнительного обследования пациентов с целью ранней диагностики осложнений и назначения терапии в соответствии с установленным диагнозом.
Из уровня техники известен метод молекулярно-генетического анализа определения вариаций (мутаций и однонуклеотидных полиморфизмов) первичной структуры ДНК в генах BRCA (BRest CAncer) BRCA1 и BRCA2, который проводится с помощью конформационно-чувствительного электрофореза (CSGE). Последующее подтверждение наличия вариаций и выявление их характеристик осуществляется путем секвенирования (А.В.Карпухин и др., 2001); (Sanger F., et al., Proc. Natl. Acad. Sci. USA, 1977, V. 74, P. 5463). Суммарное количество выделенных фрагментов дезоксирибонуклеиновой кислоты (ДНК) для амплификации и исследования генов в этом известном методе составили 94. Определение результатов электрофореза осуществлялось нерадиоактивными методами (окрашивание флуоресцентным красителем либо серебрение). Данный анализ не зависит от природы исследуемой ДНК и позволяет выявить однонуклеотидные замены.
Однако этот метод требует большого количества изначальной ДНК, которое не всегда можно получить в клинической практике. Метод сложно тиражировать ввиду его затратности, так как результаты требует постоянного подтверждения проведением дорогостоящего секвенирования.
Также известен способ мини-секвенирования на олигонуклеотидном микрочипе (http://www.jurilab.com/default.asp?toc=66 DrugMEtTM microarray-based pharmacogenetic test processes 16 samples on one slide and includes 27 variations (SNPs) in 8 genes that code for major drug metabolising enzymes (DME). Согласно этому способу при подготовке проб используют предварительную амплификацию исследуемого фрагмента ДНК, содержащего SNP (однонуклеотидный полиморфизм), и последующую гибридизацию его с зондом на биочипе. Последовательность зонда должна быть комплементарна последовательности исследуемой ДНК, причем последнее основание на 3'-конце зонда должно быть комплементарно основанию в ДНК, после которого следует вариабельный нуклеотид. Принцип детекции заключается в следующем: после гибридизации зонда с образцом в реакцию добавляют меченые дидезоксинуклеотиды и ДНК-полимеразу. Возможно присоединение только одного единственного нуклеотида к 3'-концу иммобилизованного зонда, поскольку дидезоксинуклеотиды не содержат на 3'-конце гидроксильной группы, с которой соединяется 5'-фосфатный остаток следующего нуклеотида. После отмывки чипа флуоресценция его ячеек определяется именно этим меченым дидезоксинуклеотидом. Указанный известный метод позволяет с высокой степенью достоверности выявлять генотипы и анализировать более 20 мутаций одновременно. Однако он не обладает мультиплексностью и не позволяет полностью определить все мутации в гене. Кроме того, известный способ не предполагает проведение популяционных исследований.
Также известен Способ экспресс-анализа генетического полиморфизма для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям (Патент РФ №2303634). Анализ генетического полиморфизма проводят путем амплификации полиморфных фрагментов генов посредством двухэтапной полимеразной цепной реакции и гибридизации полученных продуктов на биочипе с последующей регистрацией и интерпретацией результатов гибридизации. Для амплификации полиморфных фрагментов генов используют набор специфичных праймеров. В качестве биочипа на стадии гибридизации используют подложку с набором иммобилизованных на ней специфичных олигонуклеотидов. Применение изобретения обеспечивает выявление функционально-значимых полиморфных локусов. Недостатком этого метода является его значение только для определения фармакогенетического статуса и выявления функционально-значимых полиморфных локусов, без определения конкретных генов предрасположенности к развитию заболеваний.
Известен способ ранней диагностики заболеваний, связанных с нарушением функционирования генетического аппарата клетки (Патент РФ №2249820). Сущность способа заключается в том, что после забора крови ее разделяют на плазму и фракцию клеток крови, затем фракцию клеток крови разделяют на эритроциты и лейкоциты, далее последовательно элюируют внеклеточные нуклеиновые кислоты, связанные с поверхностью эритроцитов и лейкоцитов, из полученных элюатов выделяют фракции внеклеточных нуклеиновых кислот и проводят амплификационный анализ не менее 2-х известных специфических последовательностей нуклеиновых кислот, ассоциированных с определенным заболеванием, с использованием соответствующего метода (полимеразная цепная реакция ПЦР, лигазная цепная реакция, мультиплексная ПЦР и др.). Преимущество изобретения заключается в повышении точности диагностики. Однако его недостатком является то, что в реакциях амплификации, в отличие от секвенирования, выявляется только одна мутация и необходимо проводить несколько реакций для выявления множества полиморфизмов какого-либо гена.
При этом из уровня техники не были выявлены известные способы выявления кандидатных генов для проведения популяционных исследований генетического полиморфизма у детей, ассоциированного с внешнесредовым воздействием стронция, поэтому сделать выбор ближайшего аналога к заявляемому объекту не представляется возможным.
Технический результат, достигаемый предлагаемым изобретением, заключается в обеспечении возможности выявления через генетический полиморфизм кандидатных генов у детей, ассоциированных с воздействием стронция, с использованием в дальнейшем полученной информации для популяционных исследований, с целью применения в прогностических целях при установлении на ранней стадии определенных заболеваний, обусловленных именно таким (нарушенным) геном.
Указанный технический результат обеспечивается предлагаемым способом выявления кандидатных генов для проведения популяционных исследований генетического полиморфизма у детей, проживающих в условиях стронциевой геохимической провинции, согласно которому производят отбор пробы крови у детей, проживающих в стронциевой геохимической провинции не менее 3 лет, из этой пробы крови выделяют дезоксирибонуклеиновую кислоту ДНК, далее создают библиотеку коротких отрезков ДНК и проводят их гибридизацию с набором заданных праймеров, представляющих в совокупности жидкий ДНК-биочип, после чего гибридизованные участки подвергают секвенированию, устанавливая фактическую последовательность нуклеотидов, составляющих гены, и сравнивают эту последовательность с референтной последовательностью нуклеотидов в генах, устанавливая отклонения в последовательности или в замене нуклеотидов, принимая такие отклонения, как ассоциированные с возможными нарушениями здоровья ребенка под действием стронция, при этом в качестве указанных генов используют: CYP1A2 - фермент детоксикации 1 фазы; TLR4 - ген толл-подобного рецептора; TERT - ген теломеразы; FAS - ген рецептора смерти; FOXP3 - транскрипционный фактор клеточной супрессии; ТР53 - транскрипционный фактор онкосупрессии; MTHFR - ген метилентетрагидрофолатредуктазы; SULT1A1 - ген сульфтрансферазы; VEGF - ген васкулярного эндотелиального фактора роста; ZMPSTE - ген цинкметаллопептидазы; SOD - ген супероксиддисмутазы; SIRT3 - ген сиртуина; NOS3 - ген эндотелиальной NO-синтазы, и PPARD - ген рецептора, активируемого пролифераторами пероксисом; СРОХ - ген копропорфириногеноксидазы; далее в каждой последовательности нуклеотидов каждого из указанных генов идентифицируют количество однонуклеотидных полиморфизмов, и в случае наличия таких полиморфизмов в гене в количестве 6 и более прогнозируют связь такого измененного гена с воздействующей на ребенка стронциевой экспозицией в условиях стронциевой геохимической провинции, принимая этот ген в качестве кандидатного гена для проведения последующих популяционных исследований генетического полиморфизма у детей, проживающих в условиях стронциевой геохимической провинции.
Указанный технический результат достигается за счет следующего.
Следует пояснить, что генетические особенности приоритетных регуляторных белков могут служить важным показателем идентификации и прогнозирования состояния реактивности организма и его жизненной программы. Использование инновационных подходов в генотипировании, таких как секвенирование, обеспечивает высокий методический уровень и корректность полученных результатов.
В условиях воздействия факторов окружающей среды имеет значение приспособительная реактивность организма человека для противодействия вреду, связанному с влиянием этих факторов на здоровье. Одним из древних биологических механизмов выживания человеческой популяции является биологическое разнообразие индивидуумов, обусловленное генетическим полиморфизмом.
Для выявления гетерогенности популяции и выявления направления изменения генетического материала под воздействием естественных и антропогенных факторов среды обитания необходим маркер, имеющий помимо качественных характеристик, также количественное выражение.
Одними из таких маркеров являются точечные замены (однонуклеотидные полиморфизмы), так как при увеличении генетического разнообразия изменяется количество и качество таких замен либо в сторону уменьшения, либо в сторону увеличения.
Раннее выявление в крови специфических последовательностей нуклеиновых кислот, ассоциированных с воздействием химических токсикантов, в частности стронция, является важной проблемой в диагностической медицине. Каждый ген является ответственным за белок или фермент, который определяет метаболическую активность ткани и/или органа. При ранней диагностике нарушения в здоровье, когда еще нет симптоматики, кровь является легкодоступным биологическим материалом для получения нуклеиновых кислот, которые могут быть использованы для установления генетического полиморфизма.
При реализации предлагаемого способа отбор пробы крови производится у детей, проживающих в стронциевой геохимической провинции не менее 3 лет, т.к. этот период обеспечивает достаточную экспозиции мутагена для реализации его негативных эффектов на здоровье детей.
Необходимость выделения из пробы крови ДНК обусловлена тем, что ДНК является исходным объектом для последующих исследований. Благодаря тому, что выделенную из пробы крови ДНК подвергают секвенированию, появилась возможность установить в ДНК последовательность нуклеотидов, составляющих гены, ассоциированные с возможными нарушениями здоровья ребенка под действием стронция.
При реализации метода секвенирования в предлагаемом способе рекомендуется использовать жидкие биочипы, так как они обладают целым рядом преимуществ: мультиплексностью (обеспечивают выявление полиморфизма сразу нескольких генов), носят жидкостный характер (не предполагают использования дорогостоящей подложки в форме носителей), дешевизной (так как нет необходимости использовать особые способы их приготовления и обработки), универсальностью (так как один и тот же перечень зондов можно использовать для тестирования разных групп детей).
Выбор в качестве генов в предлагаемом способе следующих генов: CYP1A2 - фермент детоксикации 1 фазы; TLR4 - ген толл-подобного рецептора; TERT - ген теломеразы; FAS - ген рецептора смерти; FOXP3 - транскрипционный фактор клеточной супрессии; ТР53 транскрипционный фактор онкосупрессии; MTHFR - ген метилентетрагидрофолатредуктазы; SULT1A1 - ген сульфтрансферазы; VEGF - ген васкулярного эндотелиального фактора роста; ZMPSTE - ген цинкметаллопептидазы; SOD - ген супероксиддисмутазы; SIRT3 - ген сиртуина; NOS3 - ген эндотелиальной NO-синтазы, и PPARD - ген рецептора, активируемого пролифераторами пероксисом; СРОХ - ген копропорфириногеноксидазы, обусловлен тем, что все они могут быть ассоциированы с возможными нарушениями здоровья ребенка под действием стронция, т.е. того токсиканта, которым преимущественно и подвергаются воздействию дети, проживающие в стронциевой геохимической провинции.
Кроме того, выбранные в качестве кандидатных гены относятся либо к группе генов отвечающих за экспрессию ферментов детоксикации, либо к группе генов, отвечающих за иммунный ответ. То есть, все кандидатные гены ответственны за развитие возможных негативных ответов, связанных с избыточным поступлением стронция в организм.
Последующая операция заявляемого способа состоит в сравнении фактической последовательности нуклеотидов каждого из указанных генов с данными библиотеки последовательности нуклеотидов этих генов у здоровых детей (с референтной последовательностью нуклеотидов в генах). И в каждом гене идентифицируют количество имеющихся однонуклеотидных полиморфизмов.
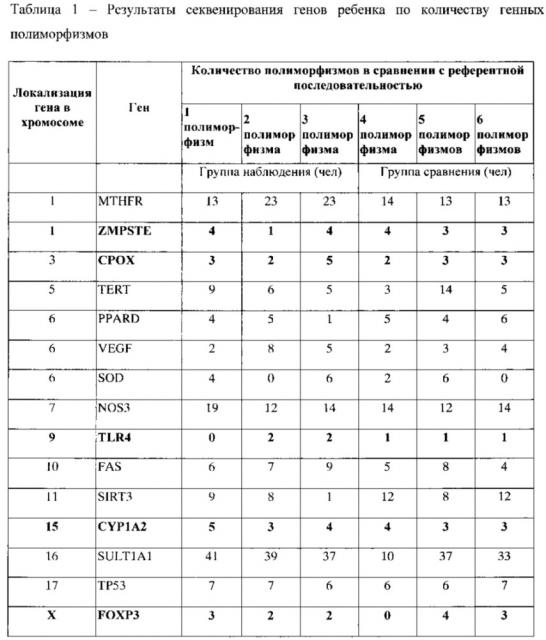
Экспериментальным путем было установлено, что при наличии таких полиморфизмов (замен) в каждом вышеуказанном гене в количестве 6 и более можно прогнозировать связь такого измененного гена с воздействующей на ребенка стронциевой экспозицией в условиях стронциевой геохимической провинции. То есть такие гены и являются кандидатными для последующих популяционных исследований детей. В дальнейшем полученные результаты по указанному критическому генетическому числу могут быть использованы для проведения популяционных исследований выявленных кандидатных генов методом полимеразной цепной реакции в режиме реального времени на значительных контингентах населения для установления популяционных генетических дрейфов.
Апробированный методический подход в предлагаемом способе позволил обосновать критическое число замен в гене (6 и более), что в дальнейшем упрощает подбор кандидатных генов для адекватного популяционного генетического анализа детей, проживающих в стронциевой геохимической провинции.
При наличии 6 замен и более в одном гене методом таргетного секвенирования на жидких чипах подбираются кандидатные гены для дальнейшего популяционного анализа и мониторирования генетических нарушений уже более простым и менее дорогостоящим методом полимеразной цепной реакции в режиме реального времени, т.к. метод секвенирования, хоть и обладает высокой точностью, однако мало пригоден для массового использования в медицинской практике, т.к. предъявляет высокие требования к качеству тестируемых образцов ДНК и требует исключительно дорогостоящего оборудования и реагентов.
Практическое использование результатов предлагаемого способа будет происходить следующим образом. Например, у ребенка, проживающего в условиях воздействия стронция, выявлены методом секвенирования 5 генов из 15 анализируемых, в нуклеотидных последовательностях которых обнаружены 6 и более полиморфных вариаций. Эти гены принимаются в качестве кандидатных, реализация полиморфных изменений которых представляет риск для здоровья контингента, проживающего в аналогичных с этим обследуемым ребенком условиях. Поэтому далее проводится популяционный генетический анализ у детей именно установленных кандидатных генов, но более доступным, чем секвенирование, методом полимеразной цепной реакции (далее - ПЦР) в режиме реального времени, с установлением адресных генетических нарушений у всех подвергающихся опасной экспозиции стронцием.
Полученные данные позволяют рекомендовать к использованию методологию генотипирования нуклеотидных замен, заключающуюся в алгоритмической последовательности проведения персонального таргетного сиквенса на бичипах и на основании его результатов по выявлению кандидатных генов (6 однонуклеотидных замен и более), выполнение популяционного ПЦР-типирования выявленных кандидатных (маркерных) генов.
Перечень операций предлагаемого способа будет проиллюстрирован примером конкретного осуществления.
1. Обследовано детское население (30 детей в возрасте от 3 до 11 лет, группа наблюдения), европеоиды, проживающие на территории стронциевой геохимической провинции не менее 3 лет и постоянно потребляющие питьевую воду несоответствующего качества по содержанию стронция (предельно допустимая концентрация - ПДК, стронция в воде 7 мг/л, на указанной же территории содержание стабильного стронция в воде в более чем в 1,3 раза превышает ПДК).
Группу контроля (группа сравнения) составили 33 ребенка из «условно чистого» района, где содержание стабильного стронция в воде было в пределах ПДК. Группы были сопоставимы по возрасту, полу, соматической заболеваемости и этносу.
2. У указанных детей отбирали пробы цельной крови утром натощак в две специальные пробирки с антикоагулянтом (использовали тризол - для стабилизации РНК, и ЭДТА (Этилендиаминтетрауксусная кислота) (0,05М раствор ЭДТА в соотношении 500 мкл крови на 50 мкл антикоагулянта). Одну пробирку используют для определения содержания стронция в цельной крови. Вторую пробирку - для выделения из нее ДНК.
3. В цельной крови из первой пробирки определяли уровень содержания стронция, например, на атомно-абсорбционном спектрофотометре Perkin Elmer 3110, с использованием в качестве окислителя ацетилентно-воздушной смеси с детектированием в режиме пламенной атомизации. Это определение проводили с информационной целью для подтверждения хронического поступления в организм стронция, и в принципе эта операция не является существенной для реализации предлагаемого способа.
4. Из крови, находящейся во второй пробирке, выделяли ДНК. Использовали общеизвестную сорбентную технологию выделения нуклеиновой кислоты, например, с помощью наборов, описанных на сайте http://www.dna-technology.ru/files/images/DNK&RNK.pdf. При этом известном методе выделения удается максимально сохранить весь объем получаемой ДНК и при этом хорошего качества, без повреждений и примесей. Для выделение ДНК из пробы крови в количестве 100 мкл (лизируют) используют 300 мкл лизирующего раствора, представляющего собой 0,5%-ный раствор саркозила и протеиназы К (20 мг/мл) в ацетатном буфере (pH 7,5). Затем добавляют сорбент (каолин) и последовательными процедурами промывки отмывают фосфатно-солевым буфером (pH 7,2) пробы от белков и смесью изопропиловый спирт : ацетон от липидов. Нуклеиновые кислоты остаются при этом на сорбенте. Далее адсорбированные на сорбенте ДНК из пробы экстрагируют ТЕ-буфером, представляющим собой смесь 10 мМ трис-HCl и 1 мМ ЭДТА (pH 8,0). Экстракт подвергают центрифугированию. После центрифугирования пробирки надосадочная жидкость содержит очищенную ДНК.
5. На основе литературных публикаций сформировали базовую нормальную панель для каждого из следующих генов: CYP1A2; TLR4; TERT; FAS; FOXP3; ТР53; MTHFR; SULT1A1; VEGF; ZMPSTE; SOD; SIRT3; NOS3; PPARD, СРОХ.
6. После выделения ДНК готовили геномную библиотеку, которую впоследствии можно было бы использовать для последующего секвенирования (https://ru.wikipedia.org/wiki. Википедия «Геномная библиотека»). Библиотека должна быть представлена относительно короткими участками ДНК, так как современные технологии секвенировния ограничены длиной прочтения порядка 800 нуклеотидов. Поэтому для этого использовалась процедура небулизации (т.е. разрушения длинной ДНК путем протягивания нити через узкое отверстие под давлением азота. Азот при высоком давлении в 2.1 бар способствует расщеплению молекулы ДНК на отдельные участки различного размера, которые, после последующей очистки с помощью колонок из набора MiniElute ® PCR Purification Kit (Cat. No 28006, Qiagen), можно подобрать по длине, подходящей для секвенирования.
Полученная библиотека носит название Шотган библиотека (ее понятие приведено на сайте: http://www.makhaon.com/index.php «Получение случайной массированной выборки клонированных фрагментов ДНК данного организма (т.е. дробление генома), на основе которых может быть составлена его геномная библиотека; полученные в результате Шотган-эксперимента последовательности нуклеотидов после дополнительного клонирования могут быть использованы в различных генетических экспериментах»), или быстрая библиотека, так как для ее создание требуется всего около двух минут. В такой библиотеке каждая уникальная последовательность содержится в незначительном числе копий, так как изначально полученная ДНК была разрезана в случайном порядке, интересующие нас последовательности нуклеиновых кислот, составляющих гены, могли сохраниться только в нескольких копиях.
Поэтому необходимо обогатить библиотеку, то есть амплифицировать все последовательности, содержащиеся в библиотеке несколько раз (амплификация - это увеличение числа копий ДНК. Лежит в основе полимеразной цепной реакции (ПЦР). Википедия https://ru.wikipedia.org/wiki). Для этого с разрезанными последовательностями ДНК проводилась LM-ПЦР. При этом готовили смесь для LM-ПЦР в пробирке на 1,5 мл из реагентов набора FastStart High Fidelity PCR System, dNTPack (каталожный No. 04738292001): FastStart High Fidelity Buffer w/ 18 mM MgCl2 20 мкл, DMSO 4 мкл, PCR Grade Nucleotide Mix 4 мкл, 40 μМ 454 Rapid-A Oligo 20 мкл, 40 μM 454 Rapid-B Oligo 20 мкл, вода для ПЦР 90 мкл и FastStart High Fidelity Enzyme Blend 2 мкл. Добавляли в каждую пробирку со смесью 20 мкл суспензии стрептавидиновых частиц, связанных с ДНК. Помещали раскапанные пробирки в амплификатор и устанавливали следующую программу амплификации: 95°C 10 минут; 95°C 30 секунд; 64°C 30 секунд; 72°C 3 минуты. Затем отмывали и восстанавливали обогащенную ДНК-библиотеку, подготовливая ее для эмульсионной ПЦР и секвенирования. Эмульсионная ПЦР (эмПЦР) библиотеки ДНК включала 7 основных этапов:
1. Подготовка реагентов и эмульсии:
При подготовка реагентов открывали коробку с реагентами для эмПЦР (emPCR Reagents) и проводили оттаивание компонентов набора при комнатной температуре, за исключением смеси ферментов (Enzyme Mix) и фермента PPiase, которые должны храниться при -15°C - -25°C. После оттаивания перемешивали на вортексе 5 секунд. Перемешивали на вортексе и нагревали пробирку с добавкой (Additive) до 55°C в течение 5 минут для полного растворения. Осаждали все компоненты в мини-центрифуге в течение 10 секунд. Возвращали ферменты на -15°C - -25°C.
При приготовлении Mock Mix и предварительной эмульсии тщательно перемешивали на вортексе пробирку с эмульсионным маслом в течение 10 секунд на максимальной скорости и выливали все содержимое (4 мл) в перемешивающую пробирку Turrax. Готовили 1x Mock Mix, добавив 430 мкл Mock Mix в 1,72 мл воды для молекулярной биологии. Перемешивали на вортексе. Вносили 2,0 мл 1x Mock Mix в перемешивающую пробирку Turrax, содержащую эмульсионное масло. Устанавливали шейкер Ultra Turrax Tube Drive (UTTD) на 4000 rpm в течение 5 минут. Помещали перемешивающую пробирку в UTTD и запускали перемешивание эмульсии в UTTD. По окончании перемешивания доставали пробирку из UTTD.
Готовили смесь Live Amp Mix, как указано в таблице. Вносили реагенты в следующем порядке и количестве: Вода для мол. Биологии - 410 мкл; Additive - 515 мкл; Amp Mix - 270 мкл; Amp Primer - 80 мкл; Enzyme Mix - 70 мкл; PPiase - 2 мкл. Перемешивали на вортексе смесь Live Amp Mix в течение 5 секунд и помещали ее на лед.
2. Связывание библиотеки ДНК.
Готовили 1х промывочный буфер, смешивая 0,5 мл промывочного буфера (Wash Buffer) с 4,5 мл воды для молекулярной биологии. Перемешивали на вортексе связывающие частицы (Capture Beads). Осаждали связывающие частицы (Capture Beads) в настольной мини-центрифуге в течение 10 секунд, переворачивали пробирки на 180°, и снова осажали в течение 10 секунд по схеме: осадить-перевернуть-осадить. Осторожно удаляли супернатант, не задев осадок частиц.
Промывали связывающие частицы (Capture Beads) дважды, внося по 1 мл 1x промывочного буфера (Wash Buffer). Перемешивали на вортексе частицы, по схеме осадить-перевернуть-осадить, удаляли супернатант после каждого этапа промывки. Оттаивали аликвоты библиотеки ДНК. Проводили тепловую денатурацию ДНК, запустив в термоциклере следующую программу, при включенной греющей крышке: 95°C - 2 минуты; 4°C - удержание. Рассчитывали необходимый объем библиотеки ДНК по следующему уравнению:
Например:
Вносили рассчитанный объем библиотеки ДНК в пробирку с промытыми связывающими частицами (Capture Beads). Перемешивали на вортексе в течение 5 секунд.
3. Эмульсификация.
При эмульсификации вносили 1,2 мл смеси Live Amp Mix в пробирку со связанной библиотекой. Перемешивали на вортексе и переносили все содержимое пробирки в пробирку Turrax. Устанавливали UTTD на 2000 rpm в течение 5 минут. Помещали перемешивающую пробирку в UTTD и запускали перемешивание эмульсии в UTTD. По окончании перемешивания доставали пробирку из UTTD.
4. Амплификация
При амплификации сначала проводили раскапывание эмульсии. Для этого с помощью наконечника Combitip, раскапывали по 100 мкл эмульсии в девять стрипов по 8 пробирок с крышками или в 96-луночную плашку (64 лунки). Запечатывали лунки.
Далее проводили реакцию амплификации. Для этого помещали плашку в амплификатор и запускали программу амплификации при включенной греющей крышке амплификатора. Программа длится 6 часов: 1х 4 минуты при 94°C; 50x 30 секунд при 94°C; 4.5 минуты при 58°C; 30 секунд при 68°C; окончание 10°C - удержание.
5. Выделение частиц
Для выделения частиц вначале проводили разрушение эмульсии с помощью вакуума. Переносили набор с маслом и для разрушения эмульсии (GS Junior Titanium emPCR Oil and Breaking Kit) в помещение с внешней вентиляцией. Присоединяли пробирку объемом 50 мл к крышке из набора GS Junior Titanium Oil and Breaking Kit. Вставляли синий переходник в отверстие многоканального дозатора. Подсоединяли другой конец трубки в источник вакуума (с колбой-ловушкой для изопропанола).
Далее выполняли сбор эмульсии и первичную промывку. Для этого включали вакуум и отбирали эмульсии из всех лунок и собирали их в пробирку объемом 50 мл. Промывали лунки дважды 100 мкл изопропанола на лунку с помощью многоканальной пипетки. Отбирали изопропанол в ту же пробирку и переворачивали дозатор вверх, чтобы собрать в пробирку весь материал из наконечников и трубки. Отбирали дозатором в пробирку дополнительные 5 мл изопропанола, чтобы собрать частицы, оставшиеся в трубке. Выключали вакуум, отсоединяли и закрывали пробирку объемом 50 мл, содержащую частицы с клонально амплифицированной ДНК. Переносили пробирку объемом 50 мл из вытяжного шкафа.
Производили отмывку и выделение частиц. Для этого перемешивали на вортексе пробирку с собранной эмульсией. Добавляли изопропанол до конечного объема 35 мл и перемешивали на вортексе, чтобы перемешать осадок. Осаждали частицы в центрифуге при 930 g в течение 5 мин (2813 rpm для центрифуги 5430, ротор F-35-6-30) и осторожно сливали супернатант. Добавляли 10 мл усиливающего буфера (Enhancing Buffer) и тщательно перемешивали на вортексе. Добавляли изопропанол до 40 мл. Осаждали частицы в центрифуге при 930 g в течение 5 мин и осторожно удаляли супернатант. Добавляли изопропанол до конечного объема 35 мл и тщательно перемешивали на вортексе. Осаждали частицы в центрифуге при 930 g в течение 5 мин и осторожно удаляли супернатант. Добавляли этанол до 35 мл и тщательно перемешивали на вортексе. Осаждали частицы в центрифуге при 930 g в течение 5 мин и осторожно удаляли супернатант. Добавляли Enhancing Buffer до конечного объема 35 мл и тщательно перемешивали на вортексе. Осаждали частицы в центрифуге при 930 g в течение 5 мин и осторожно удаляли супернатант, оставив примерно 2 мл Enhancing Buffer. С помощью пипетки объемом 1000 мкл переносили суспензию частиц, несущих ДНК в прилагаемую к набору микроцентрифужную пробирку объемом 1,7 мл. Осаждали-переворачивали-осаждали и удаляли супернатант. Промывали содержимое пробирки объемом 50 мл 1 мл Enhancing Buffer и переносили содержимое пробирки в пробирку объемом 1,7 мл. Осаждали-переворачивали-осаждали и удаляли супернатант. Промывали осадок дважды 1 мл Enhancing Buffer. Осаждали-переворачивали-осаждали и удаляли супернатант.
6. Насыщение частиц с библиотекой ДНК
Вначале проводили насыщение. Для этого включали термоблок и устанавливали его на 65°С. Подготавливали раствор для плавления ДНК (Melt Solution), смешав 125 мкл NaOH (10 N) с 9,875 мл воды для молекулярной биологии. Добавляли 1 мл Melt Solution в пробирку объемом 1,7 мл, содержащую частицы, и перемешивали на вортексе. Инкубировали 2 минуты при комнатной температуре. Осаждали-переворачивали-осаждали и удаляли супернатант. Повторяли это еще один раз. Вносили 1 мл буфера для отжига (Annealing Buffer) в пробирку объемом 1,7 мл, содержащую частицы, и перемешивали на вортексе. Осаждали-переворачивали-осаждали и удаляли супернатант. Повторяли это еще два раза. Вносили 45 мкл буфера для отжига (Annealing Buffer), 25 мкл праймера для обогащения (Enrich primer) в пробирку объемом 1,7 мл, содержащую частицы и перемешивали на вортексе. Помещали пробирку в термоблок на 65°С на 5 минут и затем сразу охлаждали на льду в течение 2 минут. Добавляли 1 мл Enhancing Buffer в пробирку объемом 1,7 мл, содержащую частицы, и перемешивали на вортексе. Осаждали-переворачивали-осаждали и удаляли супернатант. Повторяли это еще два раза. Добавляли 1 мл Enhancing Buffer в пробирку объемом 1,7 мл, содержащую частицы и перемешивали на вортексе.
Затем готовили насыщающие частицы (Enrichment Beads). Для этого перемешивали на вортексе пробирку с коричневыми насыщающими частицами (Enrichment Beads) в течение 1 минуты. Помещали пробирку в концентратор магнитных частиц (КМЧ) и ждали 3 минуты, пока частицы не осядут. Удаляли супернатант, не задев насыщающие частицы. Вносили 500 мкл Enhancing Buffer и перемешивали на вортексе. Осаждали Enrichment Beads в КМЧ. Удаляли супернатант, не задев Enrichment Beads. После удаления супернатанта доставали пробирку из КМЧ. Вносили 80 мкл Enhancing Buffer и перемешивали на вортексе.
Затем производили насыщение несущих ДНК частиц. Для этого вносили 80 мкл промытых Enrichment Beads в пробирку объемом 1,7 мл, содержащую частицы, и полностью перемешивали на вортексе. Перемешивали пробирку в ротаторе пробирок при комнатной температуре в течение 5 минут. Помещали пробирку в КМЧ и ждали 3-5 минут до полного осаждения Enrichment Beads. Переворачивали КМЧ несколько раз и дожидались осаждения частиц. Осторожно удаляли супернатант с помощью наконечника пипетки объемом 1000 мкл, не задевая коричневые Enrichment Beads. Промывали частицы буфером Enhancing Buffer до полного исчезновения видимых белых частиц в супернатанте, следующим образом: вносили в пробирку 1 мл Enhancing Buffer; доставали пробирку из КМЧ и тщательно перемешивали на вортексе; помещали пробирку назад в КМЧ, чтобы осадить частицы на стенке пробирки с помощью магнита. Переворачивали КМЧ и дожидались осаждения частиц; осторожно удаляли супернатант с помощью наконечника объемом 1000 мкл, не задевая Enrichment Beads; повторяли 6-10 раз до полного исчезновения белых частиц в супернатанте.
Далее производили сбор насыщенных частиц ДНК. Для этого доставали пробирку с Enrichment Beads (пробирку насыщения) из КМЧ и растворяли осадок частиц в 700 мкл раствора Melt Solution. Перемешивали на вортексе 5 секунд и помещали пробирку насыщения в КМЧ до полного осаждения насыщенных частиц. Переносили супернатант, содержащий насыщенные частицы ДНК, в чистую микроцентрифужную пробирку объемом 1,7 мл. Снова добавляли 700 мкл раствора Melt Solution в пробирку насыщения. Перемешивали на вортексе 5 секунд, и помещали пробирку насыщения в КМЧ до полного осаждения насыщенных частиц. Переносили супернатант, содержащий насыщенные частицы ДНК, в ту же микроцентрифужную пробирку объемом 1,7 мл, полученную на этапе 3. Осаждали-переворачивали-осаждали пробирку на 1,7 мл и удаляли супернатант. Вносили 1 мл буфера Annealing Buffer и перемешивали на вортексе 5 секунд. Осаждали-переворачивали-осаждали и удаляли супернатант. Повторяли эти этапы дважды. Вносили 100 мкл буфера Annealing Buffer и перемешивали на вортексе.
7. Отжиг праймера для секвенирования Seq Primer.
Добавляли 25 мкл праймера Seq и перемешивали на вортексе. Помещали пробирку объемом 1,7 мл в термоблок на 65°С на 5 минут и затем сразу охлаждали на льду в течение 2 минут. Добавляли 1 мл буфера Annealing Buffer и перемешивали на вортексе в течение 5 секунд. Осаждали-переворачивали-осаждали и удаляли супернатант. Повторяли этот этап дважды. Добавляли 1 мл буфера для Annealing Buffer к осажденным частицам и перемешивали на вортексе. Осаждали-переворачивали-осаждали, чтобы осадить частицы на дне пробирки. Для постановки секвенирования в GS Junior (секвенатор) необходимо внести 500000 насыщенных частиц.
Каждое новое присоединение нуклеотида к достраивающейся цепочке нуклеотидов в ДНК и в последующем обрабатывалось с помощью программного обеспечения и преобразовывалось в привычного для нас вида последовательность нуклеотидов.
Метод секвенирования значительно увеличивает скорость генотипирования по множественным маркерам и позволяет выявлять новые мутации. Если ген вариабелен, то можно в одном исследовании изучить все варианты мутаций, имеющиеся у пациента, а их количество позволяет проводить статистический анализ генетической вариабельности в отличие от возможностей ПЦР.
8. Далее в каждой полученной при секвенировании последовательности нуклеотидов каждого из указанных генов идентифицировали количество однонуклеотидных полиморфизмов, т.е. участков, отличающихся от референтной последовательности, взятой из базы данных NCBI (www.ncbi.nlm.nih.gov).
При этом изучение встречаемости генного полиморфизма аналогичных генов проведено на популяционной выборке 30 детей (группа наблюдения) и 33 ребенка (контроль - группа сравнения) методом ПЦР в реальном времени. Обработка данных по генотипированию проводилась с использованием унифицированной программы «Ген Эксперт». Данная программа служит для расчета статистических параметров исследований “случай-контроль”, использующих SNP (однонуклеотидные полиморфизмы). Для выбора критериев оценки значимости межгрупповых различий использовали непараметрический U-критерий Манна-Уитни. Зависимости между признаками оценивали методом корреляционно-регрессионного анализа. Достоверными считались различия между группами при p<0,05.
Результаты исследования.
Оценка вариабельности указанных генов с использованием результатов сиквенса (т.е. результатов секвенирования) выявила различия в полиморфизмах по более чем двумстам точкам у каждого отдельного индивидуума. При этом выявлена достоверная зависимость количества мутаций от концентрации стронция в крови (R2=0,59; p=0,00016), которое возрастало в среднем с 210 нуклеотидных замен в группе сравнения до 226 в группе наблюдения. Этим было доказано, что именно стронций влияет на возникновение генетического полиморфизма в рассматриваемых генах.
По результатам секвенирования, проведенного на ограниченном контингенте (10 человек из группы н