Способ выявления структурных перестроек генома опухолевых клеток (химерных генов), определяющих выбор терапии и прогноз при острых лейкозах у детей, с использованием от-пцр и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом)
Иллюстрации
Показать всеИзобретение относится к области биохимии и генетики. Заявлен способ выявления структурных перестроек генома опухолевых клеток (химерных генов), определяющих выбор терапии и прогноз при острых лейкозах у детей, с помощью ОТ-ПЦР с последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом). Изобретение позволяет выявлять 22 известных химерных гена. Выявление этих транслокаций в опухолевых клетках костного мозга пациентов является стандартом современной диагностики лейкозов и используется в большинстве современных протоколов для разделения пациентов на группы риска и выбора терапии, включая лекарственную терапию высокими дозами цитостатических препаратов и/или трансплантацию костного мозга. 2 з.п. ф-лы, 2 ил., 3 табл., 4 пр.
Реферат
Область техники, к которой относится изобретение
Изобретение относится к области генетики, молекулярной биологии и медицины и касается выявления структурных перестроек генома опухолевых клеток (химерных генов), определяющих выбор терапии и прогноз при острых лейкозах у детей, с использованием обратной транскрипции - полимеразной цепной реакции (ОТ-ПЦР) и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом).
Уровень техники
Известен ряд способов определения структурных перестроек генома опухолевых клеток, в которых используются различные подходы.
Цитогенетический анализ определяет различные структурные изменения генома, включая изменения числа хромосом, однако обладает низким разрешением, не способен выявлять скрытые (криптические) перестройки, не дает возможности установить молекулярную природу перестроенного участка. Метод заключается в окраске в очень кислой среде краской Гимзы (Giemsastain), в результате чего хромосомы приобретают специфическое неравномерное окрашивание в виде полос, которые принято называть G-бэндами. Каждая пара хромосом имеет свое специфическое окрашивание, которое изменяется в случае хромосомных аберраций (делеции, транслокации, инверсии). Несмотря на ограничения в аналитической чувствительности и доступности делящихся клеток в клиническом образце, этот метод продолжает предоставлять ценную информацию о перемещениях, а также числовые изменения в числе хромосомы и структуре [Fluorescence immunophenotypic and interphase cytogenetic characterization of nodal lymphoplasmacytic lymphoma./ Sargent RL, Cook JR, Aguilera NI, Surti U, Abbondanzo SL, Gollin SM, Swerdlow SH.// Am J Surg Pathol. - 2008 - Vol. 32. - P. 1643-53.; Chromosomal aberrations in childhood acute lymphoblastic leukemia: 15-year single center experience/ Jarosova M, Volejnikova J, Porizkova I, Holzerova M, Pospisilova D, Novak Z, Vrbkova J, Mihal V.//Cancer Genet. - 2016 – Vol. 209 - Р. 340-7].
С помощью FISH (флуоресцентной гибридизации insitu) возможно определение нормальной или аномальной локализации гена, числа его копий, а также структурных перестроек хромосомного аппарата опухолевой клетки. Зонд, меченный биотином или непосредственно флуоресцентным красителем, наносят на образец интерфазных клеток или метафазных пластинок, иммобилизованных на подложке или в твердом субстрате. Ядерную ДНК иммобилизованных клеток и меченный зонд предварительно денатурируют, далее происходит ренатурация последовательности ДНК ядра и зонда и образование гибридных дуплексов, в том случае если в ДНК ядра или хромосом имеется комплементарная зонду последовательность. Не полностью связанный и некомплементарный меченый зонд удаляют интенсивным промыванием препарата. Метод FISH является одним из базовых методов в молекулярной диагностике лейкозов. С помощью этого метода можно обнаружить криптические транслокации, что является его преимуществом по сравнению с цитогенетическим методом. Более высокая разрешающая способность позволяет выявлять изменения на субмикроскопическом уровне, например, определять делеции или амплификации участков хромосом, содержащих несколько генов, или даже один ген [Cytogenetic abnormalities detected by fluorescence in situ hybridization on paraffin-embedded chronic lymphocytic leukemia/small lymphocytic lymphoma lymphoid tissue biopsy specimens./ Flanagan MB, Sathanoori M, Surti U, Soma L, Swerdlow SH.// Am J Clin Pathol. - 2008. - Vol. 130. - P. 620-7.; Soszynska K, Mucha B, Debski R, Skonieczka K, Duszenko E, Koltan A, Wysocki M, Haus O. The application of conventional cytogenetics, FISH, and RT-PCR to detect genetic changes in 70 children with ALL.Ann Hematol. 2008 Dec; 87(12):991-1002]. К недостаткам метода относится невозможность точного определения места слияния генов, необходимость в дорогостоящем оборудовании и расходных материалах, высокие требования к специалистам-исследователям.
Использование нескольких флуоресцентных меток одновременно позволяет проводить многоцветную гибридизацию и определять широкий спектр хромосомных перестроек в одной клетке. Этот подход получил название спектрального кариотипирования (SKY, spectralcaryotyping) и удобен при анализе пациентов, имеющих сложный кариотип с множественными аберрациями [Betts DR, Stanchescu R, Niggli FK, Cohen N, Rechavi G, Amariglio N, Trakhtenbrot L. SKY reveals a high frequency of unbalanced translocations involving chromosome 6 in t(12;21)-positive acute lymphoblastic leukemia. Leuk Res. 2008. 32(1):39-43].
Полимеразная цепная реакция (ПЦР) широко используется для выявления химерных генов при диагностике острых лейкозов. Сущность метода заключается в многократном избирательном увеличении определенного участка нуклеиновой кислоты. Существует большое количество методов обнаружения химерных генов, основанных на ПЦР. Анализ последовательностей химерных генов можно проводить как на уровне ДНК, так и на уровне РНК.
Для определения уникальных для пациента мест разрывов на уровне геномной ДНК используется длинная инвертированная ПЦР (LDI-PCR). Метод применяется в том случае, когда известен один из генов, вовлеченных в перестройку. Геномную ДНК фрагментируют с помощью рестриктаз, полученные фрагменты сшивают в кольцо и проводят серию ПЦР с различными праймерами, специфичными для разных участков известного гена. Секвенирование полученных ПЦР-продуктов позволяет идентифицировать второй ген-партнер [Meyer С, Schneider В, Reichel М, Angermueller S, Strehl S, Schnittger S, Schoch C, Jansen MW, van Dongen JJ, Pieters R, Haas OA, Dingermann T, Klingebiel T, Marschalek R. Diagnostic tool for the identification of MLL rearrangements including unknown partner genes. ProcNatlAcadSciUSA2005; 102(2):449-54]. С одной стороны, этот метод открывает возможности для очень специфичного анализа характерной для данного пациента перестройки. С другой стороны, такой анализ является трудоемким, длительным, требует высококвалифицированного персонала и не подходит для рутинной диагностики.
Для определения химерных генов широко применяют обратную транскрипцию-полимеразную цепную реакцию (ОТ-ПЦР). Суть метода заключается в выявлении химерной РНК в опухолевых клетках на основе ее перевода с кДНК с помощью реакции обратной транскрипции. Химерная кДНК далее может быть амплифицирована в стандартной ПЦР реакции со специфическими к данной перестройке праймерами [Detection of minimal residual disease in acute myelomonocytic leukemia with abnormal marrow eosinophils by nested polymerase chain reaction with allele specific amplification./ J, Cayuela JM, Daniel MT, Berger R, Sigaux F.// Blood. - 1994. - Vol. 84. - P. 2291-2296]. В американском патенте US 20120171670 (дата публикации 2012-07-05) применяют гнездовую ПЦР амплификации области 195INS BCR-ABL1 и 243INS BCRABL1. Полученный фрагмент экстрагируют из геля, очищают и секвенируют.
В диагностике хромосомных перестроек используют ОТ-ПЦР в реальном времени. Суть метода состоит в регистрации классической S-образной кривой накопления амплифицируемого продукта, в случае наличия соответствующей матрицы, содержащей транслокацию. Отсутствие S-образной кривой свидетельствует об отсутствии соответствующей химерной матрицы [Novel real-time polymerase chain reaction assay for simultaneous detection of recurrent fusion genes in acute myeloid leukemia./ Dolz S, E, Fuster Llop M, Cervera J, Such E, De Juan I, Palanca S, Murria R, Bolufer P, Luna I, Gomez I, M, M, Sanz MA.// J Mol Diagn. - 2013. - Vol. 5. - P. 678-686.9]. Преимуществом метода является быстрота получения результата, высокая чувствительность, недостатком - малопараметричность. Иными словами, для получения информации, необходимо поставить отдельные тесты с праймерами к каждой транслокации и большинству мест перестройки. Метод в большей степени подходит для подтверждения и определения более точной локализации уже обнаруженной другими методами транслокации, а также для отслеживания рецидивов (MRD, minimal residual disease, минимальная остаточная болезнь).
Различные варианты постановки ПЦР в реальном времени описаны в ряде патентов. Китайский патент CN 102251031 (дата публикации 2013-07-24) основан на применении TaqMan MGB probe для определения ряда химерных генов: AML1-ETO, BCR-ABL190, BCR-ABL210, CBFb-MYH11(A), CBFb-MYH11(D), CBFb-MYH11(E), Е2А-РВХ1, MLL-AF4, PML-RARa (bcr1), PML-RARa(bcr2), PML-RARa(bcr3), SIL-TAL1 and TEL-AML1. В китайском патенте CN 102827937 (дата публикации 2014-03-19) описан метод для определения транслокаций в генах BCR-ABL, AML1-ETO и PML-RARa, а в CN 102925558 (дата публикации 2014-09-17), французском ЕР2204457 (дата публикации 2010-07-07) и корейском KR 1020060000840 (дата публикации 2014-09-17) только гена PML-RARA. Такое большое количество тест-систем, основанных на методе ПЦР в реальном времени, объясняется тем, что данный метод достаточно прост в освоении, часто не требует дополнительных навыков, но для его осуществления требуется приобретение дорогостоящего оборудования.
В исследованиях природы рака для анализа свойств опухолевых клеток и классификации опухолей широкое применение нашли микрочипы [A diagnostic biochip for the comprehensive analysis of MLL translocations in acute leukemia./ Maroc N, Morel A, Beillard E, De La Chapelle AL, Fund X, Mozziconacci MJ, Dupont M, Cayuela JM, Gabert J, Koki A, Fert V, Hermitte F.//Leukemia.l - 2004. - Vol. 18. - P. 1522-1530; Development of a biochip-based assay integrated in a global strategy for identification of fusion transcripts in acute myeloid leukemia: a work flow for acute myeloid leukemia diagnosis./ Giusiano S1, Formisano-Treziny C, Benziane A, Maroc N, Picard C, Hermitte F, Taranger-Charpin C, Gabert J.// International Journal of Laboratory Hematology. - 2010. - Vol. 32. - P. 398-409.; A pipeline with multiplex reverse transcription polymerase chain reaction and microarray for screening of chromosomal translocations in leukemia./ Xiong FF, Li BS, Zhang CX, Xiong H, Shen SH, Zhang QH.// BioMed Research International. - 2013. - Vol. 2013].
Опубликовано несколько патентов и патентных заявок, в которых приведены различные технологии гибридизации с биочипами. Например, в американском патенте US 20030198977 (дата публикации 2003-10-23) разработан метод определения хромосомных транслокаций с образованием химерных генов E2A-Pbx1, MLL-AF4, AML1-ЕТО, BCR-ABL (P190), BCR-ABL1 (Р210), TEL-AML1, PML-RARA, CBFB-MYH11, SIL-TAL1 в единой реакционной смеси с использованием микрочипов. В китайском патенте CN 101955991 (дата публикации 2014-05-28) и патентной заявке WO 2011120398 (дата публикации 2011-10-06) ПЦР-продукты химерных генов, связанных с лейкемией, гибридизуют со смесью зондов для обнаружения флуоресцентных сигналов.
Гелевые биочипы, которые давно и успешно разрабатываются в Лаборатории биологических микрочипов Федерального государственного бюджетного учреждения науки Института молекулярной биологии им. В.А. Энгельгардта Российской академии наук, являются примером диагностических микрочипов низкой плотности. Метод гибридизации с аллель-специфическими олигонуклеотидами включает амплификацию фрагментов ДНК и последующую гибридизацию флуоресцентно меченой ДНК мишени с аллель-специфичными олигонуклеотидами [Т.V. Nasedkina, V.S. Zharinov, Е.A. Isaeva et al., "Clinical Screening of Gene Rearrangements in Childhood Leukemia by Using a Multiplex Polymerase Chain Reaction-Microarray Approach," Clinical Cancer Research, vol. 9, no. 15, pp. 5620-5629, 2003.].
Используя метод ultra high-throughput sequencingChromPET в американском патенте US20120178635 (дата публикации 2012-07-12), выявляли хромосомные аберрации BCR-ABL1. Анализ обладает хорошей чувствительностью и специфичностью, но для его осуществления требуется приобретение дорогостоящего оборудования, расходных материалов и реактивов. В патентной заявке WO 1994026930 (дата публикации 1994-11-24) используют Northern и Southern блот для идентификации AF-9, AF-4, AF-6, AF-17. Данная технология не дешевая, трудоемкая и требует высокой квалификации персонала.
Раскрытие изобретения
Задача настоящего изобретения состоит в создании способа выявления структурных перестроек генома опухолевых клеток. Сущность изобретения заключается в обеспечении способа выявления хромосомных транслокаций в образцах костного мозга или периферической крови, которые приводят к образованию химерных генов и определяют выбор терапии и прогноз при острых лейкозах. Данный способ позволяет выявлять 22 хромосомные транслокации, которые являются наиболее информативными маркерами, определяющими различные подтипы заболевания, выбор терапии и прогноз у детей с острыми лейкозами.: ETV6/RUNX1; BCR/ABL1p210; MLL/AFF1; BCR/ABL1p190; PML/RARA; RUNX1/RUNX1T1; TCF3/PBX1; MLL/MLLT3; CBFB/MYH11; MLL/MLLT10; MLL/MLLT1; SIL/TAL1; MLL/MLLT4; NPM1/ALK; TCF3/HLF; MLL/ELL; MLL/EPS15; PICALM/MLLT10; MLL/MLLT11; DEK/NUP214; RBM15/MKL1; FUS/ERG.
Основными признаками данного изобретения являются ОТ-ПЦР с последующей ПЦР и гибридизацией на биочипе, содержащем набор иммобилизованных олигонуклеотидных зондов.
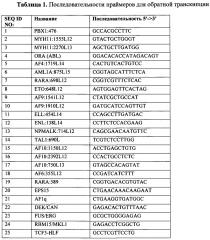
Первый важный признак изобретения - смесь специфических праймеров для обратной транскрипции, которая используется для получения кДНК на матрице РНК, выделенной из костного мозга или периферической крови пациента. Последовательность праймеров для обратной транскрипции приведена в табл. 1 и Перечне последовательностей (SEQ ID NO: 1-25).
Второй важный признак изобретения - праймеры для специфичной амплификации последовательностей химерных генов, с помощью которых флуоресцентно меченый ПЦР-продукт нарабатывается только при наличии химерного транскрипта в образце. ПЦР осуществляется в 4 отдельных мультиплексных реакциях. Последовательности праймеров для проведения ПЦР приведены в табл. 2 и Перечне последовательностей (SEQ ID NO: 26-75).
Третий важный признак изобретения - биочип, содержащий набор иммобилизованных олигонуклеотидных зондов, высоко специфичных и комплементарных участкам химерных генов, в том числе и в местах слияния генов. Последовательности зондов приведены в табл. 3 и Перечне последовательностей (SEQ ID NO: 76-138). Олигонуклеотидные зонды иммобилизуются в ячейках гидрогелевого микрочипа, как описано в патенте RU 2206575 (дата публикации 2003-06-20) в концентрации 4000 мкМ. Схема расположения зондов в ячейках биочипа для выявления структурных перестроек генома опухолевых клеток (химерных генов) приведена на Фиг. 1.
Смеси специфических праймеров для обратной транскрипции, комплементарных 3'-участкам исследуемых химерных генов используются для получения кДНК на матрице РНК, выделенной из образцов костного мозга или периферической крови. Набор праймеров для ПЦР используется для амплификации интересующих фрагментов с одновременным флуоресцентным мечением продукта. Далее проводится гибридизация флуоресцентно меченных фрагментов ДНК, полученных в ходе ПЦР, с иммобилизованными в ячейках геля олигонуклеотидными зондами, расположенными на пластиковой подложке. После проведения гибридизации и отмывки биочипа проводится анализ полученной флуоресцентной картины, на основании которого делается вывод о наличии химерного гена в исследуемом образце. Примеры картин гибридизации приведены на Фиг. 2.
Краткое описание чертежей
Фиг. 1. Схема биочипа для выявления структурных перестроек генома опухолевых клеток (химерных генов): (а) схема расположения ячеек на биочипе; (б) расшифровка условных обозначений. В угловых позициях нанесены ячейки (М), содержащие флуоресцентный краситель Су5, светящийся перманентно, для ориентировки и контроля интенсивности свечения.
Фиг. 2. Гибридизационные картины, полученные с помощью биочипа для выявления структурных перестроек генома опухолевых клеток (химерных генов). А - образец с транслокацией t(8;21) RUNX1/RUNX1T1; Б - контрольный образец, не несущий транслокацию.
Осуществление изобретения
Для обеспечения оптимальных условий обратной транскрипции и ПЦР необходимо использовать специфические праймеры, отжиг которых на мишени происходит при одинаковой температуре (для каждого этапа), и они не образуют при этой температуре вторичных структур. Для оптимальной наработки необходимого для гибридизации на биочипе количества флюоресцентно-меченого ПЦР-продукта должны быть подобраны такие параметры как концентрации реагентов, состав мультиплексных реакций, временной и температурный режим обратной транскрипции и ПЦР. В результате проведенной работы подобраны праймеры для ОТ, условия ПЦР, которые позволяют осуществлять эффективную специфическую наработку фрагмента, соответствующего последовательностям химерных генов.
Олигонуклеотиды для иммобилизации на биочипе подбираются таким образом, чтобы идентифицировать все выбранные для анализа химерные гены.
При дизайне ДНК-зондов руководствовались следующими принципами:
1) последовательность зонда выбирали комплементарной «плюс» или «минус» - цепи ДНК, нарабатывающейся в ходе асимметричного этапа ПЦР, в зависимости от того, с какого праймера (прямого или обратного) было целесообразно проводить ассиметричную ПЦР;
2) при выборе структур избегали стабильных вторичных структур - шпилек и димеров, т.к. их присутствие снижает эффективность гибридизации;
3) выбранная последовательность не должна быть комплементарной другим областям ПЦР-продукта, кроме целевой, либо другим ПЦР-продуктам, нарабатывающимся в мультиплексной ПЦР.
На биочипе для выявления структурных перестроек генома опухолевых клеток (химерных генов) иммобилизовано 63 высокоспецифичных олигонуклеотидных зондов (табл. 2 и Перечень последовательностей (SEQ ID NO: 76-138)), структура которых обеспечивает специфичное связывание с полностью комплементарными ДНК-мишенями. Зонды подобраны таким образом, что флюоресцентный сигнал наблюдается только в ячейках, с которых образовался совершенный дуплекс между олигонуклеотидным зондом и флюоресцентно-меченым ПЦР-продуктом.
Приведем последовательность анализа с использованием данного метода. Обратная транскрипция на матрице РНК для получения кДНК проводится со смесью специфических праймеров с использованием обратной транскриптазы вируса лейкемии мышей Молони (Moloney Murine Leukemia Virus) - MMLV различных производителей. Наработка флюоресцентно меченных ПЦР-продуктов для последующей гибридизации на биочипе проводится посредством ПЦР, при этом в качестве матрицы для проведения реакции используется кДНК, полученная на этапе обратной транскрипции.
ПЦР может быть проведена с использованием любого вида термостабильной полимеразы (Taq-полимераза, Tth-полимераза, Tf1-полимераза, Pfu-полимераза, Vent-полимераза, DeepVent-полимераза и другие коммерчески доступные ферменты, выделенные из термофилов), работающей в соответствующем буфере. Для построения новой цепи в буфер добавляется смесь дНТФ (дАТФ, дГТФ, дЦТФ, дТТФ), при этом вместо дТТФ может быть использован дУТФ. Для проведения ПЦР могут быть использованы готовые коммерчески доступные наборы, содержащие все необходимые компоненты за исключением праймеров, например QIAGEN Multiplex PCR Kit.
На этапе ПЦР проходит амплификация ДНК-локусов, соответствующих только химерной кДНК. ПЦР проходит в 2 стадии: на первой амплификация целевых продуктов осуществляется с использованием комбинированных праймеров, состоящих из участка, комплементарного последовательности химерного гена, и короткой универсальной вставки на 5'-конце, на второй стадии в работу вступают универсальные праймеры, последовательности которых соответствует последовательностям универсальных вставок. ПЦР осуществляется в 4 мультиплексных реакциях. В мультиплексе №1 содержатся праймеры для выявления транслокаций MLL/AFF1; MLL/MLLT4; MLL/ELL; MLL/MLLT1; CBFB/MYH11 (SEQ ID NO: 26-35), в мультиплексе №2 - праймеры для контрольного гена ABL1 и праймеры для транслокаций MLL/MLLT10; PICALM/MLLT10; SIL/TAL1; DEK/NUP214; TCF3/HLF (SEQ ID NO: 36-51), в мультиплексе №3 - праймеры для транслокаций ETV6/RUNX1; BCR/ABL1p210; BCR/ABL1p190; PML/RARA; RUNX1/RUNX1T1; TCF3/PBX1 (SEQ ID NO: 52-63), в мультиплексе №4 - праймеры для транслокаций MLL/MLLT3; NPM1/ALK; MLL/EPS15; MLL/MLLT11; RBM15/MKL1; FUS/ERG (SEQ ID NO: 26-28, 64-73). Помимо локус-специфичных праймеров все мультиплексы содержат универсальные праймеры Т3 и Т7 (SEQ ID NO: 74-75) в ассиметричном соотношении (например, 1:50) для преимущественной наработки одноцепочечного продукта. В качестве флуоресцентной метки используется Су5-дУТФ, встраивающийся de novo в синтезируемую ДНК-цепь. В качестве флуоресцентной метки также может быть использован любой флуорохром без ограничения (например, FITC, Texas red, Су-3 и т.д.), а также биотин.
Для синтеза праймеров и олигонуклеотидных зондов используют такие химические подходы как фосфодиэфирный метод, гидрофосфорильный метод и т.д., при этом наиболее распространенным в настоящее время является фосфоамидитный синтез. Синтез праймеров осуществляется на автоматических ДНК/РНК синтезаторах, например производства фирмы «Applied Biosystems» (США). Олигонуклеотидные зонды модифицируются путем добавления к 5'- или 3'-концу активной группы, обеспечивающей иммобилизацию, в астоматическом или постсинтетически в ручном режиме. Например, при синтезе олигонуклеотидных зондов с помощью 3'-Amino-Modifier С7 CPG 500 («Glen Research)), США) на 3'-конец олигонуклеотидов вводится спейсер со свободной аминогруппой, используемый для последующей иммобилизации олигонуклеотида на биочипе.
Далее проводится гибридизация флуоресцентно меченных фрагментов ДНК, полученных в ходе ПЦР, с иммобилизованными в ячейках геля олигонуклеотидами, которые представляют собой участки, соответствующие последовательностям участков химерных генов.
Гибридизация ПЦР-продукта с олигонуклеотидными зондами на биочипе может быть проведена в любом гибридизационном буфере, например в гуанидиновом или SSPE-буфере. Перед постановкой гибридизации ПЦР-продукт прогревают при 95°С в течение 5 мин., затем охлаждают на льду в течение 2 минут, после чего наносят гибридизационную смесь на биочип. Гибридизация проводится 8-14 ч. при 37°С. Отмывка биочипа после проведения гибридизации может быть проведена в любом известном в данной области техники буфере с добавлением соли (SSC, SSPE и т.п.) или более короткое время в дистиллированной воде [Molecular cloning: a laboratory manual / J.F. Sambrook, D.W. Russell // Cold Spring Harbor Laboratory Press. - 2001].
ПЦР-продукт, нанесенный в гибридизационную камеру биочипа, в условиях (состав реакции, время и температура), при которых осуществляется гибридизация, образует совершенные дуплексы только с комплементарными ему олигонуклеотидными зондами. Сигнал флюоресценции детектируется только в ячейках, в которых образовался совершенный дуплекс. Таким образом, при наличии в образце химерного гена, происходит его амплификация в ходе ПЦР и специфичная гибридизация с зондами на биочипе.
После проведения гибридизации и отмывки биочипа проводится визуализация результатов гибридизации с помощью любой детектирующей системы, возбуждающей флюоресценцию и распознающей флуоресцентный сигнал (флуоресцентный микроскоп с ПЗС-камерой, лазерный сканер, портативный анализатор биочипов и т.п. коммерчески доступные флуоресцентные анализаторы, например, портативный анализатор биочипов, снабженный ПЗС-камерой и специальным программным обеспечением, производства ООО «БИОЧИП-ИМБ» (Москва, Россия).
Изготовление биочипов может осуществляться посредством последовательного нанесения на поверхность подложки из стекла ячеек акриламидного геля, активации ячеек и иммобилизации в ячейках модифицированных олигонуклеотидов [Analysis of SNPs and other genomic variations using gel-based chips / A. Kolchinsky, A. Mirzabekov // Hum Mutat. - 2002. - Vol. 19. - P. 343-360. Review]. В качестве подложки кроме стекла может быть использован другой материал, в том числе металл, гибкие мембраны и пластик (Патент RU 2309959, опубликован 2007-11-10). Биочипы также могут быть изготовлены любыми другими известными специалисту в данной области способами [Arrays of immobilized oligonucleotides--ontributions to nucleic acids technology / H. Seliger, M. Hinz, E. Happ // Curr Pharm Biotechnol. - 2003. - Vol.4. - P. 379-395].
Для изготовления биочипа в настоящем изобретении используется набор олигонуклеотидов SEQ ID NO: 76-138, приведенный в Перечне последовательностей, а также в табл. 2. Расположение конкретных олигонуклеотидных зондов на биочипе может варьироваться в зависимости от удобства интерпретации результатов.
Далее приводятся примеры, которые показывают применение способа выявления структурных перестроек генома опухолевых клеток, определяющих выбор терапии и прогноз при острых лейкозах у детей. Следует понимать, что приводимые примеры служат исключительно для иллюстрации и не предназначены для ограничения объема притязаний, выраженных в формуле изобретения. На основании настоящего описания специалист в данной области сможет легко предложить свои варианты и модификации осуществления изобретения, не отходя от общей концепции настоящего изобретения и без привлечения собственной изобретательской деятельности, так что должно быть понятно, что такие варианты и модификации также будут входить в объем притязаний настоящего изобретения.
Пример 1. Амплификация участков химерных генов методом ОТ-ПЦР с целью получения флуоресцентно меченного ПЦР-продукта в количестве, неоходимом для гибридизации на биочипе.
Из костного мозга пацента, больного острым лейкозом, выделяли общую РНК с помощью набора RNeasy MiniKit (Qiagen, США).
Проводили обратную транскипцию в объеме 25 мкл. РНК (2 мкг) инкубировали при 70°С в течение 5 мин со смесью специфичных праймеров (по 5 пикомоль каждого праймера на реакцию), затем проводили реакцию с обратной транскриптазой M-MLV («Силекс», Россия) 200 ед. на 25 мкл реакционной смеси, содержащей 25 ед. ингибитора РНКазы («Promega», США), по 1 мМ каждого dNTP («Силекс», Россия), 10 мМ дитиотрейтол, 50 мМТ рис-HCl, рН 8.3, 75 мМ KCl и 3 мМ MgCl2 при 42°С в течение 90 мин с последующей инактивацией при 70°С в течение 10 мин.
Наработку ПЦР-продуктов, соответствующих последовательности контрольного гена ABL1 и исследуемых химерных генов проводили методом ПЦР в 4 реакциях. ПЦР проводили на приборе Dyad («Bio-Rad», США). ПЦР-смесь общим объемом 25 мкл включала в себя: 1×ПЦР-буфер (67 мМ Трис-HCl, рН 8.6, 166 мМ (NH4)2SO4, 0,01% Тритон Х-100), 1.5 мМ MgCl2, 0.2 мМ каждого из дНТФ («Силекс», Россия), 1 ед. акт. HotTaq-полимеразы («СибЭнзим», Россия), по 2 пмоль каждого локус-специфичного праймера, праймеры-адаптеры Т3 и Т7 по 1 и 50 пмоль соответственно, 1 мкл кДНК. Амплификацию проводили по следующей схеме: денатурация при 95°С (10 мин); 45 циклов: 94°С (30 с), 62°С (40 с), 72°С (30 с); 40 циклов: 94°С (30 с), 52°С (30 с), 72°С (20 с).
Пример 2. Олигонуклеотидный биочип для выявления структурных перестроек генома опухолевых клеток.
Олигонуклеотиды для иммобилизации на микрочипе синтезируют на автоматическом синтезаторе 394 DNA/RNA Synthesizer («Applied Biosystems», США) с использованием стандартной фосфоамидитной процедуры. 3'-конец олигонуклеотидов содержит спейсер со свободной аминогруппой, который вводят в состав олигонуклеотида при синтезе путем использования 3'-Amino-Modifier С7 CPG 500 («Glen Research)), США). Присоединение флуоресцентной метки к олигонуклеотидам по свободной аминогруппе осуществляют в соответствии с рекомендациями производителя.
Биочип изготовляют методом сополимеризации олигонуклеотида в акриламидном геле, как описано ранее в патентах RU 2309959 (дата публикации 2007-11-10) и RU 2175972 (дата публикации 2001-11-20). Биочип содержит 63 иммобилизованных олигонуклеотидных зонда (SEQ ID NO: 76-138), список которых представлен также в табл.2. Ячейки наносят согласно схеме на Фиг. 1.
Пример 3. Гибридизация меченного продукта на биочипе
Реакционную смесь, полученную после проведения ПЦР, описанного в Примере 1, используют для гибридизации на биочипе. Гибридизационная смесь общим объемом 44 мкл состояла из 6х SSPE («Promega», США), 2М раствора гуанидин-изотиоцианата и амплификата. Гибридизационную смесь денатурировали при 95°С 5 минут, охлаждали во льду 2 минуты, наносили на биочип и оставляли на 12 часов при 37°С. После проведения гибридизации биочип отмывали в 1x SSPE в течение 10 мин при комнатной температуре.
Пример 4. Регистрация и интерпретация результатов гибридизации
Регистрацию гибридизационной картины производят с помощью портативного анализатора биочипов, снабженного ПЗС-камерой, производимого ООО «Биочип-ИМБ». Описание алгоритма автоматического анализа изображения с помощью программы Image Ware™ выходит за рамки настоящего изобретения.
Пример картины гибридизации для образца с транслокацией t(8;21) RUNX1/RUNX1T1 приведен на Фиг. 2 (А). Наблюдается свечение ячеек, соответствующих последовательностям химерного гена RUNX1/RUNX1T1 и в ячейках, соответствующих последовательности контрольного гена ABL1. Пример картины гибридизации для контрольного образца без транслокации приведен на Фиг. 2 (Б). Наблюдается свечение ячеек только контрольного гена ABL1.
Таблица 1. Последовательности праймеров для обратной транскипции
| SEQ ID NO: | Название | Последовательность 5’->3’ |
| 1 | PBX1:476 | GCCACGCCTTC |
| 2 | MYH11:1555L12 | GTACTGCTGGGT |
| 3 | MYH11:2270L13 | AGCTGCTTGATGG |
| 4 | ORA (ABL) | GGACACACCATAGACAGT |
| 5 | AF4:1719L14 | CACTGTCACTGTCC |
| 6 | AML1A:875L15 | CGGTAGCATTTCTCA |
| 7 | RARA:698L12 | CGGTCGTTTCTCAC |
| 8 | ETO:648L12 | AGTGGAGTTCACTAG |
| 9 | AF9:1541L12 | CTATCGCTGCCAT |
| 10 | AF9:1910L12 | GATGCATCCAGTTGT |
| 11 | ELL:454L14 | CCAGCCTTGATGAC |
| 12 | ENL:138L14 | CCTTCTCCACGAAG |
| 13 | NPMALK:714L12 | CAGCGAACAATGTTC |
| 14 | TAL1:690L | TCGTCTCCTTGG |
| 15 | AF10:1150L12 | ACCTGAGCTGTG |
| 16 | AF10:2392L12 | CCACTGCCTCTC |
| 17 | AF10:750L13 | GTAGCCACAGTAT |
| 18 | AF6:355L12 | CCGATCATCTTT |
| 19 | RARA:589 | CGGTGACACGTGTAC |
| 20 | EPS15 | CTGAACAAACAAGAAT |
| 21 | AF1q | CTGAAGGTGATGGC |
| 22 | DEK/CAN | GAGACACTGTTTAAC |
| 23 | FUS/ERG | GCGCTGGGGAGAG |
| 24 | RBM15/MKL1 | GAGACCTCGGCTG |
| 25 | TCF3-HLF | GCCTCGTTCCTG |
Таблица 2. Последовательности праймеров для ПЦР
| SEQ ID NO: | Название | Последовательность 5’->3’ |
| 26 | MLL 3964-T7 | GTAATACGACTCACTATAGGACCACTCCTAGTGAGCCCAA |
| 27 | MLL 4178-T7 | GTAATACGACTCACTATAGGTTCCAGCAGATGGAGTCCACA |
| 28 | MLL 4252-T7 | GTAATACGACTCACTATAGGGGAGGCTTAGGAATCTTGACTTC |
| 29 | AF4 1629-T3 | AACCCTCACTAAAGGGAGAGTGAGTTTTTGAAGATGTATCATATTGT |
| 30 | L-AF6 257-T3 | AACCCTCACTAAAGGGAGAGTGAATACTTGGGAGAGGACA |
| 31 | ELL 326-T3 | AACCCTCACTAAAGGGAGACAGGCAGTCCAGGTGAACTT |
| 32 | ENL 121-T3 | AACCCTCACTAAAGGGAGAAAGTGCTGGATGTCACATTG |
| 33 | CBFB344-T7 | GTAATACGACTCACTATAGGGGGCTGTCTGGAGTTTGATG |
| 34 | II_Rj1-T3 | AACCCTCACTAAAGGGAGAAGCTGCGTCTTCATCTCCTC |
| 35 | MYH 1387-T3 | AACCCTCACTAAAGGGAGATCCTCGTCCAGCTGGTCTTG |
| 36 | ABL Fw-T7 | GTAATACGACTCACTATAGGAGAGTGAGAGCAGTCCTGGCCAG |
| 37 | ABL Rw-T3 | AACCCTCACTAAAGGGAGAGAGACGTAGAGCTTGCCATCAGAA |
| 38 | MLL Fw1-T7 | GTAATACGACTCACTATAGGACCGCCAAGAAAAGAAGTTCCCA |
| 39 | MLL Fw3-T7 | GTAATACGACTCACTATAGGCAGCAGATGGAGTCCACAGGAT |
| 40 | L-AF10 823-T3 | AACCCTCACTAAAGGGAGAGTGAGAGGAAGAATCACTTAAACTTTG |
| 41 | L-AF10 1032-T3 | AACCCTCACTAAAGGGAGAGCATTTGTAAATCGTGCAGTAGTATCT |
| 42 | L-AF10 2327-T3 | AACCCTCACTAAAGGGAGAAACTGCTGTTGCCTGGTTGAT |
| 43 | PICALM Fw_B-T7 | GTAATACGACTCACTATAGGGGATCTAACTGGCAACCAAAGGT |
| 44 | PICALM Fw_A+C-T7 | GTAATACGACTCACTATAGGGAAGTGTTCCTGTAATGACGCAACC |
| 45 | SIL_TAL Fw-T3 | AACCCTCACTAAAGGGAGAGCGACCCCAACGTCCCAGAGGC |
| 46 | SIL_TAL Rw-T7 | GTAATACGACTCACTATAGGATGCACACCCGGGTCTTTGCTTTCC |
| 47 | DEK_CAN Fw-T3 | AACCCTCACTAAAGGGAGACCCCTACAGATGAAGAGTTAAAGGAAA |
| 48 | DEK_CAN Rw-T7 | GTAATACGACTCACTATAGGAACATCATTCACATCTTGGACAGCAA |
| 49 | E2A_HLF_ex12 Fw-T3 | AACCCTCACTAAAGGGAGAACACGCTGCTGCCTGGCCACGG |
| 50 | E2A_HLF_ex13 Fw-T3 | AACCCTCACTAAAGGGAGATCATGCACAACCACGCGGCCCT |
| 51 | E2A_HLF Rw-T7 | GTAATACGACTCACTATAGGGACGCTTGGCTGCCATGTTGTTC |
| 52 | TEL/AML1 Fw_v2-T7 | GTAATACGACTCACTATAGGCGGGAAGACCTGGCTTACATGAACC |
| 53 | TEL/AML1 Rw v2-T3 | AACCCTCACTAAAGGGAGACCTCGCTCATCTTGCCTGGG |
| 54 | p190 Fw v1-T7 | GTAATACGACTCACTATAGGGCAGATCTGGCCCAACGATG |
| 55 | p190 Rw-T3 | AACCCTCACTAAAGGGAGATGGAGTTCCAACGAGCGGCTTC |
| 56 | p210 Fw v3-T7 | GTAATACGACTCACTATAGGGCTGCAGATGCTGACCAACTCG |
| 57 | PML/RARA Fw1-T7 | GTAATACGACTCACTATAGGCGCCTGCAGGACCTCAGCTCTT |
| 58 | PML/RARA Fw2-T7 | GTAATACGACTCACTATAGGACAGCAACCACGTGGCCAGT |
| 59 | PML/RARA Rw-T3 | AACCCTCACTAAAGGGAGAAGGCTTGTAGATGCGGGGTAGAG |
| 60 | AML/ETO Fw-T7 | GTAATACGACTCACTATAGGCATCAAAATCACAGTGGATGGGCC |
| 61 | AML/ETO Rw-T3 | AACCCTCACTAAAGGGAGAGAGGTGGCATTGTTGGAGGAGTCAG |
| 62 | E2A/PBX1 Fw-T7 | GTAATACGACTCACTATAGGCACCCTCCCTGACCTGTCTCG |
| 63 | E2A/PBX1 Rw-T3 | AACCCTCACTAAAGGGAGACATGTTGTCCAGCCGCATCAG |
| 64 | AF9(I) 1818-T3 | AACCCTCACTAAAGGGAGATCTTCTATAAGGTTCACGATCTGCT |
| 65 | AF9(2) 1426-T3 | AACCCTCACTAAAGGGAGATCCTCCTCATTGTCATCAGAATG |
| 66 | EPS Rw _2-T3 | AACCCTCACTAAAGGGAGACAGCAGCATCAGAAGCCAACACCCT |
| 67 | AF1q Rw-T3 | AACCCTCACTAAAGGGAGACCTTCCAGCTCCGACAGATCCAGTT |
| 68 | NPM 403U21-T3 | AACCCTCACTAAAGGGAGAGGTTCAGGGCCAGTGCATATT |
| 69 | NPMALK 590L19-T7 | GTAATACGACTCACTATAGGCTTGGGTCGTTGGGCATTC |
| 70 | RBM15_MKL1 Fw-T3 | AACCCTCACTAAAGGGAGACAGTTCCTGGATTCCCCTGCCA |
| 71 | RBM15_MKL1 Rw-T7 |