Способ скрининга противоопухолевых препаратов - ингибиторов parp1 на основе биохимических методов анализа
Иллюстрации
Показать всеИзобретение относится к биохимии, в частности к способу скрининга противоопухолевых препаратов - ингибиторов PARP1. Для осуществления указанного способа проводят сборку нуклеосом из очищенных гистонов на ДНК-матрицах, затем лигирование с РНК-полимеразой и внесение в полученный комплекс молекулярной мишени - белка PARP1, с последующим внесением буфера транскрипции, а также тестируемых химических соединений. При этом указанный буфер транскрипции содержит дезоксинуклеотидтрифосфаты (дНТФ) и меченый α-32Ф-ГТФ. Далее проводят транскрипцию и оценку продуктов транскрипции путем их разделения с помощью электрофореза. Вывод о наличии у тестируемого соединения PARP1-ингибирующей активности делают при наличии на электрофореграмме полос в точках +15, +45 и в области run-off, показывающих паузирование транскрипции, как показано на фиг. 1, причем указанную оценку активности проводят с референсным значением PARP1 с НАД. Настоящее изобретение позволяет повысить специфичность и селективность системы отбора молекул на основе нуклеосом. 18 з.п. ф-лы, 2 ил., 7 пр.
Реферат
Область техники, к которой относится изобретение
Изобретение относится к области медицинской и молекулярной биологии, преимущественно к области регуляции экспрессии генов на уровне транскрипции, и может быть использовано, например, для поиска противоопухолевых препаратов, направленных на PARP1 с использованием способа, основанного на применении системы из специальных нуклеосомных матриц, белка PARP1 и буфера внесения. При внесении различных соединений в данную систему может происходить взаимодействие данных веществ с PARP1 и, соответственно, ухудшение связывания PARP1 с нуклеосомными матрицами, что в дальнейшем позволит обнаружить ингибиторы PARP1.
Уровень техники
В основе поиска и получения современных лекарственных средств лежит нацеленность на молекулярную мишень. Одна из мишеней, используемых при разработке противоопухолевых препаратов, - фермент поли(ADP-рибозо)полимераза 1 (PARP1), участвующий во многих клеточных процессах, начиная от репарации ДНК до гибели клеток [Kraus W.L., Hottiger М.O. // Mol. Aspects Med. 2013. V. 34. P. 1109-1123]. Недавно показали, что одним из самых ранних событий, происходящих при повреждениях ДНК, является узнавание ферментом PARP1 разрывов ДНК. При возникновении разрывов ДНК, вызванных, в частности, алкилирующими агентами и радиацией, PARP1 связывается с местами разрывов за счет так называемых «цинковых пальцев», расположенных в ДНК-связывающем домене и одновременно синтезирует олиго- или поли(ADP-рибозные) цепочки, ковалентно связываемые с разными акцепторными белками или с собственной молекулой путем перемещения единицы ADP-рибозы от НАД+. В результате в месте разрыва происходит деконденсация хроматина, что облегчает доступ ферментов репарации. Модифицированные поли(ADP-рибозил)ированные белки хроматина привлекают факторы, ремоделирующие хроматин. Следует отметить, что репарация ДНК при активном участии PARP1 происходит лишь при минимальном генотоксическом повреждении. При более сильном повреждении запускается процесс апоптоза, а при обширном повреждении ДНК наблюдается сверхактивация PARP, приводящая к некрозу.
Получено множество данных об участии PARP1 в канцерогенезе. Потеря PARP1 приводит к нарушениям процесса репарации ДНК, ингибированию транскрипции некоторых генов, вовлеченных в репликацию ДНК, регуляцию клеточного цикла. Повышенная экспрессия PARP1 наблюдается в меланомах, опухолях легкого и молочной железы [, Peralta-Leal A, O'Valle F, Rodriguez-Vargas JM, Gonzalez-Flores A, Majuelos-Melguizo J, , Serrano S, de Herreros AG, Rodriguez-Manzaneque JC, et al. // PLoS Genet. 2013. V. 9. №6. P. el003531; Nowsheen S, Cooper T, Stanley JA, Yang ES. // PLoS One. 2012. V. 7. №10. P. e46614; Galia A, Calogero AE, Condorelli R, Fraggetta F, La Corte A, Ridolfo F, Bosco Ρ, Castiglione R, Salemi M. // Eur. J. Histochem. 2012. V. 56. №1. P. e9; Csete Bl, Lengyel Z, , Battyáni Ζ. // Pathol. Oncol. Res. 2009. V. 15. №1. P. 47-53; Telli ML, Ford JM. // Clin. Breast Cancer. 2010. V. 10 Suppl 1. №P. E16-22; Shimizu S, Nomura F, Tomonaga T, Sunaga M, Noda M, Ebara M, Saisho H. // Oncol. Rep.2004. V. 12. P. 821-825]. При этом повышенный уровень экспрессии рассматривается как прогностический признак, связанный с худшим прогнозом выживаемости [Rojo F, Garcia-Parra J, Zazo S, Tusquets I, Ferrer-Lozano J, Menendez S, Eroles Ρ, Chamizo С, Servitja S, Ramirez-Merino Ν, et al. // Ann. Oncol. 2012. V. 23. №P. 1156-1164]. Было показано, что высокий уровень экспрессии PARP1 коррелирует с более агрессивным фенотипом злокачественных опухолей молочной железы (РМЖ) (эстроген-негативный тип РМЖ) [Domagala Ρ, Huzarski Τ, Lubinski J, Gugala K, Domagala W. // Breast Cancer Res. Treat. 2011. V. 127. P. 861-869]. Экспрессия PARP1 может коррелировать с устойчивостью опухолей к терапии [Michels J, Vitale I, Galluzzi L, Adam J, Olaussen KA, Kepp O, Senovilla L, Talhaoui I, Guegan J, Enot DP, et al. // Cancer Res. 2013. V. 73. P. 2271-2280]. Подобная более высокая «злокачественность» связана, видимо, с тем, что повышенная экспрессия PARP1 способствует репарации повреждений ДНК и тем самым преодолению генетической нестабильности, свойственной трансформированным клеткам.
Практически все существующие ингибиторы PARP1 являются миметиками никотинамида, т.е. ориентированы на связывание с каталитическим доменом PARP1 и конкуренцию с НАД+. При проведении расширенных клинических испытаний ингибиторов PARP1 вскрылся целый ряд проблем. Во-первых, соединения, ингибирующие связывание НАД+, имеют довольно низкую специфичность к PARP1, а также блокируют другие ферментативные пути с участием НАД+. Следует отметить, что НАД+ - это кофактор, который взаимодействует со многими ферментами, вовлеченными в ряд клеточных процессов, поэтому конкуренция с НАД+ приводит к высокой токсичности. Во-вторых, остается открытым вопрос о безопасности длительного применения существующих ингибиторов PARP1. Известно, что опухолевые клетки обладают способностью быстро приобретать устойчивость к препаратам, применяемым в качестве длительной монотерапии [Mandery K., Fromm M.F. // Br. J. Pharmacol. 2012. V. 165. Р. 345-362]. Эти проблемы стали причиной того, что многие ингибиторы PARP1 не прошли длительные систематические клинические испытания. Испытания некоторых ингибиторов PARP1 прекращены уже на I и II стадиях из-за высокой токсичности и ряда побочных эффектов. Обнаруженный в КИ ряд побочных эффектов заставляет менять стратегию разработки новых ингибиторов PARP1. Поскольку PARP1 состоит из нескольких функциональных доменов и обладает дополнительными активностями, помимо ферментативной, то активность PARP1 можно регулировать ингибированием данных функциональных доменов. В частности, разрабатываются препараты, направленные на ингибирование связывания PARP1 с ДНК [Kirsanov ΚΙ, Koto va Ε, Makhov Ρ, Golovine K, Lesovaya EA, Kolenko VM, Yakubovskaya MG, Tulin AV. // Oncotarget. 2014 V.5. P. 428-437]. PARP1 играет также ключевую роль в регуляции транскрипции [Maluchenko N.V., Kotova Ε., Chupyrkina A.A., Nikitin D.V., Kirpichnikov M.P., Studitsky V.M. // Mol. biol. Mosc. 2015. V. 49. №1. P. 1-15; Kotova E., Tulin A.V. // Proc. Natl. Acad. Sci. USA. 2010. V. 107. №14. P. 6406-6411]. По мнению авторов, поиск соединений, способных предотвращать участие PARP1 в процессе транскрипции, может привести к разработке нового класса лекарственных средств, имеющих более высокую специфичность и менее выраженные побочные эффекты. Используя полученную ранее авторами систему транскрипции в нуклеосомных системах, становится возможным обнаружение новых ингибиторов PARP1.
Нуклеосомные поверхности можно использовать в качестве основы для разработки способа скрининга соединений, вызывающих изменение транскрипционной активности различных белков, в частности PARP1.
Известен способ использования нуклеосом в качестве биосенсоров [United States Patent Application 20090062130 Nucleosome-based biosensors // Application Number 11/687859]. Подобный биосенсор состоит, по крайней мере, из одной нуклеосомы, состоящей из ДНК-регулирующего транскрипционного элемента, в котором ДНК помечена двумя метками (а гистоновый октамер остается немеченым). В ДНК введена последовательность, взаимодействующая с лигандом ядерных рецепторов. Система предложена для высокопроизводительного скрининга определенных веществ агонистов и антагонистов ядерных рецепторов, что ограничивает использование данного способа.
Авторами [Na Ζ, Peng В, Ng S, Pan S, Lee JS, Shen HM, Yao SQ // Angew Chem Int Ed Engl. 2015, V. 54. P.2515-2519] предложен способ поиска ингибиторов, направленных на BRCT. Использовали пептидную библиотеку (около 100 пептидов), в которую вносили фрагмент BRCT белка PARP1. По результатам скринирования отобрали наиболее активно BRCT-связывающий пептид. На микропланшетах, в каждую лунку вносили данный пептид и затем тестировали низкомолекулярные соединения, отбирали наиболее активно дестабилизирующие взаимодействие пептида с BRCT. Таким образом, был обнаружен ряд новых ингибиторов PARP1. Среди них самая высокая активность наблюдалась у (±) госсипола (IC50=0,54±0,06 мкМ). Этот способ перспективен для поиска новых ингибиторов PARP1, но направленность на BRCT домен в существенной степени ограничивает круг поиска новых ингибиторов PARP1.
Известен также способ, в котором новые ингибиторы PARP1 определяют с помощью иммуноферментного анализа [Kotova Ε., Tulin A.V. // Meth. Mol. Biol. 2011. V. 780. P. 491-516]. Способ заключается в нанесении на 96-луночный планшет для ИФА раствора, содержащего гистон Н4, затем следует отмывка фосфатно-солевым буфером, далее внесение различных концентраций PARP1 с дальнейшим определением комплекса Н4 и PARP1 с помощью меченых антител, направленных к PARP1. Этот способ позволяет определять ингибиторы PARP1, которые разрушают интерфейс между данным белком и гистоном Н4. Ограничение данного метода заключается в том, что он не позволяет выявлять ингибиторы PARP1, которые направлены к другим участкам нуклеосомы (поверхность Н2А/Н2 В димера гистонов, Н3 гистона, HI и др. функционально активные области нуклеосомы).
Наиболее близким техническим решением (прототипом) является метод, который позволяет определять способность веществ различного происхождения стабилизировать или дестабилизировать элонгационный комплекс РНК-полимеразы с помощью определения ∅-петли [United States Patent Application US 20120045429 Al //. Methods and Agent for Modulating the RNA Polymerase II-Histone Surface // V. Studitsky, O. Studitskaia, D. Gaykalova]. На основе предложенного подхода нуклеосомы могут быть использованы для определения агентов, влияющих на процесс прохождения транскрипции в искусственно созданной системе (ИНСТИВ - искусственная нуклеосомная система транскрипции in vitro). Для осуществления данного метода были разработаны специальные нуклеосомные матрицы [Kulaeva OI, Gaykalova DA, Pestov NA, Golovastov VV, Vassylyev DG, Artsimovitch I, Studitsky VM Mechanism of chromatin remodeling and recovery during passage of RNA polymerase II // Nat Struct Mol Biol.- 2009.- V.16.- P. 1272-1278; Bondarenko VA, Steele LM, Ujvari A, Kulaeva OI, Studitsky VM Nucleosomes can form a polar barrier to transcript elongation by RNA polymerase II // Mol Cell. - 2006. - V. 24. - P. 469-479; Kireeva ML, Walter W, Tchernajenko V, Bondarenko V, Kashlev M Studitsky VM. Nucleosome remodeling induced by RNA polymerase II. Loss of the H2A/H2B dimer during transcription // Mol Cell. 2002. - V. 9. - P. 541-552; Kulaeva OI, Hsieh FK, Studitsky VM. RNA polymerase complexes cooperate to relieve the nucleosomal barrier and evict historie //Proc Natl Acad Sci USA. - 2010. - V. 107.- P. 11325-11330; Studitsky, V. M. Preparation and analysis of positioned nucleosomes// Methods Mol. Biol. - 1999. - V. 119. - P. 17-26; Gaykalova D., Kulaeva O.I., Bondarenko VA. Studitsky V.M. Preparation and analysis of uniquely positioned nucleosomes// Methods Mol. Biol. - 2009. - V. 523. - P. 109-123; Walter, W., Studitsky, V. M. Construction, analysis, and transcription of model nucleosomal templates // Methods. - 2004. - V. 33. - P. 18-24]. Вышеупомянутый способ включает внесение тестируемого агента в соответствующем буфере в ИНСТИВ, инкубации и тестирования образования ∅-петли. Данный способ основан на данных, что в ИНСТИВ запускается механизм, включающий формирование промежуточного интермедиата - ∅-петли, который облегчает прохождение РНК полимеразы по ДНК, навернутую на нуклеосому (что имитирует процесс прохождения РНК полимеразы в хроматине в естественных условиях в клетке). Предполагается, что нарушения в системах, контролирующих сохранение хроматина в процессах репликации и транскрипции, в том числе нарушение образования ∅-петли, приводят к развитию различных патологий, включая опухолеобразование и преждевременное старение клетки. Таким образом, вещества, которые будут способствовать корректному формированию ∅-петли, могут быть потенциальными противоопухолевыми агентами. Есть ряд недостатков способа, предложенного в US 20120045429 Al, которые могут ограничивать внедрение и практическое применение данного способа. Ограничения связаны с трудностью детекции образования ∅-петли. Есть небольшое число методов, достоверно и напрямую детектирующих наличие ∅-петли. Как правило, это высокотехнологичные методы, такие как электронная микроскопия, рентгено-структурный анализ и, возможно, зондовая сканирующая микроскопия (очень высокого разрешения). Эти методы трудоемки и трудозатраты, требуют привлечения высококвалифицированных специалистов для проведения и интерпретации результатов. Кроме того, занимают продолжительное время и дорогостоящие при реализации. Есть более экономичные методы детекции ∅-петли (например, с помощью ДНКаза1 футпринтинга), но при их реализации происходит потеря в достоверности и повышение погрешности измерений. Кроме того, идея использовать непосредственно сами нуклеосомные поверхности в качестве основы для скрининга веществ, обуславливает низкую специфичность и селективность такой системы отбора молекул. Недостатки данного способа можно преодолеть, если данную систему модифицировать путем введения конкретной молекулярной мишени, против которой осуществлять скрининг соединений. И вторым необходимым преобразованием способа, предложенного в US 20120045429 A1, должно быть значительное упрощение в системе детекции без потери в чувствительности, специфичности и достоверности. Способ использования нуклеосомных матриц US 20120045429 A1 был выбран в качестве прототипа предложенного решения. Но предлагаемый способ устраняет вышеперечисленные недостатки за счет введения в систему ИНСТИВ конкретной молекулярной мишени, против которой будут тестироваться соединения и за счет упрощения детекции продуктов в системе.
Раскрытие изобретения
Задачей предлагаемого технического решения является создание способа простого в реализации, не требующего дорогостоящих реактивов, при сохранении достоверности результатов.
Поставленная задача решается способом скрининга противоопухолевых препаратов - ингибиторов PARP1, включающим сборку нуклеосом из очищенных гистонов на ДНК-матрицах, лигирование с РНК-полимеразой, внесение в полученный комплекс молекулярной мишени - белка PARP1, с последующим внесением буфера транскрипции, содержащего дезоксинуклеотидтрифосфаты (дНТФ) и меченый α-32Ф-ГТФ, a также тестируемых или контрольных химических соединений, проведение транскрипции, оценку продуктов транскрипции.
Предпочтительно РНК полимераза, используемая в способе, является РНК полимеразой 2 натурального или рекомбинантного происхождения из различных организмов, включая дрожжей, млекопитающих, пресмыкающихся и др.
Возможен вариант использования в качестве РНК полимеразы РНК полимеразы натурального или рекомбинантного прокариотического происхождения, например РНК полимеразы Е. coli.
Предпочтительно для сборки нуклеосомных матриц использовать очищенные препараты гистонов Н2А, Н2В, Н3, Н4 натурального происхождения из различных организмов, включая дрожжей, млекопитающих, пресмыкающихся.
Предпочтительно, что при использовании очищенных гистонов Н3 и Н4 их добавляют в двухкратном, а очищенные гистоны Н2А и Н2В в трехкратном избытке по отношению к матричной ДНК.
Возможен вариант, когда для сборки нуклеосомных матриц дополнительно используют гистон HI как натурального, так и рекомбинантного происхождения.
Предпочтительно для сборки нуклеосомных матриц использовать мутантные гистоновые белки, например Sin-мутанты.
Возможен вариант, когда осуществляют сборку неполных нуклеосом, например гексасом или тетрасом.
Предпочтительно для сборки гибридных нуклеосомных матриц использовать рекомбинантные или синтетические гистоны эукариотического и прокариотического происхождения.
Возможен вариант, когда для сборки нуклеосом используют ДНК- матрицу, несущую мутации, ассоциированные с различными заболеваниями или со старением.
Еще одним вариантом воплощения изобретения является введение в систему вместе с тестируемым агентом факторов, ремоделирующих хроматин, например SWI/SNF, ISW2, ACF и др.
Предпочтительно сборку нуклеосом проводить на поверхности лунок планшета или зерен сорбента.
Предпочтительно оценку продуктов транскрипции проводить биохимическими методами анализа, например электрофоретически.
Возможен вариант, когда перед оценкой продуктов транскрипции, дополнительно проводят обработку смеси реагентами для футпринтинга, например гидроксилами или ДНКАазой1.
Предпочтительно перед внесением агентов в систему проводят предварительное математическое моделирование.
Технический результат предлагаемого изобретения заключается в снижении погрешности измерений, а также в повышении их достоверности, а также в повышении специфичности (селективности) системы. Кроме того, функциональные возможности использования ИНСТИВ для тестирования противоопухолевых соединений расширены за счет введения дополнительного компонента - белка PARP1. Снижение погрешности измерений и повышение достоверности происходит за счет того, что в системе детектируется не образование ∅-петли, а образование транскриптов, образующихся при работе РНК полимеразы в системе.
Указанный технический результат достигается тем, что применяется способ использования системы ИНСТИВ, включающий:
1) сборку нуклеосом из очищенных гистонов на ДНК-матрицах,
2) сшивку с РНК-полимеразным комплексом,
3) внесение молекулярной мишени - белка PARP1,
4) внесение различных химических соединений, агентов, способных регулировать транскрипционную активность PARP1,
5) внесение буфера, содержащего все четыре буквы в виде дезоксинуклеотидтрифосфатов (дНТФ), необходимых для построения РНК-транскриптов, а именно «АТФ», «ТТФ», «УТФ», «ГТФ», плюс одной меченой буквы, в данном случае - «α-32Ф-ГТФ». Подробный состав буфера для транскрипции приведен в разделе «Методика реализации способа»,
6) оценку продуктов транскрипции, и отличающийся тем, что способ используют для тестирования ингибиторов PARP1 - потенциальных противоопухолевых препаратов.
В такой модификации (после добавления PARP1) система будет обозначаться как ИНСТИВ_PARPl. Тестируемые и контрольные вещества вносят согласно описанному ниже протоколу (см. «Методика реализации способа»). Любые вещества различного происхождения (высокомолекулярные, низкомолекулярные, экзогенные, эндогенные и пр.), приводящие в тестируемой системе к изменениям транскрипционной активности PARP1 будут рассматриваться как потенциальные противоопухолевые агенты. Таким образом, в предлагаемой тестируемой системе возможно осуществление скрининга большого количества веществ с целью последующего отбора лекарственных препаратов.
Возможен вариант, когда транскрипцию проводят различными РНК полимеразами натурального или рекомбинантного происхождения, из различных видов животных, например РНК полимеразу 2 (РНКП2) дрожжей, пресмыкающихся, млекопитающих и др.
Транскрипцию также можно проводить различными РНК полимеразами натурального или рекомбинантного прокариотического происхождения, например РНК полимеразу Е. Coli.
Возможно применение варианта, в котором для сборки нуклеосомных матриц используют рекомбинантные гистоны эукариотического и прокариотического происхождения, включая использование коммерческих препаратов фирм INVITROGENE, CLONTECH, STRATAGENE, PROMEGA и др. Рекомбинантные гистоны могут содержать различные модификации и быть очищены различными способами. Также могут быть использованы синтетические гистоны, полученные с помощью автоматических белковых синтезаторов.
Возможен вариант, в котором сборка нуклеосомных матриц происходит на поверхности лунок планшета или зерен сорбента, при этом нуклеосомы оказываются ковалентно или нековалетно прикреплены к твердой поверхности носителя. Применение носителей позволяет оптимизировать разделение компонентов смеси и облегчить процедуру отмывок от нежелательных компонентов и примесей. Твердые поверхности могут быть изготовлены из различных материалов, таких как полистирол, поливинилхлорид, полипропилен, целлюлоза, нитроцеллюлоза, нейлон, гели декстрана, полиакриламида, агарозы и др. Твердая фаза может быть в виде пробирок, планшетов для микротитрования, шариков, пленок и др.
Можно использовать вариант, в котором к твердой поверхности прикрепляют ДНК, а не нуклеосомы.
Оценку продуктов транскрипции или интермедиатов проводят электрофоретически.
Способ использования специальных функциональных нуклеосомных матриц для проведения скрининга для тестирования ингибиторов PARP1 реализуется следующим образом (см. раздел «Методика реализации способа»).
Реализация данного способа может служить основой для получения коммерческой системы поиска и функциональной проверки новых противоопухолевых лекарственных препаратов, направленных на PARP1. Известно, что получение лекарств - долгий и дорогостоящий процесс, при этом из 5-10 тысяч тестируемых соединений лишь 0,1% доходит до стадии клинических испытаний, в результате которых только 10-20% разрешают к применению. Большинство молекул отсеивается на стадии преклинических исследований по показателям эффективности и безопасности. Доклинические эксперименты (на культурах клеток, тем более животных моделях) являются высокозатратными, трудоемкими и длительными. Предлагаемая в данном способе система ИНСТИВ_PARPl является миниатюрной функциональной системой, воспроизводящей клеточный процесс транскрипции. Поскольку в данную систему внесена молекулярная мишень, то в такой модификации система позволяет проверять PARP1-ингибирующую активность соединений. Таким образом, использование данной системы позволит оптимизировать стадию «отсева» молекул по показателям эффективности, что в значительной степени может удешевить и ускорить процесс поиска новых лекарственны соединений.
Для оценки конкурентоспособности и патентоспособности результатов авторами заявки был проведен информационный поиск по патентным базам данных и по работам, опубликованным в открытой печати. Анализ результатов поиска показал, что предлагаемый подход не используется в исследовательской и медицинской практике, не описан в открытых источниках и не защищен патентами. Следовательно, предложение отвечает критериям новизны и изобретательского уровня.
Следует отметить, что ингибиторы PARP1 представляют большой интерес и имеют практическую ценность не только в онкологии, но и в терапии различных воспалительных процессов, сердечно-сосудистых и неврологических заболеваний, а также заболеваний, связанных со старением [Mouchiroud L., Auwerx J. // Crit. Rev. Biochem. Mol. Biol. 2013. V. 48. №4. P. 397-408; Biirkle A. // Mol. Aspects Med. 2013. V. 34. P. 1046-1065; Ma Y., He X., Nie H., Hong Y., Sheng C, Wang Q., Xia W., Ying W. // Curr. Drug Targets. 2012. V. 13. №2. P. 222-229; Ying W. // Scientifica (Cairo). 2013. V. 2013. Article ID 691251; Baxter P., Xu Y., Swanson R.A. // Transi. Stroke Res. 2014. V. 5. №1. P. 136-144; Rosado M.M., Novelli F., Pioli C. // Immunology. 2013. V. 139. №4. P. 428-137]. Поэтому использование данного способа в дальнейшем может позволить осуществлять поиск новых лекарственных субстанций, направленных не только против опухолей, но и обладающих другим спектром активностей.
Краткое описание чертежей
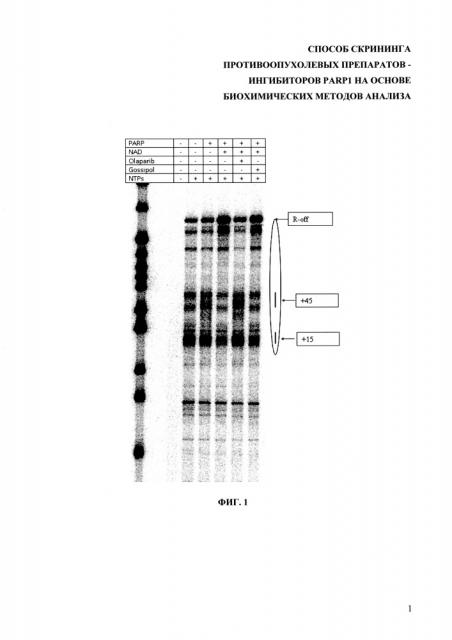
На фиг. 1 представлены результаты электрофореза в присутствии PARP1 и ингибиторов PARP1. Дорожки подписаны сверху. Первая дорожка - маркер молекулярного веса.
На фиг. 2 показан выход транскриптов в %. На фигуре: PARP-PARP1, NAD - НАД, Olap - олапариб, Goss - госсипол
Осуществление изобретения
Методика реализации способа
1. Очистка РНК- полимераз (РНКП)
Можно использовать дрожжевую РНК полимеразу 2 с концевым тагом из шести гистидинов. РНК полимеразу 2 выделяют согласно разработанному и опубликованному протоколу [Kireeva ML, L.L., Komissarova Ν, Kashlev Μ., Assays and affinity purification of biotinylated and nonbiotinylated forms of double-tagged core RNA polymerase II from Saccharomyces cerevisiae. Methods Enzymol., 2003. 370: p.138-55]. Либо можно использовать РНК полимеразу E.coli, которую выделяют из штамма бактерии RL721 согласно протоколу, опубликованному ранее [Kashlev, M., et al., Histidine-tagged RNA polymerase of Escherichia coli and transcription in solid phase. Methods Enzymol., 1996. 274: p.326-34]. Очищенные РНКП хранят небольшими аликвотами при -70°С.
2. Приготовление транскрипционных комплексов.
2.1. Приготовление раствора ДНК-РНК гибридной смеси, ДНК-РНК гибридную смесь готовят следующим образом. Синтетический РНК-нуклеотид RNA9 (5' AUCGAGAGG 3') и синтетический ДНК-нуклеотидом TDS 50 (5' GGTGTCGCTTGGGTTGGCTTTTCGGGCTGTCCCTCTCGATGGCTGTAAGT 3') соединяют в эквимолярных количествах (по 1.5 мкМ). Кратко: полученную смесь гибридизуют с помощью ПЦР по программе: 45°С - 5 мин, 42°С, 39°С, 36°С, 33°С, 30°С, 27°С, - 2 мин каждый шаг, 25°С - 10 мин. В результате получают ДНК-РНК гибридную смесь.
2.2. Обработка РНКП ДНК-РНК гибридной смесью
15 мкл 1,33 нМ ДНК-РНК гибридной смеси соединяют с 4 мкл раствора РНК полимеразы (либо РНК полимеразы 2, либо РНКП полимеразы E. coli. Инкубируют при комнатной температуре 15 мин.
2.3. Подготовка нематричной ДНК
В качестве нематричной (59ДНК) используют ДНК с последовательностью 5' ACTTACAGCCATCGAGAGGGACACGGCGAAAAGCCAACCCAAGCGA CACCGGCACTGGG 3'. Вносят 1 мМ 59 ДНК и инкубируют при комнатной температуре 15 мин.
2.4. Инкубация транскрипционного комплекса на Ni-NTA агарозе
Добавляют смесь, состоящую из ДНК-РНК гибридов и инкубированный вместе с ними раствор РНК полимеразы к подготовленной заранее Ni-NTA агарозе (Qiagen, Chatsworth, С А). Инкубируют при комнатной температуре 10 мин. Проводят однократную отмывку буфером ТВ40 с помощью центрифугирования 1 мин (1600 g Eppendorf). Супернатант удаляют. Проводят однократную отмывку буфером ТВ300 с помощью центрифугирования 1 мин (1600 g Eppendorf). Супернатант удаляют. Отмывают дважды буфером ТВ40 с помощью центрифугирования 1 мин (1600 g Eppendorf). Супернатант удаляют. Доводят до объема 30 мкл водой деионизованной. Добавляют к смеси раствор имидазола, инкубируют при комнатной температуре 5 мин. Центрифугируют в течение 1 мин (1600 оборотов/мин, Eppendorf, (Германия) и собирают супернатант. Доводят объем супернатанта до 21 мкл раствором ТВ40.
3. Приготовление ДНК-матриц для транскрипции
3.1. Подготовка ДНК-матриц
ДНК, содержащую нуклеосом-позиционирующую последовательность, получали из плазмид pGEM-3Z/601, pGEM-3Z/603 и GEM-3Z/605 [Walter, W., et al., Assay of the fate of the nucleosome during transcription by RNA polymerase II. Methods Enzymol., 2003. 371: p.564-77]. Фрагменты ДНК из плазмид амплифицируют с помощью праймеров [Lowary, Р.Т. and J. Widom, New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. J. Mol. Biol., 1998. 276(1): p.19-42] и очищают с помощью набора QIAfilter Plasmid Maxi Kit (Qiagen, Нидерланды) согласно протоколу фирмы-разработчика. Для сборки можно использовать различные типы матриц, например 601, 603, 605. Ранее было показано, что ДНК-последовательности данных матриц негомологичны, но все они обладали характерным свойством связывать коровые гистоны с высокой аффинностью и в точно определенных позициях на ДНК.
3.2. Присоединение промотерной части к ДНК-матрицам
Вначале готовят линкерный Т7 промотер-содержащий ДНК-фрагмент согласно протоколу [38] путем амплификации с соответствующими праймерами. Фрагменты ДНК из плазмид амплифицируют с помощью праймеров [Lowary, Р.Т. and J. Widom, New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. J. Mol. Biol., 1998. 276(1): p.19-42] и очищают с помощью набора QIAfilter Plasmid Maxi Kit (Qiagen) согласно протоколу фирмы-разработчика. Полученные PCR-продукты (Т7 промотер-содержащий ДНК-фрагмент и фрагмент, содержащий нуклеосом-позиционирующую последовательность) расщепляют с помощью рестриктазы TspRI, лигируют и проводят ПЦР с последующей очисткой ампликонов. Полученная ДНК-матрица содержит сильный Т7 промотер.
4. Сборка нуклеосом
Готовят буфера CRB, содержащих 10 мМ Tris-HCl (рН 8.0), 0.2 мМ ЭДТА, 5 мМ в-меркаптоэтанола, 0.1% NP-40, и различающиеся концентрации NaCl, а именно: в буфере CRB1 - 2М NaCl, в буфере CRB2 - 1,5М NaCl в буфере CRB3 - 1 M NaCl, в буфере CRB4 -0,75М NaCl, 0.75 M (CRB4); 0.5 M (CRB5); и 0.01 M (CRB6). Все буфера для проведения анализа охлаждают до 4°С. Растворяют 0.5-3 мкг ДНК-матрицы в буфере CRB1. Добавляют ДНК спермы лосося (Sigma-Aldrich, St.Louis, МО) в двухкратном избытке по отношению к матричной ДНК. Затем добавляют очищенные гистоны в молярном избытке, а именно очищенные гистоны Н3 и Н4 в двухкратном, а очищенные гистоны Н2А/H2B в трехкратном избытке по отношению к матричной ДНК. Реакционный объем доводят до 40-100 мкл и переносят в малые диализные мешки. Проводят диализ против CRB1 в течение ночи при +4°C. Затем проводят диализ против CRB2 в течение 1 часа при +4°C. Затем проводят диализ против CRB3 в течение 1 часа при +4°С. Затем проводят диализ против CRB4 в течение 1 часа при +4°C. Затем проводят диализ против CRB5 (в течение 1 часа при +4°C. Затем проводят диализ против CRB6 в течение ночи при +4°C. Таким образом, сборку нуклеосом проводят при субоптимальном соотношении ДНК-матриц и гистоновых белков (в качестве источников гистоновых белков может выступать донорный хроматин, рекомбинантные прокариотические и эукариотические гистоны, синтетические гистоны, а также гистоны, полученные от разных видов животных) с помощью модифицированного метода переноса с использованием продолжительного диализа. В ходе диализа получаются коровые (безлинкерные) нуклеосомы, которые хранят в силиконизированных пробирках (Eppendorf) при +4°C (заморозка не допускается). Сборку нуклеосом можно выполнять как в растворе, так и на различных сорбентах. Оценку качества сборки хроматина осуществляют различными способами, например электрофорезом в нативных условиях, с помощью определения чувствительности к рестриктазам, с помощью определения чувствительности к рестриктазам и последующего удлинения праймера и др. При необходимости (если нужны не коровые нуклеосомы, а нуклеосомы с линкерными хвостами ДНК, проводят сшивку с линкерной частью коровых нуклеосом с помощью Т4 ДНК-лигазы в течение 3 часов при 15°C. Проверка образцов нуклеосом как коровых, так с линкерами проводят с помощью нативного электрофореза.
5. Приготовление элонгационного комплекса ЭК-39. Проводят лигирование транскрипционного комплекса с нуклеосомами. Для этого готовят следующую смесь:
- 20,2 мкл транскрипционного комплекса, приготовленного, как описано в подразделе 2.4 настоящей методики.
- 0,8 мкл раствора бычьего сывороточного альбумина (БСА). Для приготовления раствора БСА используют бычий сывороточный альбумин (St. Louis, МО), который готовят в концентрации 10 мг/мл разбавлением в буфере ТВ40 (20 мМ Tris-HCl (рН 7.9), 5 мМ MgC12, 1 мМ β-меркаптоэтанола и 40 мМ КС1).
- 2 мкл раствора АТФ. Используют коммерческий препарат АТФ (GE Helthcare Life Science) с исходной концентрацией 100 мМ.
- 1 мкл раствора ПЭГ. Для приготовления раствора ПЭГ используют реактив ПЭГ8000 (Sigma). Взвешивают 40 грамм ПЭГ8000, доводят деионизованной водой до 100 мл, растворяют путем перемешивания на магнитной мешалке.
- 13 мкл раствора нуклеосом, приготовленных, как описано в подразделе 4 настоящей методики.
- 2 мкл 10-кратного раствора ТВ40.
- 1 мкл раствора Т4 лигазы. Используют коммерческий препарат Т4 лигазы (фирма -производитель - New England Biolabs, США). Исходная концентрация (400,000 units/ml). Добавляют 1 мкл стокового раствора на 20-50 мкл реакционной смеси. Общий объем смеси - 40 мкл. Смесь инкубируют при 13°С в течение 1 часа.
Затем в полученную смесь 40 мкл добавляют 4,5 мкл раствора АТФ и 2 мкл α-32Ф-ГТФ. Для приготовления раствора АТФ используют коммерческий препарат АТФ (GE Helthcare Life Science). Исходная концентрация (100 мМ). Подготовку проводят в соответствии с инструкцией производителя. Стоковый раствор концентрацией 100 мМ разводят деионизованной водой до концентрации 100 мкМ, т.е. в 100 раз. Для приготовления раствора α-32p-ΓΤΦ используют коммерческий препарат α-32р-ГТФ (Perkin Elmer). Исходная концентрация ГТФ, [α-32Ф]- 3000Ci/mmol 10mCi/ml). Подготовку проводят в соответствии с инструкцией производителя.
Общий объем смеси составляет 46,5 мкл. Инкубируют 15 мин при комнатной температуре. В полученную смесь 46.5 мкл добавляют 5 мкл раствора ГТФ, для приготовления которого использовали коммерческий препарат ГТФ (GE Helthcare Life Science). Исходная концентрация (100 мМ), подготовку проводили в соответствии с инструкцией производителя. Стоковый раствор концентрацией 100 мМ разводят деионизованной водой до концентрации 100 мкМ, т.е. в 100 раз Общий объем смеси составляет 51.5 мкл. Инкубируют 5 мин при комнатной температуре. Смесь доводят до объема 130 мкл добавлением 1-кратного раствора ТВ40. Проведение данных процедур позволяет получить комплексы, в которых РНК полимераза успевает транскрибировать 11-мерный олигонуклеотид. При этом РНК полимераза останавливается за 39 нуклеотидов от входа в нуклеосому. Такие комплексы получили название «ЭК-39». После подготовки смеси (объемом 51.5 мкл) аликвотят и для дальнейших экспериментов по транскрипции используют 10 мкл.
Оценку получившихся ЭК-39 проводят следующим образом. ЭК-39 сорбируют на Ni2+-NTA агарозных зернах в течение 5 мин, аккуратно перемешивая каждые 40 сек. Затем трижды промывают 0,5 мл ТВ40, инкубируют в присутствии 0,5 мг/мл ацетилированного БСА (Sigma-Aldrich) в течение 10 мин и промывают два раза 0,5 мл ТВ40. Центрифугируют образцы при 1600 оборотах в минуту в течение 1 мин при комнатной температуре. Отмывают зерна с иммобилизованными ЕС-39 комплексов один раз с ТВ40, дважды ТВ300 (20 мМ Tris-HCl (рН 7.9), 5 мМ MgC12, 1 мМ β-меркаптоэтанола и 300 мМ KCl) и дважды ТВ40. Центрифугируют после каждой отмывки на микроцентрифуге Eppendorf при 1600 оборотах в минуту в течение 1 мин при комнатной температуре. Инкубируют отмытые зерна в 30 мкл буфера ТВ 150 ((20 мМ Tris-HCl (рН 7.9), 5 мМ MgC12, 1 мМ β-меркаптоэтанола и 150 мМ КС1) с 100 мМ имидазола при комнатной температуре в течение 5 мин при осторожном пипетировании каждые 30-40 сек. Для измерения ЭК-39 используют небольшую аликвоту (3-4 мкл).
6. Транскрипция in vitro в присутствии PARP1. Используя Ni2+-NTA агарозные зерна (50% суспензии в этаноле), можно сорбировать полученные комплексы затем, после отмывки, добавлять другие «буквы» нуклеотидов - ТТФ либо УТФ, чтобы продвинуть РНК полимеразу далее во время транскрипции. Если добавить все «буквы», то транскрипция идет на всей матрице и получается полноразмерный транскрипт. Реализуется следующим образом.
6.1. Транскрипция до получения полноразмерного транскрипта
После подготовки ЭК-39 (объемом 51.5 мкл) аликвотят и для 1 эксперимента транскрипции используют 10 мкл, добавляют 2 мкл стокового раствора 4 дезоксинуклеотидтрифосфатов (дНТФ) и 2 рМ очищенного фактора PARP-1, которые разводят буфером для транскрипции до 20 мкл. Буфер для транскрипции готовят следующим образом: 1,3 мкл 2М KCL (до конечной концентрации 150 мМ KCL), 6,7 мкл буфера ТВ 150 (20 мМ Tris-HCl, рН 7.9, 5 mM MgC12, 10 тМ ZnC12, 2 тМ 2-меркаптоэтанола).
Полученную смесь инкубируют 15 мин для качественных экспериментов. В таком виде получают полноразмерные транскрипты. Остановку транскрипции и выделение транскриптов описано в разделе 6.3. Оценку полученных результатов проводят способом, описанным в разделе 7.
6.2. Остановка транскрипции и выделение транскриптов
Транскрипцию останавливают эквивалентным объемом раствора фенол/хлороформа (1:1). Для приготовления раствора фенол/хлороформа использовали фенол производства Sigma, уравновешенный буфером состава 10 mM Tris НС1, рН 8.0, 1 тМ EDTA, с хлороформом производства J.T. Baker в соотношении 1:1). Тщательно перемешивали фенол-хлороформную смесь с транскрипционной смесью. Затем центрифугируют 14800 оборотов/мин на центрифуге (Eppendorf) при комнатной температуре 15 мин. Отбирают супернатант, содержащий нуклеиновые кислоты. К супернатанту добавляют раствор преципитации, приготовленный соединением раствора гликогена концентрации 100 мкг/мкл производства Sigma из расчета 1 мкл/100 мкл и 3М ацетат натрия рН 5 из расчета 1/10 объема супернатанта. Из полученной смеси экстрагированные транскрипты высаживают с помощью спирта. Добавляют 95% этанол (стоковый раствор - J.T. Baker, до конечной концентрации ~70%, т.е. 3 объема спирта к 1 объему супернатанта. Инкубируют в течение 20 мин при -70°C. Центрифугируют 14800 оборотов/мин (Eppendorf) 15 мин. Отмывают холодным 70-75% этанолом. Центрифугируют 14800 оборотов/мин (Eppendorf) 15 мин. Отбирают максимально всю надосадочную жидкость с помощью вакуумного насоса. Затем сушат несколько минут пассивно либо на бане 42°С. Осадок используют для оценки транскрипции. Для проведения анализа полученных транскриптов и ДНК-гистоновых комплексов используют электрофорез в денатурирующем геле. Для дополнительной информации можно использовать электрофорез в нативном геле, анализ чувствительности к рестриктазам с последующим проведением электрофореза в нативном геле, ДНКаза1-футпринтинг и пр.
7. Оценка транскрипции с помощью электрофореза
Метод оценки транскрипции представлен на конкретном примере. На фиг. 1 приведен скан ПААГ электрофореза после прохождения транскрипции в присутствии или отсутствии PARP1.
На электрофореграмме (фиг. 1) наблюдается ряд полос различного размера и интенсивности, отражающих силу паузирования РНК полимеразы во время процесса транскрипции: чем сильнее паузирование, тем ярче полоска на геле. Каждая из полос характеризует транскрипционные барьеры для полимеразы - зоны паузирования. Паузирование РНК полимеразы происходит в результате прохождения фермента по ДНК, накрученной на нуклеосому. Зона нуклеосомы выделена слева овалом. Под влиянием различных факторов паузирование может ослабевать, в таком случае полоски на геле внутри нуклеосомной области становятся более бледными в нижней части и сдвигаются все выше, вплоть до положения “R-off” (от англ. - "run-off»). В положении “r-off” РНК-полимераза успешно проходит всю матрицу и заканчивает синтез полного транскрипта. Каждая пауза внутри нуклеосомы обозначается по положению нуклеотида, на котором происходит остановка, по отношению к “входу” в нуклеосому.
Приведен электрофорез после транскрипции нуклеиновых кислот в присутствии PARP-1 как в присутствии НАД+, так и без НАД+. Было обнаружено, что, если только PARP1 присутствует в реакции, транскрипционные бар