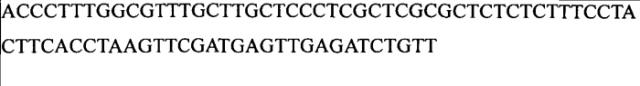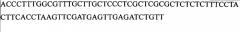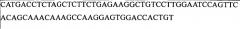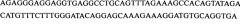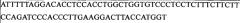Способ селективного отбора целевых участков днк
Иллюстрации
Показать всеИзобретение относится к области биохимии и может быть использовано при разработке методов молекулярно-генетической диагностики. Заявлен способ селективного отбора целевых участков ДНК. Сущность способа заключается в следующем. Геномная ДНК расщепляется случайным образом УЗ на короткие, размером 150-500 нуклеотидов, участки. Синтезируются якорные олигонуклеотиды, которые комплементарны целевым участкам генома. Якорные олигонуклеотиды ковалентно связываются с магнитными микросферами. Связанные с магнитными частицами якорные олигонуклеотиды используются для гибридизации в растворе с расщепленной геномной ДНК. Далее методом магнитной сепарации проводится удаление неспецифически связавшейся ДНК. Далее проводится анализ результатов гибридизации методом ПЦР в реальном времени с использованием праймеров с последовательностями, комплементарными геномной ДНК вне последовательностей якорных олигонуклеотидов. Изобретение позволяет с высокой скоростью и эффективностью проводить целевой отбор участков ДНК. 4 з.п. ф-лы, 3 табл.
Реферат
Изобретение относится к области молекулярной биологии, генетическим исследованиям, клиническим исследованиям и может быть использовано при разработке методов молекулярно-генетической диагностики.
Большая часть молекулярно-генетических исследований основана на выделении и последующем анализе целевых участков ДНК, т.к. исследовать геном целиком, как правило, является нецелесообразным и экономически невыгодным. В связи с этим возрастает необходимость предварительного выделения специфических участков генома, таких как: отдельные хромосомы, участки хромосом, отдельные гены, специфические семейства генов, экзоны, маркерные участки генома. К тому же для многих исследовательских и диагностических целей необходимо определение нуклеотидных последовательностей специфических участков генома одновременно в большом количестве образцов. Таким образом, создание системы для определения большого числа селективно отобранных локусов и/или протяженных регионов генома одновременно из большого множества различных образцов значительно ускорило и упростило бы анализ в клинических исследованиях. В настоящее время для локус-специфичного выделения участков генома используют методы, основанные на амплификации и гибридизации искомых фрагментов ДНК.
Селективный отбор с помощью ПЦР.
Полимеразная цепная реакция традиционно используется для выделения необходимого участка генома на протяжении нескольких десятков лет. Однако применение метода стандартной ПЦР для амплификации большого числа молекул ограниченно в виду трудоемкости и требования значительных материальных затрат. Для выделения разных целевых фрагментов в одной реакции проводят так называемую мультиплексную ПЦР, путем комбинирования более чем одной пары праймеров [1].
F.Dahl с соавторами разработали процедуру селективного отбора нескольких генов человека путем комбинирования мультиплексной ПЦР с мишень специфичной амплификацией. Хотя предложенный метод позволяет выделять и обогащать одновременно до 177 фрагментов, с его помощью все еще невозможно осуществить выделение протяженных участков ДНК, например всех экзонов человеческого генома [2].
Существует методика, разработанная R.Tewhey с коллегами, выделения и обогащения ДНК, основанная на амплификации целевых фрагментов генома в микрокаплях эмульсии [3]. Метод основан на проведении эмульсионной ПЦР, каждая капля которой содержит только одну пару праймеров, матрицу и все необходимые компоненты для протекания реакции. Представленная методика позволяет проводить около 1,5 млн отдельных ПЦР реакций одновременно в одной пробирке с незначительными количествами геномной ДНК. Тем не менее, несмотря на высокие показатели специфичности и точности определения нуклеотидных последовательностей целевых участков генома, амплифицированных в микрокаплях, использование данного метода ограничивается из-за необходимости синтеза огромного числа праймеров, требующих существенного финансирования.
Гибридизация целевого фрагмента генома с клонированным комплементарным участком
Для быстрого и эффективного генотипирования и идентификации всех нуклеотидных вариантов в участке генома длиной более 100,000 п.н. применяют метод гибридизации геномной ДНК с бактериальной искусственной хромосомой. S.Bashiardes [4]. С коллегами провели идентификацию SNPs в транскрибируемом участке гена длиной 150 тыс. п.н. Уникальный однокопийный участок гена длиной 300 п.н. был клонирован в биотинилированную бактериальную искусственную хромосому. Отбор дуплексных молекул проводили на основе иммуномагнитной сепарации путем внесения в реакционную смесь магнитных частиц, покрытых стрептавидином, при помощи внешнего магнита, после чего проводили элюцию и амплификацию фрагментов искомой ДНК. Результатом множественного проведения ПЦР реакций являются дополнительные ошибки, вносимые ферментом ДНК-полимеразой в ходе синтеза нуклеотидной цепи, что отрицательно сказывается на результате генотипирования. Также данный метод не эффективен в исследовании коротких участков ДНК, например экзонов. В силу многих недостатков и трудоемких манипуляций в работе с бактериальной искусственной хромосомой разработанный метод не получил широкого практического применения.
Гибридизация целевого фрагмента генома с олигонуклеотидами, иммобилизованными на твердофазном носителе.
T.Albert с коллегами предложили альтернативный мультиплексный ПЦР способ прямого выделения специфических локусов генома как существенной длины, так и большого числа относительно коротких участков, расположенных на некотором удалении друг от друга. На микрочипе были прикреплены синтезированные олигонуклеотидные пробы для связывания 6726 участков ДНК и проводили гибридизацию с участком нуклеиновой кислоты общей протяженностью от 200 тыс до 5 млн п.н. [5].
Существует метод компании Affymetrix, Inc. (США), номер патента CA 2356396, Target enrichment and amplification for array analysis - метод селективного отбора при помощи твердофазного носителя - микроматрицы, на которую нанесены олигонуклеотиды для проведения самого отбора. Однако данный метод подходит для связывания достаточно длинных фрагментов геномной ДНК (около 500 п.о.) и ограничивает исследования локусов меньшей длины, что не позволяет применять его в изучении коротких белок-кодирующих последовательностей.
Гибридизация целевого фрагмента генома с олигонуклеотидами в растворе.
В ряде последних работ несколько групп ученых совместили преимущества метода гибридизации на твердофазном носителе с проведением реакции в растворе, что позволило выделить необходимые участки генома с высокой кинетикой проведения реакции. Группа ученых медицинской школы Гарварда под руководством Shendure [6] предложили способ выделения экзонов с использованием инвертированных проб для массового параллельного секвенирования. Авторы разработали олигонуклеотидные пробы, включающие уникальные последовательности, которые в свою очередь были комплементарны участкам, фланкирующим интересующие фрагменты генома. Пробы были подобраны к 10 тыс экзонов длиной от 60 до 200 п.н. и синтезировали параллельно на микрочипе. По результатам двух параллельных экспериментов было показано, что только 20% целевых участков генома были выделены и отсеквенированы. Авторы полагают, что разработанный метод может использоваться для выделения, амплификации и секвенирования большого количества экзонов генома человека. Однако представленная методика требует дальнейшей оптимизации.
Krishnakumar с коллегами [7] усовершенствовали способ выделения целевых участков генома путем использования инвертированных проб, которые были в три раза длиннее таковых в работе Shendure. Инвертируемые последовательности проб были комплементарны 485 участкам, фланкирующим целевые регионы генома длиной 500 п.н. Всего было синтезировано 557 проб. В результате сравнения и анализа данных, полученных в нескольких повторностях на разных геномных секвенаторах, было показано, что разработанные пробы способны выделить до 90% специфических участков генома, для 70% было показано десятикратное перекрывание последовательности чтениями.
Хотя рассмотренные выше методы не показывают высокой производительности выделения целевых участков, они обладают существенным преимуществом в том, что олигонуклеотидные зонды синтезируются на микрочипах и амплифицируются с помощью одной пары праймеров, и, следовательно, не требуют больших материальных и трудозатрат по сравнению с синтезом большого числа праймеров.
Существует способ, патент на который принадлежит компании F. Hoffmann-La Roche AG (Швейцария), CA 2699835, Methods and systems for solution based sequence enrichment and analysis of genomic regions, 2008 год и компаниям Hoffmann La Roche (Швейцария), Roche Diagnostics GmbH (Германия), EP 20070020660, Enrichment and sequence analysis of genomic regions, 2007, при котором отбор проводится методом иммуномагнитной сепарации, с помощью биотинилированных олигонуклеотидов в растворе с последующим использованием твердофазного носителя (магнитных микрочастиц со стрептавидином) для выделения из жидкой фазы целевого комплекса олигонуклеотид-нуклеиновая кислота. Недостатком данного метода является введение дополнительной метки при синтезе олигонуклеотидов и дополнительных методических шагов при очистке, что удлиняет всю процедуру селективного отбора ДНК.
В общем смысле, магнитные частицы представляют собой ядра из оксидов двух- и трехвалентного железа, покрытые диэлектрической оболочкой и обладающие суперпарамагнитными свойствами. Как правило, для работы с нуклеиновыми кислотами используют магнитные частицы диаметром от 0,5 до 3 мкм, тогда как более крупные (до 10 мкм) применяют в работах с органеллами и целыми клетками. Применение магнитных частиц обладает рядом преимуществ по сравнению с использованием других твердофазных носителей, важными из которых являются [8, 9]:
1. Частицы обладают высокой магнетической способностью, обеспечивающей легкое и быстрое удаление их из раствора путем перемещения пробирок в магнитное поле (магнитный штатив) без дополнительных этапов центрифугирования, фильтрации, разделение с помощью мембран, что значительно уменьшает потери, повреждения биологического материала и ускоряет проведение эксперимента. Магнетические свойства пропадают сразу же после переноса частиц из магнитного штатива в обычный и, таким образом, частицы не слипаются и легко ресуспендируются. Магнитные частицы обладают незначительным межчастичным взаимодействием и имеют низкую скорость седиментации в растворе благодаря плотным диэлектрическим оболочкам, покрывающим металлическое ядро частиц.
2. Частицы химически инертны, не ингибируют ферментативные реакции, их можно использовать в экспериментах in vitro и in vivo.
3. Частицы имеют большую площадь поверхности, что позволяет иммобилизовать высокую плотность активных групп.
4. Частицы стабильны, долго хранятся без потери функциональной активности в водных растворах.
5. С использованием магнитных частиц возможно автоматизировать процессы выделения макромолекул, органелл и целых клеток.
На сегодняшний день компания Agilent Technologies (США) разработала технологию «SureSelect», для которой синтезирует рибонуклеотидные зонды, комплементарные целевым фрагментам генома, для проведения массового параллельного секвенирования под индивидуальный заказ клиентов. Преимуществом технологии «SureSelect», основанной на однофазной гибридизации, является возможность одновременного выделения специфических участков генома из многочисленных образцов, простота использования и затрата незначительного времени. Для эффективного проведения эксперимента и получения достоверного результата с использованием техники «SureSelect» не требуется большого количества образца, что особенно важно в клинических исследованиях пациентов.
В Российской Федерации в настоящий момент нет прямых или косвенных аналогов данных методик целевого отбора участков ДНК. На территории РФ не зарегистрировано ни одного патента или заявки на патент, которая бы описывала возможные способы или методы для проведения селективного отбора ДНК. Технической задачей настоящего изобретения является упрощение способа, повышение чувствительности и точности анализа описанных выше зарубежных аналогов.
Сущность способа заключается в следующем. Синтезируются якорные олигонуклеотиды, которые комплементарны целевым районам генома. Данные якорные олигонуклеотиды ковалентно связываются с магнитными микросферами, в дальнейшем, в виде такого комплекса используются для гибридизации с геномной ДНК, расщепленной случайным образом УЗ на короткие, размером 150-500 нуклеотидов, участки. В ходе гибридизации отдельные участки геномной ДНК за счет комплементарного взаимодействия ДНК-ДНК присоединяются к олигонуклеотиду на магнитной микросфере. Далее методом магнитной сепарации проводится удаление неспецифически связавшейся ДНК и на магнитных микросферах остаются только целевые участки геномной ДНК, которые необходимо генотипировать - происходит обогащение ДНК целевыми участками.
Метод гибридизации искомых участков нуклеиновой кислоты с иммобилизованными олигонуклеотидными зондами на твердых носителях широко применяется в современной молекулярной биологии для выделения, идентификации и генетического анализа специфических нуклеотидных последовательностей. Однако в методах гибридизационного анализа, проводимых на твердых носителях, кинетика протекания реакции гибридизации в десятки раз уменьшена по сравнению с таковой в гомогенной системе. Сочетание применения олигонуклеотидных зондов, фиксированных на твердофазных носителях с проведением реакции в водной реакционной смеси, значительно повышает эффективность и скорость протекания реакций гибридизации. Таким образом определяющими отличительными признаками предлагаемого способа, по сравнению с зарубежными аналогами, являются: высокая скорость и эффективность целевого отбора участков ДНК.
Технология селективного отбора целевых участков ДНК иллюстрируется следующим примером конкретного выполнения способа.
Якорные олигонуклеотиды, представленные в таблице 1, и комплиментарные участкам генома человека ковалентно связывали с магнитными частицами в течение 120 минут при 45°C. Состав смеси: 10 мг частиц в DMSO и 10 нмоль олигонуклеотида. Далее происходила отмывка магнитных частиц от неспецифически связавшихся олигонуклеотидов по следующей схеме:
1. 1×SSC/0.2% SDS, 10 мин, 50°C,
2. удаление супернатанта на магнитном штативе,
3. 1×SSC/0.2% SDS, 5 мин, 40°C,
4. удаление супернатанта на магнитном штативе,
5. повторение шагов 3 и 4,
6. 0.1×SSC,5MHH., RT,
7. удаление супернатанта на магнитном штативе,
8. повторение шагов 6 и 7,
9. ресуспендировать магнитные частицы в воде.
| Таблица 1. Последовательности якорных олигонуклеотидов, для связывания с магнитными частицами. | ||
| № | Название олигануклеотида/гена | Последовательность 5'NH2→3' |
| 1 | COL2A1/COL2A1 | |
| 2 | AMT/aminomethyltransferase |
| 3 | TOP1/Top1 | |
| 4 | myosin/myosin-If | |
| 5 | GFD/glyceraldehyde-3 -phosphate dehydrogenase | |
| 6 | TOP3A/TOP3A | |
| 7 | AURKA/AURKA | |
| 8 | ZBTB38/ZBTB38 | |
| 9 | desaturase/fatty acid desaturase 1 | |
| 10 | TyrM/tyrosine 3-monooxygenase |
Далее полученные магнитные частицы с якорными олигонуклеотидами были использованы для проведения гибридизации с геномной ДНК. Гибридизацию проводили следующим образом.
Гибридизационный буфер: 10×SSPE, 10х раствор Денхарда, 10 мМ ЭДТА, 0.2% SDS.
1. Смешивали 2.5 мкг Cot-1 ДНК (ДНК из спермы лосося) и 500 нг геномной ДНК, прогреть 5 минут на 95°C.
2. Смешивали магнитные частицы с олигонуклеотидами, все вместе по 1 мкл.
3. Смешивали 25 мкл гибридизационного буфера, прогретого до 65°C, 10 мкл магнитных частиц с олигонуклеотидами и смесь 2.5 мкг Cot-1 ДНК и 500 нг геномной ДНК и 11.5 мкл воды.
4. Инкубировали 48 часов при 65°C.
После проведения гибридизации проводили отмывку магнитных частиц от неспецифически связавшейся ДНК в 1×SSC в течение 15 минут при 25°C и далее в 0.1×SSC в течение 15 минут при 65°C 2 раза.
Результат гибридизации анализировали методом ПЦР в реальном времени с использованием. Для этого были использованы праймеры, последовательности которых комплиментарны геномной ДНК вне последовательности якорного олигонуклеотида, связанного с магнитной частицей (табл. 2).
| Таблица 2 | |||
| Последовательность праймеров для проверки эффективности обогащения. | |||
| № | Название олигонуклеотида | Последовательность 5'→3' | Длина праймера, н.п. |
| 1 | COL2A1_F | 5'-ACCAGCCACCAAGCCTTGGAC-3' | 21 |
| COL2A1_Rev | 5'-CAGCTCGCCTCTTCGAAGCAC-3' | 21 | |
| 2 | AMT_F | 5'-TGGGGCCATTGACCTATCCATG-3' | 22 |
| AMT_Rev | 5'-GAGCCAGGGGAGCACACTGG-3' | 20 | |
| 3 | TOP1_F | 5'-GGGCGCAGAGAATTGTCAGGG-3' | 21 |
| TOP1_Rev | 5'-AAGGTCAAAAGGACTTCTTACATCC-3' | 25 | |
| 4 | Myosin_F | 5'-CCCCACAGAAGTCCACCATGC-3' | 21 |
| Myosin_Rev | 5'-CAGGAGCCCAGACACCAGC-3' | 19 | |
| 5 | GFD_F | 5'-CCCTTTTGTAGGAGGGACTTAGAG-3' | 24 |
| GFD_Rev | 5'-GCTCAGCTGCACCCTTTAGG-3' | 20 | |
| 6 | TOP3A_F | 5'-TATGTCTCCCGTGGCACCTCC-3' | 21 |
| TOP3A_Rev | 5'-TACAGCAACACGGTAAGGTTTGG-3' | 23 | |
| AURKA_F | 5'-AAGAGTCCACTAACACTGAGGTC-3' | 23 | |
| AURKA_Rev | 5'-GGGTTAAGCCTGATGGCTAGC-3' | 21 | |
| 8 | ZBTB38_F | 5'-CTCCTCTTGTTTCAGATGACAGTC-3' | 24 |
| ZBTB38_Rev | 5'-CTTCCACAATGATAGTGACATCGC-3' | 24 | |
| 9 | Desaturase_F | 5'-TGGCATTGTCCCTCAAGCTCC-3' | 21 |
| Desaturase_Rev | 5'-GCAGCTCACTAAAGGAGTCAGG-3' | 22 | |
| 10 | TyrM_F | 5'-GTGGGATTTCCCTGGGAAGG-3' | 20 |
| TyrM_Rev | 5'-CTAATCCACACAGTGACACCACAG-3' | 24 | |
| 11 | MTOR Fw | 5'-CTGCTGAGTTGGGCAGTGTC-3' | 20 |
| MTOR Rev | 5'-AGGTGGGAGGATCACCTGAG-3' | 20 | |
| 12 | APOA1 Fw | 5'-GTGGGCTCAGCTTCTCTTGC-3' | 20 |
| APOA1 Rev | 5'-AGCCCTACCTGGACGACTTC-3' | 20 | |
| 13 | CA2 Fw | 5'-GCGGGAAAGGGTTGTAACGG-3' | 20 |
| CA2 Rev | 5'-AGCCACAGGTGGTGCTGAAG-3' | 20 | |
| 14 | ANXA5 Fw | 5'-TCCGAGACAGAAGGGCAGAA-3' | 20 |
| ANXA5 Rev | 5'-TTTGGCTGGCCCAAAGGACA-3' | 20 |
Пары праймеров с 11 по 14 были использованы в качестве «отрицательных контролей». Они были синтезированы на участки геномной ДНК, которые не должны обогащаться, т.к. на них не были синтезированы олигонуклеотиды. После проведения ПЦР в реальном времени получили следующие значения разницы в циклах выхода на линейный участок аплификации, таблица 3.
| Таблица 3 | ||||||||
| Разница в циклах выхода на линейный участок амплификации между специфическими праймерами (№1-10) и неспецифическим (№11-14). Данные приведены для магнитных частиц Dynabeads® MyOne™ Carboxylic Acid (Invitrogen). гДНК-праймеры на геномную ДНК результат до гибридизации, МЧ-праймеры на геномную ДНК, результат после гибридизации с магнитными частицами. | ||||||||
| Название гена | Разница с праймером №11 | Разница с праймером №12 | Разница с праймером №13 | Разница с праймером №14 | ||||
| гДНК | МЧ | гДНК | МЧ | гДНК | МЧ | гДНК | МЧ | |
| COL2A1 | 0,5 | 3.6 | 0.1 | 3.8 | 0.1 | 3.9 | -0.2 | 4.4 |
| АМТ/aminomethyl-transferase | -0,2 | 4,8 | 0,3 | 3,2 | 0,6 | 3,5 | 0,4 | 3,9 |
| TOP1 | 0,6 | 4,2 | 0,6 | 4,3 | 0,2 | 5.0 | -0,5 | 4,1 |
| myosin/myosin-If | 0,3 | 4,7 | -0,2 | 4,2 | 0,1 | 4,7 | 0,3 | 3,9 |
| GFD/glyceraldehy de-3-phosphate | -0,2 | 4,3 | 0,1 | 3,8 | 0,6 | 4,4 | -0,2 | 4,7 |
| dehydrogenase | ||||||||
| TOP3A | 0,1 | 4,5 | 0,2 | 3,9 | 0,3 | 3,2 | 0,5 | 3,9 |
| AURKA | -0,2 | 3,7 | 0,4 | 2,9 | 0,1 | 3,4 | -0,3 | 3,6 |
| ZBTB38 | 0,1 | 3,5 | 0,2 | 3,7 | 0,2 | 3,9 | 0,2 | 4,4 |
| desaturase/fatty acid desaturase 1 | 0,5 | 3,7 | 0,4 | 3,6 | 0,4 | 3,8 | -0,2 | 3,9 |
| TyrM/tyrosine 3-monooxygenase | 0,2 | 4,3 | 0,6 | 4,2 | 0,5 | 4,1 | 0,3 | 3,8 |
В связи с тем что эффективность ПЦР не одинаковая для разных участков генома и разных пар праймеров, данные, полученные после амплификации ДНК, обогащенной с помощью магнитных частиц (столбцы МЧ, табл.3), нормировались на данные после амплификации геномной ДНК целевых и контрольных участков - пар праймеров 11-14. Таким образцом происходит существенное обогащение, примерно в 10 раз, специфическими последовательностями относительно неспецифических, т.е. осуществляется отбор целевых последовательностей ДНК.
Данный пример иллюстрирует, что разработанный способ может быть использован в качестве технологии селективного отбора целевых участков ДНК.
Исследование выполнено в рамках федеральной целевой программы «Исследования и разработки по приоритетным направлениям развития научно-технологического комплекса России на 2007-2012 годы», государственный контракт №16.512.11.2031. от 14 февраля 2011 г.
Источники информации
1. Shigemori Y, Mikawa T, Shibata Т, Oishi M. Multiplex PCR: use of heat-stable Thermus thermophilus RecA protein to minimize non-specific PCR products. Nucleic Acids Res. 2005. 33 (14): 126
2. Dahl F, Stenberg J, Fredriksson S, Welch K, Zhang M, Nilsson M, Bicknell D, Bodmer WF, Davis RW, Ji H. Multigene amplification and massively parallel sequencing for cancer mutation discovery. Proc Natl Acad Sci USA. 2007. 104 (22): 9387-92.
3. Tewhey R, Warner JB, Nakano M, Libby B, Medkova M, David PH, Kotsopoulos SK, Samuels ML, Hutchison JB, Larson JW, Topol EJ, Weiner MP, Harismendy O, Olson J, Link DR, Frazer KA. Microdroplet-based PCR enrichment for large-scale targeted sequencing. Nat Biotechnol. 2009. 27 (11): 1025-1031.
4. Bashiardes S, Veile R, Helms C, Mardis ER, Bowcock AM, Lovett M. Direct genomic selection. Nat Methods. 2005. 2 (1): 63-9.
5. Albert, Т.J. et al. Direct selection of human genomic loci by microarray hybridization. Nature Methods. 2007. 4, 903-905.
6. Porreca GJ, Zhang К, Li JB, Xie B, Austin D, Vassallo SL, LeProust EM, Peck BJ, Emig CJ, Dahl F, Gao Y, Church GM, Shendure J. Multiplex amplification of large sets of human exons. Nat Methods. 2007. 4 (11): 931-6.
7. Krishnakumar, S. et al. A comprehensive assay for targeted multiplex amplification of human DNA sequences. Proc. Natl Acad. Sci. USA. 2008. 105, 9296-9301.
8. Lund V, R. Schmid, D. Rickwood and Er. Homes. Assessment of methods for covalent binding of nucleic acids to magnetic beads, Dynabeadsm, and the characteristics of the bound nucleic acids in hybridization reactions. Nucleic acids research. 1988. V.16.
9. Uhlen M. Magnetic separation of DNA. 1989. Nature 340, 733-734.
1. Способ селективного отбора целевых участков ДНК, включающий:а) расщепление геномной ДНК;б) ковалентное связывание якорных олигонуклеотидов, комплементарных целевым участкам генома, с магнитными частицами;в) гибридизацию участков расщепленной геномной ДНК с якорными олигонуклеотидами, связанными с магнитными частицами, в растворе;г) отмывку магнитных частиц от неспецифически связавшейся ДНК;д) анализ результатов гибридизации методом ПЦР в реальном времени с использованием праймеров с последовательностями, комплементарными геномной ДНК вне последовательностей якорных олигонуклеотидов.
2. Способ по п.1, отличающийся тем, что геномная ДНК подлежит предварительной фрагментации.
3. Способ по п.1, отличающийся тем, что якорные олигонуклеотиды являются искусственно синтезированными олигонуклеотидами и комплементарны целевым участкам генома.
4. Способ по п.1, отличающийся тем, что якорные олигонуклеотиды ковалентно соединены с парамагнитными микросферами.
5. Способ по п.1, отличающийся тем, что отбор целевых участков ДНК проводится посредством магнитной сепарации.