Рекомбинантный штамм бактерий escherichia coli n16 (pm.alubi) - продуцент днк-метилтрансферазы m.alubi
Иллюстрации
Показать всеИзобретение относится к биотехнологии и представляет собой рекомбинантный штамм бактерий Escherichia coli N16 (pM.AluBI), являющийся продуцентом ДНК-метилтрансферазы AluBI, переносящей метальную группу с S-аденозил-L-метионина (SAM) на 6-й атом азота аденинового основания сайта узнавания, с образованием метилированной последовательности 5′-(m6A)GCT-3′/3′-TCG(m6A)-5′ на обеих цепях ДНК. Штамм получен путем трансформации клеток Escherichia coli RR1 плазмидой pM.AluBI-16. Данная плазмида сконструирована на основе вектора pUC19 и содержит ген, кодирующий ДНК-метилтрансферазу AluBI из природного штамма Arthrobacter luteus В. Изобретение позволяет получить рекомбинантный штамм бактерий Escherichia coli N16 (рМ.AluBI), продуцирующий с высоким выходом ДНК-метилтрансферазу с новой специфичностью. 4 ил., 1 табл., 3 пр.
Реферат
Изобретение относится к области молекулярной биологии, биотехнологии и микробиологической промышленности, и касается нового рекомбинантного штамма бактерий Escherichia coli, который может быть использован для получения препарата ДНК-метилтрансферазы M.AluBI.
Главными биотехнологически значимыми параметрами ДНК-метилтрансфераз являются узнаваемая последовательность ДНК, узнаваемые нуклеотиды этой последовательности, метилируемые атомы оснований этих нуклеотидов, а также активность препарата фермента, выделенного из соответствующего штамма-продуцента.
ДНК-метилтрансферазы переносят метальную группу с S-аденозил-L-метионина (SAM) на основания ДНК и классифицируются на цитозин (С5)-ДНК-метилтрансферазы, цитозин (N4)-ДНК-метилтрансферазы и аденин (N6)-ДНК-метилтрансферазы. Метилированные основания обозначают соответственно m5C, m4С и m6A (1). Наиболее разнообразна специфичность ДНК-метилтрансфераз у бактерий, в которых открыто около 350 прототипов этих ферментов (2).
Известен рекомбинантный штамм бактерии Escherichia coli, который продуцирует ДНК-метилтрансферазу M.Sse9I, образующую метилированную последовательность 5′-А(m6А)ТТ-3′ (3). M.Sse9I, также как и M.AluBI, метилирует аденин в последовательности из 4 оснований.
Недостатком известного штамма является то, что ДНК-метилтрансфераза M.Sse9I не узнает последовательность нуклеотидов 5′-AGCT-3′ и не образует метилированную последовательность 5′-(m6A)GCT-3′.
Известен штамм бактерии Arthrobacter luteus, который продуцирует ДНК-метилтрансферазу M.AluI, образующую метилированную последовательность 5′-AG(m5C)T-3′ (4). Недостатком этого штамма является то, что ДНК-метилтрансфераза M.AluI не образует метилированную последовательность 5′-(m6A)GCT-3′ и является малопродуктивным в отношении ДНК-метилтрансферазы.
В настоящее время не описаны штаммы бактерий, являющиеся продуцентами ДНК-метилтрансфераз, узнающих последовательность 5′-AGCT-3′ и метилирующих остаток аденина этой последовательности с образованием 5′-(m6A)GCT-3′.
Наиболее близким к заявляемому штамму - прототипом, является рекомбинантный штамм бактерии Escherichia coli, который продуцирует эндонуклеазу рестрикции AluI и ДНК-метилтрансферазу M.AluI образующую последовательность 5′-AG(m5C)T-3′ (5). Штамм получают путем трансформации клеток Escherichia coli экспрессирующим вектором, содержащим ДНК, кодирующей ген рестриктазы AluI и вектором для экспрессии метилазы, способной защищать хозяйскую ДНК от расщепления интересуемой рестриктазой. Метилазный вектор совместим с экспрессирующим вектором, несущим ген рестриктазы. Полученный рекомбинантный штамм способен осуществлять высокоэффективную экспрессию генов эндонуклеазы рестрикции AluI и ДНК-метилтрансферазы M.AluI
Недостатком известного штамма является то, что ДНК-метилтрансфераза M.AluI метилирует цитозин, но не аденин в последовательности узнавания 5′-AGCT-3′, поэтому, в отличие от M.AluBI, не образует последовательность 5′-(m6A)GCT-3′.
Задачей изобретения является получение высокопродуктивного штамма - продуцента ДНК-метилтрансферазы M.AluBI.
Поставленная задача решается путем получения штамма бактерий Escherichia coli N16 (рМ.AluBI) - продуцента ДНК-метилтрансферазы, узнающей последовательность нуклеотидов 5′-AGCT-3′ и метилирующей в присутствии SAM остаток аденина этой последовательности с образованием 5′-(m6A)GCT-3′.
Техническим результатом изобретения является получение рекомбинантного штамма бактерий Escherichia coli N16 (рМ.AluBI), продуцирующего ДНК-метилтрансферазу заданной специфичности с более высоким выходом, по сравнению с исходным природным штаммом.
Предлагаемый штамм-продуцент получен трансформацией плазмидной ДНК pM.AluBI-16 клеток штамма Escherichia coli RR1. Плазмида pM.AluBI-16 получена в результате клонирования в E.coli участка хромосомной ДНК штамма Arthrobacter luteus В. Этот штамм, выделенный из природного материала (почвы) в результате целенаправленного систематического поиска, является продуцентом ферментов системы рестрикции-модификации AluBI (6). Однако продуктивность штамма в отношении ДНК-метилтрансферазы не высока, активность M.AluBI в клетках природного штамма составляет менее 3000 е.а./г.
Плазмида pM.AluBI-16 является рекомбинантной, содержит ген, кодирующий ДНК-метилтрансферазу, метилирующую аденин в положении N6 в последовательности 5′-AGCT-3′.
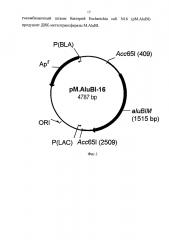
На фиг. 1 представлена физическая карта плазмиды pM.AluBI-16, где:
Асс65I - точки сшивки ДНК pUC19 (тонкая линия) и фрагмента ДНК A. luteus В (толстая линия), гидролизованных эндонуклеазой рестрикции Асс65I;
ORI - точка начала репликации;
Apr - ген bla, обеспечивающий устойчивость к ампициллину;
P(BLA) - промотор гена bla;
P(LAC) - промотор гена lacZ;
aluBIM - ген, кодирующий ДНК-метилтрансферазу AluBI.
Сконструированной плазмидой pM.AluBI-16 трансформируют клетки E. coli штамма RR1. Трансформантов отбирают на селективной агаризованной питательной среде, содержащей 100 мкг/мл ампициллина, по резистентности к антибиотику, т.е. способности к росту в его присутствии. Содержание рекомбинантной ДНК-метилтрасферазы M.AluBI составляет 70000 е.а./г влажной биомассы.
Преимуществом заявляемого штамма, по сравнению с наиболее близким аналогом, является то, что продуцируемый им фермент - M.AluBI, по своей специфичности является абсолютно новым. Этот фермент, модифицирующий ДНК по сайту 5′-AGCT-3′ с образованием 5′-(m6A)GCT-3′, предоставляет новые возможности для молекулярно-генетических работ.
Штамм Escherichia coli N16 (pM.AluBI) имеет следующие характеристики.
Культурально-морфологические признаки.
Клетки мелкие, палочковидной формы, грамотрицательные, 1×3,5 мкм, подвижные. Штамм хорошо растет на обычных питательных средах (МПА, МПБ, LB-бульон, LB-агар, минимальная среда с глюкозой). При росте на агаризованной среде LB колонии круглые, гладкие, полупрозрачные, блестящие, серые. Край ровный, диаметр колоний 1-3 мм, консистенция пастообразная. Рост в жидких средах (LB, минимальная среда с глюкозой) характеризуется ровным помутнением, осадок легко седиментирует.
Физиолого-биохимические признаки.
Клетки растут при 4-42°С, оптимум pH 6,8-7,6. В качестве источника азота используют как минеральные соли аммония, так и органические соединения: аминокислоты, пептон, триптон, дрожжевой экстракт. В качестве источника углерода при росте на минимальной среде используют глицерин, углеводы, аминокислоты.
Генетические признаки, устойчивость к антибиотикам.
Штамм-хозяин Esherichia coli RR1 (F- Δ(gpt-proA)62 leuB6 supE44 ara-14 galK2 lacY1 Δ(mcrC-mrr) rpsL20(Strr) xyl-5 mtl-1 recA+). Проявляет устойчивость к стрептомицину (25 мкг/мл).
Штамм Escherichia coli N16 (pM.AluBI) дополнительно проявляет устойчивость к ампициллину (до 300 мкг/мл), обусловленную наличием гена устойчивости в ДНК рекомбинантной плазмиды pM.AluBI-16.
Условия хранения штамма.
Штамм бактерий Escherichia coli N16 (рМ.AluBI) хранят на агаризованной среде LB со 100 мкг/мл ампициллина на чашках Петри или в стеклянных пробирках со скошенным агаром при 4°С. Пересевы на свежие среды делают один раз в месяц. Может храниться не менее года в среде LB, содержащей 30% глицерин, при -50-70°С.
Длительное хранение штамма осуществляется в лиофильно-высушенном состоянии или в растворе 30% глицерина при температуре -70°С.
Для культивирования штамма применяют среду следующего состава (г/л): пептон - 10, дрожжевой экстракт - 5, NaCl - 5, ампициллин 0,1. Культивирование проводят при 30°C с аэрацией до достижения стационарной стадии роста.
Выход препарат фермента составляет ~2 мл/г сырой биомассы с концентрацией 20000 е.а./мл.
Рекомбинантная ДНК-метилтрансфераза M.AluBI характеризуется следующими свойствами:
1. Узнает и, в присутствии SAM, метилирует последовательность 5′-AGCT-3′ с образованием на обеих цепях ДНК 5′-(m6A)GCT-3′/3′-TCG(m6A)-5′.
2. Оптимальная температура реакции 37°С.
3. Фермент проявляет высокую активность в буфере следующего состава: 100 мкМ SAM, 33 мМ Трис-ацетат (pH 7,9 при 25°С), 66 мМ калия ацетат, 1 мМ ЭДТА, 1 мМ дитиотреитол, 100 мкг/мл бычий сывороточный альбумин.
Определяющим отличием предлагаемого штамма от всех известных в настоящее время штаммов-продуцентов ДНК-метилтрансфераз является то, что он позволяет с высоким выходом получать препарат фермента M.AluBI, не имеющего аналогов, узнающего и, в присутствии SAM, метилирующего последовательность 5′-AGCT-3′ на обеих цепях ДНК, с образованием 5′-(m6A)GCT-3′/3′-TCG(m6A)-5′.
Поскольку предлагаемый штамм получен впервые и для выделения ДНК-метилтрансферазы, способной узнавать и метилировать вышеназванную последовательность нуклеотидов в указанной позиции никогда не использовался, можно сделать вывод о соответствии предлагаемого штамма критериям изобретения «новизна» и «изобретательский уровень».
Изобретение иллюстрируется примерами конкретного выполнения.
Пример 1. Конструирование рекомбинантной плазмиды pM.AluBI-16
Получение библиотеки клонов E. coli.
Штамм Arthrobacter luteus В выращивали в LM-бульоне (г/л): триптон "Organotechnie" (Франция) - 10, дрожжевой экстракт (той же фирмы) - 5, NaCl - 5, MgCl2 - 0,5, тиамин - 0,01, pH 7,0-7,4, при 30°С со встряхиванием в колбах 2 дня. Из 3 г клеток выделяли фенольным методом 1,8 мг хромосомной ДНК (7). Далее хромосомную ДНК расщепляли отдельно рестриктазами SphI и BstNSI и сшивали с помощью Т4 ДНК-лигазы по стандартной методике (8) с ДНК вектора pUC19 (9), линеаризованной SphI. Лигазной смесью трансформировали клетки E. coli RR1 методом электропорации на «Easyject Prima» (Великобритания) и выращивали в 100 мл бульона LM с 0,1 г/л ампициллина. С использованием набора «QIAGEN Plasmid Maxi Kit» (Германия) из клеток выделяли 0.4 мл ДНК суммарной плазмиды ΣpAluB/BS с концентрацией 0,2 мг/мл, представляющей собой библиотеку из 8000 рекомбинантов.
Выделение целевой плазмиды.
Плазмиду, кодирующую целевую ДНК-метилтрансферазу, отбирали по методике, описанной ранее (10). При этом ДНК ΣpAluB/BS расщепляли рестриктазой AluBI и повторно трансформировали клетки E. coli RR1. На LM-агаре с ампициллином получали 16 клонов (pAluB/BS №1-16). Из них выделяли плазмиды, в которых определяли наличие AluBI-метилирования и размеры вставок путем гидролиза этих плазмид рестриктазами AluBI и VspI, соответственно, с последующим электрофорезом в 1% агарозном геле. В результате отобрали плазмиду pAluB/BS №16 со встроенным фрагментом порядка 8100 п.н. С целью укорочения и оптимизации конечной конструкции, встроенный фрагмент из плазмиды pAluB/BS №16 подвергали гидролизу рестриктазой Асс651, и участок этого фрагмента, длиной 2101 п.н., включающий ген ДНК-метилтрансферазы M.AluBI, субклонировали в вектор pUC19/Acc65I по стандартной методике (8). В итоге отобрали плазмиду pM.AluBI-16.
На фиг. 2 представлены результаты электрофореза после обработки плазмиды pM.AluBI-16 рестриктазами AluBI и VspI, где дорожки:
1 - исходная ДНК pM.AluBI-16;
2 - pM.AluBI-16 + AluBI;
3 - pM.AluBI-16 + VspI;
M - маркер молекулярного веса ДНК 1 kb (производства ООО «СибЭнзайм»).
Данная плазмида содержит встроенный фрагмент ДНК длиной около 2100 п.н., что видно по гидролизу VspI (дорожка 3), и защищена от гидролиза AluBI (дорожка 2), то есть содержит функционально активный ген ДНК-метилтрансферазы M.AluBI. Этой плазмидой повторно трансформировали клетки Escherichia coli RR1 и получили штамм продуцент Escherichia coli N16 (рМ.AluBI).
Измерение активности фермента M.AluBI в клетках Escherichia coli N16 (рМ.AluBI).
Штамм выращивали в 300 мл LM-бульона с добавлением 100 мкг/мл ампициллина в 0,5 л колбах со встряхиванием при 30°С в течение двух суток. Культура при плотности 2,3 о.е. при 540 нм/см давала 3 г/л биомассы. 1 мл центрифугировали в пробирке «Eppendorf» при 12000 об/мин в течение 3 мин. Осадок из 1 мл суспендировали в 0,2 мл лизирующего буфера: 50 мкг/мл лизоцим, 0,4% Тритон Х-100, 10 мМ Трис-HCl (pH 8,1) и 1 мМ ЭДТА, и лизировали клетки при 22°С в течение 30 мин. 25 мкл лизата вносили в 100 мкл SM-смеси, содержащей: 50 мкг/мл ДНК фага λ (dam-, dcm-), 100 мкМ SAM, М-буфер «Y′» (33 мМ Трис-ацетат (pH 7,9), 1 мМ ЭДТА, 66 мМ ацетат калия и 1 мМ дитиотреитол), и 100 мкг/мл бычий сывороточный альбумин. Делали 10 дополнительных 2-кратных разбавлений в аликвотах по 50 мкл SM-смеси. Метилировали ДНК λ при 37°С в течение 2 ч. Прогревали смесь при 65°С в течение 10 мин для инактивации ДНК-метилтрансферазы и охлаждали до комнатной температуры. Добавляли по 5 мкл 1000 е.а./мл эндонуклеазы рестрикции AluBI в SE-буфере В100 (10 мМ Трис-HCl (pH 7,6), 50 мМ KCl, 0,1 мМ ЭДТА, 200 мкг/мл бычий сывороточный альбумин, 1 мМ дитиотреитол), дополненным 111 мМ ацетатом магния. Инкубировали реакционную смесь при 37°С в течение 1 ч и анализировали электрофорезом в 1%-ном агарозном геле. За единицу активности ДНК-метилтрансферазы принимали количество фермента, достаточное для защиты 1 мкг ДНК фага λ за 1 ч от гидролиза сопряженной эндонуклеазой рестрикции AluBI. Для простого выявления активности 5 мкл лизата, либо 5 мкл пробы хроматографии (разбавленной в 100 раз в SE-буфере В100), вносили в 50 мкл SM-смеси, метилировали ДНК 30 мин при 37°С, расщепляли сопряженной рестриктазой и проводили электрофорез в 1% агарозном геле.
На фиг. 3 представлены результаты определения активности M.AluBI в лизате клеток Escherichia coli N16 (pM.AluBI), где дорожки:
1 - метилирование 50 мкг/мл λ ДНК лизатом, разбавленным в реакционной смеси до исходной плотности клеток культуры + AluBI;
2 - метилирование лизатом, разбавленным в 2 раза + AluBI;
3 - метилирование лизатом, разбавленным в 4 раза + AluBI;
4 - метилирование лизатом, разбавленным в 8 раз + AluBI;
5 - метилирование лизатом, разбавленным в 16 раз + AluBI;
6 - метилирование лизатом, разбавленным в 32 раза + AluBI;
7 - метилирование лизатом, разбавленным в 64 раза + AluBI;
8 - метилирование лизатом, разбавленным в 128 раз + AluBI;
9 - метилирование лизатом, разбавленным в 250 раз + AluBI;
10 - метилирование лизатом, разбавленным в 500 раза + AluBI;
11 - метилирование лизатом, разбавленным в 1000 раз + AluBI;
12 - контроль без лизата + AluBI;
М - маркер молекулярного веса ДНК λ/BssT1I.
Из фиг. 3 видно, что метилирование защищает ДНК от гидролиза рестриктазой AluBI до 4-го разбавления лизата включительно, что соответствует активности M.AluBI 200 е.а./мл культуры или 70000 е.а./г клеток. Учитывая низкую активность ДНК-метилтрансфераз природных штаммов, заявляемый штамм Escherichia coli N16 (pM.AluBI) является суперпродуцентом M.AluBI.
Анализ ДНК плазмиды pM.AluBI-16.
Секвенирование встроенного фрагмента в ДНК pM.AluBI-16 было проведено методом Сэнгера (11) на автоматическом секвенаторе ABI 3130xI Genetic Analyzer («Applied Biosystems», США). Анализ первичной структуры показал, что Асс65I-фрагмент ДНК A. luteus В, клонированый в pUC19, состоит из 2101 п.н. Фрагмент содержит открытую рамку трансляции, которая является геном ДНК-метилтрансферазы M.AluBI длиной 1515 п.н.
На фиг. 4 приведена нуклеотидная последовательность гена, кодирующего ДНК-метилтрансферазу M.AluBI, а также соответствующая ей аминокислотная последовательность.
Выравнивание аминокислотной последовательности M.AluBI с помощью программы BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) выявило консервативные мотивы, характерные для аденин (N6)-ДНК-метилтрансфераз (КФ 2.1.1.72) (12).
Пример 2. Выращивание штамма и выделение рекомбинантной ДНК-метилтрансферазы M.AluBI Получение биомассы.
Клетки Escherichia coli N16 (pM.AluBI) выращивали в LM-бульоне с ампициллином в 20-лигровом ферментере «New Brunswick Scientific» (США) при 30°С, аэрации 15 л/мин, перемешивании 200 об/мин 8 ч до оптической плотности 2,2-2,5 ед/см при 540 нм. Центрифугировали на центрифуге «Beckman J2-M1» (США) в роторе JA-10 при 8000 об/мин в течение 20 минут. За два выращивания получали 124 г клеток.
Условия выделения фермента.
Фермент M.AluBI выделяли при 4°C с использованием буферных растворов:
буфер А - 10 мМ Трис-HCl pH 7,5, 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол;
буфер Б - 10 мМ К-фосфат pH 7,2, 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол.
100 г биомассы суспендировали в 500 мл буфера А, содержащего 5% глицерин, 0,5% Тритон Х-100, 1 мг/мл лизоцим, 0,1 мМ PMSF (фенилметилсульфонилфторид - ингибитор протеаз) и перемешивали 1 ч. По 100 мл суспензии разрушали ультразвуком на дезинтеграторе Soniprep 150 («MSE» Великобритания) с насадкой диаметром 2 см при амплитуде 22-24 мкм пятью импульсами по 1 мин с охлаждением во льду по 1 мин. Добавляли NaCl до 0,2 М и центрифугировали на «Beckman J2-M1» в роторе JA-20 при 18000 об/мин 30 минут.
540 мл экстракта наносили на колонку (15 см × 50 мм) фосфоцеллюлозы Р-11 «Whatman» (Великобритания) объемом 300 мл, уравновешенной буфером А с 0,2 М NaCl. Промывали 600 мл буфера А с 0,2 М NaCl. Элюировали линейным градиентом 0,2 М - 0,6 М NaCl в буфере А объемом 3 л. Собирали 200 фракций по 15 мл. Фракции 96-110, содержащие M.AluBI с максимальной активностью, объединяли.
Объединенные фракции диализовали 4 ч против 3 л буфера А с 0,05 М NaCl и наносили на колонку (10 см × 25 мм) с гепарин-сефарозой объемом 50 мл. Промывали 100 мл буфера А с 0,05 М NaCl и элюировали градиентом 0,05 М - 0,5 М NaCl в буфере А объемом 1000 мл. Собирали 100 фракций по 10 мл. Фракции 56-60, содержащие пик активности M.AluBI, объединяли.
Объединенные фракции наносили на колонку (205 см × 25 мм) со 1000 мл сефакрила S-200 «Bio-Rad» (США), уравновешенного в буфере А с 0,8 М NaCl. Элюировали 1000 мл буфера А с 0,8 М NaCl. Собирали 100 фракций по 10 мл, из которых 62-69 содержали пик целевой активности M.AluBI.
Объединенные фракции наносили на колонку (10 см × 25 мм) с 50 мл гидроксиапатита («Bio-Rad» США) в буфере Б. Промывали 100 мл буфера Б и элюировали линейным градиентом 0,01-0,2 М K-PO4 (pH 7,2) в буфере Б объемом 1000 мл. Собирали 60 фракций по 16,7 мл. Фракции 53-60, содержащие пик активности M.AluBI, объединяли и диализовали против 1 л буфера, содержащего: 50% глицерин, 10 мМ Трис-HCl (pH 7,5), 0,05 М NaCl, 0,1 мМ ЭДТА и 7 мМ β-меркаптоэтанол. 20 мл полученного препарата ДНК-метилтрансферазы M.AluBI хранили при -20°С.
Пример 3. Определение специфичности ДНК-метилтрансферазы M.AluBI
Основание, метилируемое M.AluBI, определяли путем его мечения с использованием 3H-SAM, методом IIS-кассеты (13). Олигодезоксирибонуклеотидный дуплекс содержал сайт AGCT, метилируемый M.AluBI (выделен жирным шрифтом), а также сайт GGATC, узнаваемый AclWI, эндонуклеазой рестрикции IIS-типа (выделен рамкой). При этом точки гидролиза AclWI (показаны стрелками) приходились на метилируемый сайт.
Дуплекс метилировали M.AluBI с использованием H-SAM, расщепляли AclWI, одноцепочечные фрагменты разделяли электрофорезом в 20% полиакриламидном геле, для каждого из них измеряли радиоактивность. Распределение метки во фрагментах олигонуклеотидного дуплекса, меченого M.AluBI в присутствии H-SAM, после его гидролиза AclWI представлено в таблице.
Из таблицы видно, что метка включалась преимущественно во фрагменты длиной 20 и 11 нуклеотидов. Больший фрагмент содержал только аденин метилируемого сайта (верхняя цепь), а меньший - аденин с гуанином. В тоже время, фрагменты длиной 14 и 23 нуклеотида, содержащие соответственно GCT и СТ нуклеотиды сайта AGCT, имели метку на уровне фона. Следовательно, M.AluBI метилировала только аденин, образуя 5′-(3Hm6A)GCT-3′.
Таким образом, результаты M.AluBI-3Н-метилирования сайта AGCT олигонуклеотидного дуплекса, M.AluBI-метилирования λ ДНК, блокирующего расщепление сайта AGCT эндонуклеазой рестрикции AluBI; а также выравнивание аминокислотной последовательности M.AluBI с доменами, характерными для аденин (N6)-ДНК-метилтрансфераз, доказывают, что ДНК-метилтрансфераза M.AluBI образует метилированную последовательность 5′-(m6A)GCT-3′ на ДНК и относится к аденин (N6)-ДНК-метилтрансферазам (КФ 2.1.1.72).
Предлагаемый штамм позволит получать ДНК-метилтрансферазу M.AluBI с высоким выходом и, учитывая новую специфичность фермента, использовать его в молекулярно-биологических и генно-инженерных исследованиях.
Источники информации
1. Cheng X., Blumenthal R.M. (Eds.). S-Adenosylmethionine-Dependent Methyltransferases. Structures and Functions. // Singapore, New Jersey, London, Hong Kong: World Scientific Publishing Co. Pte. Ltd. - 1999.
2. REBASE. - 2015. Available at: http://rebase.neb.com.
3. Gonchar D.A., Wolf Y.I., Degtyarev S.K. Cloning and characterization of Sse9I DNA-methyltransferase recognizing 5′-AATT-3′. // Nucleic Acids Res. - 1996. - V. 24. - P. 2790-2792.
4. Крамаров B.M., Смолянинов B.B. ДНК метилаза из Arthrobacter luteus скрывает ДНК от действия сайт-специфической эндонуклеазы AluI. // Биохимия. - 1981. - Т. 46. - С. 1526-1529.
5. Xu S., Nwankwo D.O. Method for cloning and producing the AATII and ALUI restriction endonuclease and methylase and related method for overexpressing restriction endonucleases. // Patent US 5405768 B1, оп. 11.04.1995.
6. Чернухин B.A., Болтенгаген A.A., Тарасова Г.В, Дедков B.C., Дегтярев С.Х. Новая эндонуклеаза рестрикции AluBI из Arthrobacter luteus В-изошизомер AluI, нечувствительный к присутствию 5-метилцитозина в сайте узнавания AGCT. // Вестник биотехнологии и физико-химической биологии имени Ю.А. Овчинникова. - 2007. - 3(1). - С. 21-27.
7. Smith C.L., Klco S.R. and Cantor C.R. In: Davies K. (ed.). Genome Analysis. A Practical Approach. // - 1987. - Oxford. UK: IRL Press.
8. Sambrook J., Fritsch E.F. and Maniatis T. Molecular Cloning. A laboratory manual, 2nd ed. // - 1989. - Cold Spring Harbor, New York: Cold Spring Harbor Laboratory Press.
9. Yanisch-Perron C., Vieira J., Messing J. Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mp18 and pUC19 vectors. // Gene. - 1985. - 33. - P. 103-119.
10. Mann M.В., Rao R.N., Smith H.O. Cloning of restriction and modification genes in E. coli: the HhaII system from Haemophilus haemolyticus. // Gene. - 1978. - V. 3. - P. 97-112.
11. Sanger F., Nicklen S., Coulson A.R. DNA sequencing with chain-terminating inhibitors. // Proc Natl Acad Sci USA. - 1977. - V. 74. - P. 5463-5467.
12. Timinskas A., Butkus V., Janulaitis A. Sequence motifs characteristic for DNA [cytosine-N4] and DNA [adenine-N6] methyltransferases. Classification of all DNA methyltransferases. // Gene. - 1995. - V. 157. - P. 3-11.
13. Чернухин B.A., Каширина Ю.Г., Суханова К.C., Абдурашитов М.А., Гончар Д.А., Дегтярев С.Х. ДНК-метилтрансфераза FauIA, модифицирующая второй остаток цитозина в непалиндромной последовательности 5′-CCCGC-3′ (Выделение и свойства). // Биохимия. - 2005. - Т. 70. - С. 829-837.
Рекомбинантный штамм бактерий Escherichia coli N16 (pM.AluBI), полученный трансформацией клеток штамма Escherichia coli RR1 плазмидой pM.AluBI-16, сконструированной на основе вектора pUC19 и содержащей ген, кодирующий ДНК-метилтрансферазу M.AluBI из природного штамма Arthrobacter luteus В, продуцирующий ДНК-метилтрансферазу M.AluBI, узнающую нуклеотидную последовательность ДНК 5′-AGCT-3′ и метилирующую остаток аденина этой последовательности с образованием 5′-(m6A)GCT-3′.